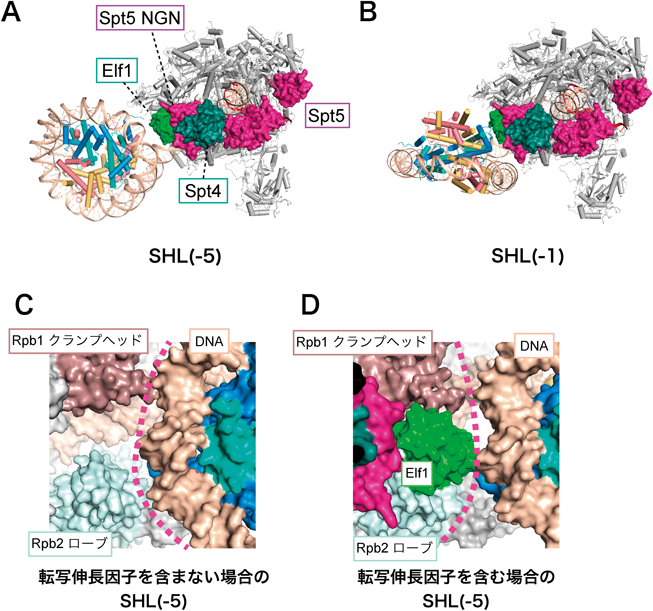
RNAポリメラーゼII-ヌクレオソーム複合体構造から得られたクロマチン転写機構への知見Insights into chromatin transcription from RNA polymerase II–nucleosome structures
東京大学定量生命科学研究所クロマチン構造機能研究分野Laboratory of Chromatin Structure and Function, Institute for Quantitative Biosciences, The University of Tokyo ◇ 東京都文京区弥生1–1–1 ◇ 1–1–1 Yayoi, Bunkyo-ku, Tokyo 113–0032, Japan