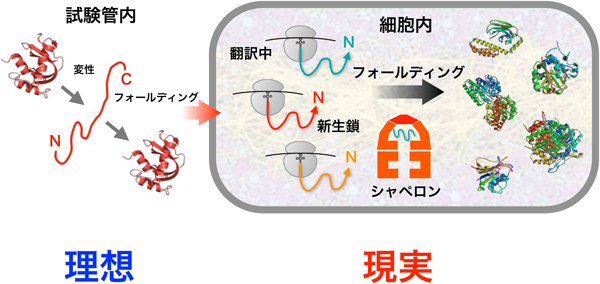
多くの生物のゲノム配列がすべて解読され,構造生物学が劇的に発展したことにより,タンパク質の種類の全貌や完成形である立体構造の多くが明らかとなってきている.しかるに,タンパク質がどのような過程を経て天然の立体構造を形成するかというフォールディング研究はどうであろうか? 1950年代に始まったAnfinsenらによる先駆的なフォールディング研究以来,半世紀以上の努力から多くの知見が得られてきたとはいえ,基本的には試験管内にてフォールディングしやすい「理想」的な挙動を示すタンパク質の研究に終始してきたと考えてよいのではなかろうか.
しかし,「現実」のタンパク質はいつも「理想」的にフォールディングするとは限らない.細胞内のように生体高分子で混み合った環境では,ちょっとした細胞内環境の撹乱によってフォールディングは破綻する.その結果,凝集体を形成したり,品質管理機構によって除去されたりする.生命はタンパク質の凝集を防ぐためにさまざまな分子シャペロンを準備して対処してきた1).また,その一方で多くのタンパク質がアミロイドやプリオンのような特殊な凝集体を形成し,それらが病気と関係することもわかってきている1).
本稿では,タンパク質フォールディングを「理想」と「現実」に分けて捉えた上で,凝集体,シャペロンについて我々が行ってきた研究を中心に概説するとともに,タンパク質の分子進化におけるフォールディングの役割についても議論したい.
2. フォールディングの「理想」:Anfinsenのドグマを出発点として
タンパク質フォールディング研究の歴史はAnfinsenらによる1950年代に遡ることができる.Anfinsenらは天然構造を持ったリボヌクレアーゼを尿素と還元剤で完全に変性させたあと,透析で尿素や還元剤を除くと元と同じ酵素活性を持ったリボヌクレアーゼが自発的に再生することを見いだした2)(図1).この簡単なフォールディング実験から導かれた「アミノ酸配列さえ決まればタンパク質の立体構造は一義に決定する」という結論はAnfinsenのドグマと呼ばれ,現在に至るまでフォールディング研究,ひいてはタンパク質科学全般の基礎となっている2).このドグマは,タンパク質の天然構造は熱力学的に最も安定な状態であるといい換えることもできる.つまり,「理想」としては,タンパク質は他からのエネルギーなどの助けなしに自発的にフォールディングできるはずということである.しかし,変性状態でのコンホメーションの多様性は膨大であり,熱力学な平衡を考えるだけではフォールディングがマイクロ秒から数十秒で完了する機構を説明できない(「Levinthalのパラドックス」と呼ばれる).そこで,速度論的な視点もフォールディングには必須である.
1)細胞内でのフォールディングの「現実」
上記のフォールディング研究は実験にせよ,計算科学にせよ,基本としてはタンパク質分子が単独でフォールディングすることを前提としており,他分子との相互作用を考えない.その意味では,物理や化学の教科書でおなじみの「単原子理想気体」になぞらえることができよう.物理化学ではまず理想気体の振る舞いを理論的に学んでから,現実に存在する「実在気体」の扱いを学ぶのが常である.
ここで,タンパク質フォールディングにこの「理想」と「現実(実在)」を当てはめてみよう.これまでの試験管内でのフォールディング研究の多くは(Anfinsenが使ったリボヌクレアーゼのように)非常にフォールディングしやすい単量体理想タンパク質を扱ってきたといえる.計算科学にしても,断りがない限りは理想状態を仮定している.つまり,これまでの多くのフォールディング研究で扱ってきたのは模範的で手のかからない優等生タンパク質であったという見方ができる.
しかし,細胞内に実在するタンパク質は理想的な状態からほど遠いのが現実だ(図1).細胞内はタンパク質濃度~300 mg/mLにも及ぶ混み合った状態であり,他の分子との相互作用を無視することはできない.また,Anfinsenが行ったような試験管内での古典的なフォールディング研究では完成したタンパク質をいったん変性したのちにフォールディングさせているので,細胞内での翻訳時におけるN末端からC末端へという方向性を反映していない.
では,タンパク質フォールディングにおける理想と現実の最も大きな違いは何であろうか.それは,物理化学における実在気体の扱いと同様,自分自身を含めた他の分子との相互作用である.ここでいう相互作用には,細胞内でタンパク質が機能するために必須のホモやヘテロでの多量体化,機能発現に必須の一過的な他タンパク質との相互作用などがある.このような機能発現に必要な相互作用に加えて,分子間の相互作用がフォールディングに障害を与える最大の問題として,フォールディング途上のポリペプチドどうしが不可逆に会合してしまう凝集形成があり,現実の細胞内フォールディングでは避けて通れない問題である.
実際,細胞内には凝集形成を防ぐ因子としてさまざまな種類のシャペロンが必ず存在する1).シャペロンが存在していたとしても,熱ショックなど細胞内環境のちょっとした変動によって細胞内のタンパク質恒常性(プロテオスタシス)が破綻し,結果としてタンパク質は凝集したり,凝集せずとも品質管理の対象となってユビキチン・プロテアソーム系など分解系に回されたりすることがわかっている1,3).
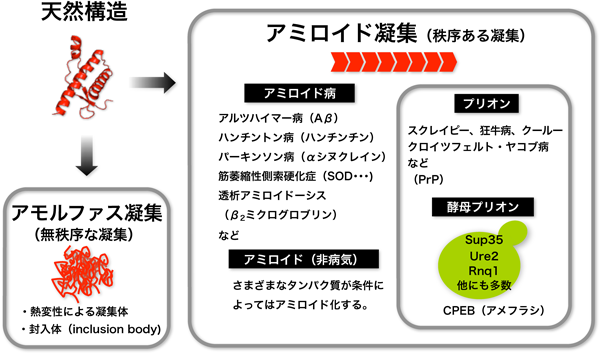
2)さまざまなタンパク質凝集体
タンパク質凝集体といっても,さまざまな凝集体が存在する(図2).
・アモルファスな凝集体
通常,タンパク質の凝集と聞いてまず思い浮かぶのは,試験管内でタンパク質溶液が白濁してしまう現象であろう.高温でタンパク質を保温した際にできる凝集体や,大腸菌で外来の組換えタンパク質を発現させたときにできる封入体(inclusion bodies)などが典型例である.ここでの凝集体は,熱変性の場合には天然の立体構造が熱で崩れて変性した状態,封入体の場合には翻訳直後の新生ポリペプチド(新生鎖)がまだ天然構造になる前の状態にて,変性ポリペプチドどうしが分子間で相互作用してできる.このときにできる凝集体は,特定の構造をとっていないという前提で,アモルファス(無定型,無構造)凝集体と呼ばれることが多い.アモルファス凝集体はありふれているとはいえ,この凝集体が何本くらいのポリペプチドから形成されているのか,どのような相互作用で絡み合っているのか,凝集体の中にどのくらい天然構造が残存しているのかなど,その詳細については実はよくわかっていない.一つには,アモルファス凝集体は本質的にヘテロな多量体分子であり,分子種の不均一性が詳細な生化学解析を困難にしている.
・アミロイド凝集体
アモルファス凝集体と違って,規則正しく並んだ凝集体としてアミロイドがよく知られている.アミロイドは分子間でβシートを形成し,結果的に線維状となるのが典型である.アミロイドには組織染色で用いられるコンゴレッドやチオフラビンTが特異的に結合する.もともとアミロイドはヒトを含む哺乳類の神経変性疾患の研究から見つかってきたタンパク質凝集体の一種であるが,近年では病気と関係のないタンパク質でもアミロイドになることが知られている4,5).感染性のタンパク質粒子として定義されるプリオンもアミロイド凝集の一種である6–8).出芽酵母のプリオン8–11)は病気どころか表現型の多様性を獲得するための戦略らしい12).これまでのところ,アミロイドは真核生物で多く見つかってきており,原核生物にはほとんど存在しないと考えられている.
なお,本稿の残りの部分では断りのない限りアミロイドやプリオンは扱わず,アモルファス凝集体について解説する.
3)新生鎖フォールディングと凝集形成
細胞内でのタンパク質フォールディングの「現実」は,翻訳に伴って合成されてくる際に直面する凝集体形成である.数千から数万種に及ぶ細胞内のタンパク質はいきなり完成するわけではなく,mRNAの情報がポリペプチド鎖へと変換される過程で,すべて翻訳途上の新生ポリペプチド鎖(新生鎖)の状態を経過する.従来,新生鎖はポリペプチド合成反応の単なる過渡的な中間体にすぎないと理解されてきたが,近年,新生鎖が自分自身のフォールディングや品質管理を制御すること,さらには,新生鎖自身が独自の生理機能を発揮して,細胞全体の生命現象の制御と調節に関わることがわかってきた13,14).すなわち,これまで受動的な反応中間体と考えられていた新生鎖そのものが積極的にさまざまな生命現象に関与している.さらに,新生鎖の成熟・品質管理機構の破綻が細胞の恒常性を撹乱し,さまざまな疾患の原因となっていることも明らかになりつつある.
この新生鎖が関わる現象の中で,ここでは翻訳時のフォールディングと競合して起こる凝集体形成について我々の研究を紹介する.
4)網羅的なタンパク質凝集体形成解析
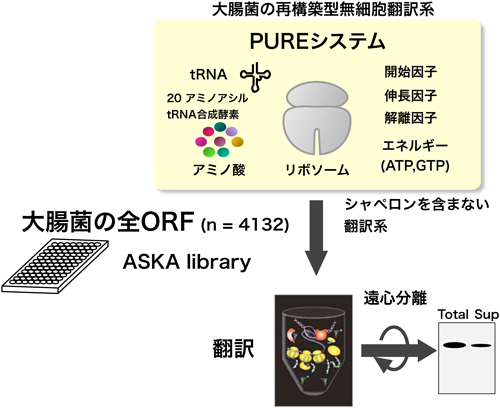
細胞内でのフォールディングは翻訳に共役しているとはいえ,翻訳時フォールディングを生化学的に調べるのは容易ではない.第一に,標準的な無細胞翻訳系は細胞を破砕して得られた粗抽出液を使うために,実験系内にフォールディングに影響を与える因子もすべて含まれている.具体的には,フォールディングを助ける因子であるシャペロンが含まれたままなので,本来は凝集しやすいタンパク質でもフォールディングしてしまう可能性がある.すなわち,タンパク質本来が持つ「素」の状態でのフォールディングの性質を調べられない.
そこで,再構築型の無細胞翻訳系であるPUREシステム15,16)を用いたフォールディング研究を行った(図3).PUREシステムは大腸菌の翻訳に必須の因子のみからなる翻訳系である15,16).シャペロンを含まないことから,調べたいタンパク質の「素」のフォールディング特性を翻訳と共役しながら調べることが可能なユニークな系である.
我々はまず,このPUREシステムを用いていくつかのモデルタンパク質を翻訳し,凝集しやすいか否か,またどのシャペロンが凝集抑制効果を持つのかについて解析を行い,翻訳時フォールディングにおけるPUREシステムの有効性を実証した17).
このPUREシステムを用いることで,通常できない翻訳に共役したフォールディング研究ができることがわかったので,フォールディング研究で取り残されていた問題である,どのタンパク質が理想的にフォールディングし,どのタンパク質が凝集しやすいのかについて数千種類のタンパク質,つまりプロテオームレベルで調べることとした.リソースとしては,大腸菌ゲノムでタンパク質をコードしている読み枠(ORF)全部,約4000種類を網羅したASKAライブラリー18)を使って,大腸菌すべてのタンパク質を個別にPUREシステムで合成し,翻訳産物のどのくらいが凝集するのかを一つ一つ遠心分離で調べた.
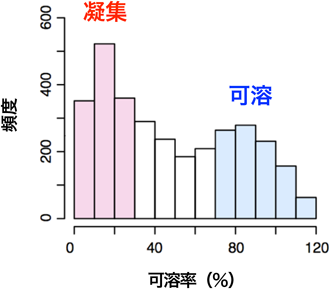
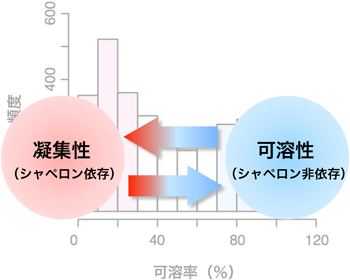
結果として,大腸菌全遺伝子の約7割にあたる3173種のタンパク質の凝集形成を遠心分離法で調べて溶けやすさ(可溶率)のデータを得た19).この3000種類以上のタンパク質による大規模解析の結果,タンパク質の凝集性分布はきれいな二峰性となることがわかった(図4).この網羅解析には膜タンパク質も含まれているが,膜タンパク質を除外してもこの二峰性には変化がなかった19).つまり,少なくとも大腸菌のプロテオームにおいて,タンパク質というのは溶けやすいグループと凝集しやすいグループに大別されるということである.
この解析により,どのタンパク質が凝集しやすいのかという点に関して,プロテオームレベルで解析できたといえる.この凝集性データとアミノ酸配列情報などから推測されるタンパク質の物性情報との相関をより詳細に調べてみたところ,以下のようなことがわかった19).
- ①分子量が小さいもの,等電点が低い(5~7)ものは可溶性が高い.
- ②負電荷を持つアミノ酸(Asp, Glu)が多いほど,また,芳香環を持つアミノ酸(Phe, Tyr, Trp)が少ないほど若干溶けやすい.
- ③疎水性アミノ酸(Val, Leu, Ile, Phe)の含量と凝集性には相関がない.
- ④非常に溶けやすい立体構造フォールド(たとえばチオレドキシンフォールド),凝集しやすいフォールド(たとえばTIMバレル)がある.
タンパク質の凝集は疎水相互作用によって引き起こされると一般にいわれているので,③については意外であった.また,④に関しても,立体構造というフォールディング後の「結果」がフォールディング途上に起こる凝集形成と関係するのは一見不思議だが,凝集が形成されるのはフォールディングが最終に近くなって特定の立体構造を特徴づけるかたちになってから起こることを示唆しているのであろう.
ただし,この凝集解析には多々注意点も存在する.たとえば,特定の遠心条件で沈殿になるかどうかだけで可溶率を得ており,可溶性画分に今回の遠心条件で沈殿しない多量体が存在する可能性は否定できない.また,可溶性になっているタンパク質が必ずしも最終的な天然構造になっていない可能性もある.さらには,ヘテロ多量体を形成するタンパク質の場合でも単独で合成している.実際,ヘテロ多量体を形成することがわかっているタンパク質はこの解析で凝集になりやすいようだ19).
注意点はあるものの,シャペロンがない同一条件下で,ある生物(大腸菌)のタンパク質すべてを翻訳してフォールディングを調べる試みにより,タンパク質が凝集しやすい集団と可溶性の集団に分かれることがわかった意義は大きいと考える.実際,この我々の網羅的な凝集体解析を元にさまざまなバイオインフォマティクス的な解析がなされてきている20–23).中でも,タンパク質の可溶性と,質量分析から得られた細胞内タンパク質の豊富さ(abundance)とが正の相関を持つことが明らかとなった20).つまり,溶けやすいタンパク質ほど細胞内に豊富に存在するということである.
1)Anfinsenのドグマからシャペロンへ
タンパク質は熱力学的に最も安定な立体構造に自発的にフォールディングするので,本来フォールディングは他の因子やエネルギーを必要としない.しかし,凝集体形成の危険に常にさらされている「現実」のフォールディングでは,凝集体を防ぐ因子が必要であり,その因子こそがシャペロン(chaperone)である24).シャペロンのもともとの定義は,「他のタンパク質の立体構造形成を助けるが,自らはその最終成分にならないタンパク質24)」であるが,現在ではタンパク質がその一生を全うするさまざまな過程(膜透過,品質管理,タンパク質分解など)における介添えタンパク質全般を指す用語として使われることも多い1).
代表的なシャペロンとしては,シャペロニン(GroEL-Hsp60,CCTなど),Hsp70-DnaK,Hsp90,Hsp104-ClpBファミリーなどの熱ショックタンパク質があげられる1,25).いずれもATPを使って多くの種類のタンパク質の構造形成や機能発現に関わっている.多くはフォールディングを助けるが,Hsp104-ClpBファミリーはHsp70系と連携して,いったんできた凝集体を脱凝集することができるスーパーシャペロンである.一方,ATPを使わないシャペロン(Hsp70の補助因子のHsp40-DnaJ,リボソーム結合型シャペロンのトリガー因子(Trigger Factor)26)や特定のタンパク質の構造形成を専属で助けるシャペロン(コラーゲンの成熟に必要なHsp4727)など)も数多く知られている.
細胞内にはさまざまなシャペロンが存在しており,作用機構もいろいろだが,基本的な作用は共通だ.シャペロンはフォールディング途上の不安定な状態や熱で変性したタンパク質が凝集にならないようにしている.図5にシャペロンがあると卵白の熱変性による凝集形成(すなわち「ゆで卵」)が防げるデモ実験を示す28).シャペロンとして働くタンパク質の多くは疎水性の高いペプチドに結合しやすいが,これはフォールディング途上や熱変性時に溶媒に露出した疎水的なアミノ酸領域にシャペロンが結合して,凝集を防ぐためである.親水的な球状タンパク質では疎水性アミノ酸が疎水性相互作用によってタンパク質内部でコアを形成することがフォールディングの駆動力となる.この疎水性相互作用が「分子内」で起これば自発的,すなわち理想的にフォールディングが進行するが,「分子間」で起これば凝集体を形成してフォールディングに失敗するという現実に直面する.タンパク質フォールディングは常に凝集体形成の危険と隣り合わせであり,細胞はその現実に対処して適応しなければならない.生命はシャペロンを準備して進化させることで,フォールディングの現実に適応してきたといえよう.
2)シャペロン効果の網羅的な解析
先に述べたように,我々はシャペロンを含まない無細胞翻訳系によって大腸菌の3000種類以上のタンパク質の溶けやすさ(可溶率)を調べた.可溶率30%以下の集団を凝集性と定義すると,凝集性タンパク質は約800種類にも及ぶが,これらの凝集性タンパク質は実際にシャペロンがあると可溶性になるのだろうか?
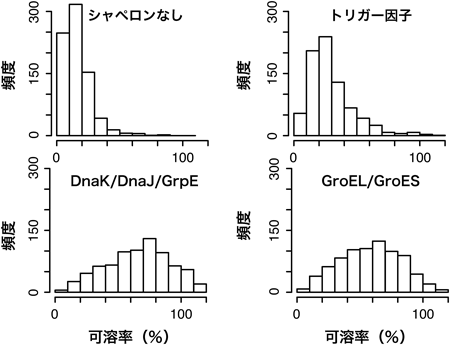
そこで,その約800種類の凝集性タンパク質をPUREシステムで翻訳する際にシャペロンを添加した解析を行った29)(図6).加えるシャペロンとしては,大腸菌での翻訳時フォールディングを助けることが知られているシャペロニンGroEL(Hsp60),DnaK(Hsp70),トリガー因子の三つのシャペロン系である.それぞれのシャペロンは細胞内での濃度に合わせた量を試験管内で加えるとともに,GroELとDnaKにはそれぞれ補助シャペロンも一緒に加えている(GroEL/GroES, DnaK/DnaJ/GrpE).これらの三つのシャペロン系を個別に加えて可溶率がどう変化するかをまず解析するとともに,必要に応じて複数のシャペロンを添加して効果を見た.結果を簡単にまとめると以下のようになった.
- ①GroELとDnaKは多くの凝集性タンパク質の凝集抑制に顕著に効果があった(図6).それに対して,トリガー因子はそれほどの効果がみられなかった.
- ②GroEL,DnaKそれぞれの「好み」が少しわかった.たとえば,GroELは分子量20,000~50,000のタンパク質に効果が高い.それに対し,DnaKは分子量の大きなタンパク質にも効果があった.
- ③GroEL,DnaKどちらを添加しても凝集抑制効果がなかったタンパク質に両シャペロンを一緒に加えると可溶性が高まること,それでも効果がないタンパク質に,トリガー因子をさらに加えるとほぼすべてが可溶になり,シャペロンの連携効果が示唆された.
このような大規模解析により,進化的によく保存されたシャペロンであるGroELやDnaKが実際に多種類のタンパク質の可溶化に貢献できることが定量的にわかった.シャペロンがないときの凝集解析と同様,この解析にも問題点がある.特に,可溶になったからといって最終的な天然構造になったかどうかについてははっきりしない.あるタンパク質が最終的に天然構造にフォールディングしたかどうかは酵素活性などタンパク質の機能で評価するのが最適だが,プロテオームレベルで多種多様なタンパク質の活性評価を行うのは現実的ではない.遠心分離に頼らないで可能な大規模フォールディング解析の開発が待たれる.
なお,ここまでに紹介した網羅的な凝集解析・シャペロン解析のデータはウェブ上でデータベースとして一般に公開されている(eSolデータベース:http,//www.tanpaku.org/tp-esol/).
5. シャペロンの作用機構:シャペロニンGroELを例として
シャペロンがどのような作用機構で働くのかについて多くの研究がなされてきている.中でも,進化的によく保存されたシャペロンであるシャペロニンとHsp70については大枠がわかってきたといえよう1,25).ここでは,シャペロン概念の確立にも貢献した主要シャペロンの一つであるシャペロニンGroELに関して,我々の研究を軸として解説する.
1)シャペロニンGroELの作用機構
・GroEL–GroES複合体の立体構造—空洞のあるリング構造
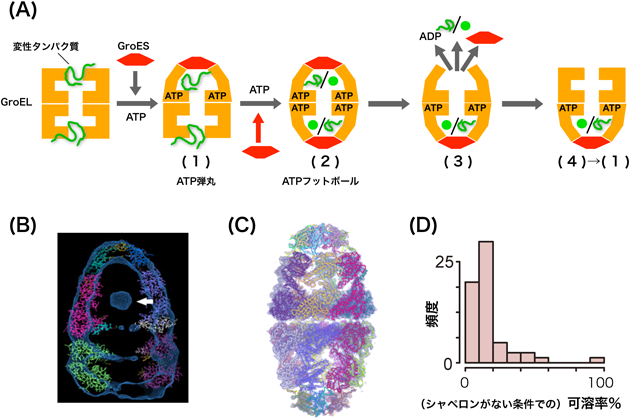
GroELは,57 kDaのサブユニット七つからなるリングが背中合わせに二つ重なった14量体構造(総分子量~800,000)を形成しており,リング内部にはそれぞれ直径約45 Åの空洞がある.ATPを結合したGroELは補助因子のGroESと結合する.ヌクレオチドが結合したGroELサブユニットは開いた構造となり,その先にドーム型構造のGroESがフタのように結合する30)(図7A).GroESが結合したGroELリング(シスGroELリング)内には体積が広がった大きな空洞が生まれる(図7B).このGroELとGroESの空洞内には分子量60,000くらいまでのポリペプチドが格納可能である31)(図7B).
GroEL–GroES複合体には2種類ある.GroELダブルリングの一方にGroESが結合した非対称複合体(その形状から「弾丸」型構造と呼ばれる32)),GroELリングのそれぞれにGroESが結合した対称型複合体(「フットボール」型複合体と呼ばれる)である.フットボール型複合体に関しては我々を含めた複数のグループがごく最近立体構造を決定した33,34)(図7C).
・GroEL–GroESの反応サイクル—ATP加水分解タイマーで制御されたゆりかご
リングの入り口付近に基質タンパク質が結合したGroELに七つのATPが結合すると構造変化が起こってGroESが結合し,基質タンパク質はGroELとGroESが形成するシスリングの空洞内に落とし込まれる35,36)(図7A(1)).ATPが加水分解してADPになるまでの時間(約8秒)が反応サイクルのタイマーとして働く.GroELとGroESの結合解離を1分子レベルで調べる実験の解析結果より,この8秒のタイマーはGroESが結合してから基質タンパク質が空洞内に落し込まれるまでの3秒とフォールディングが進行する5秒の2段階に分けられると考えられた37,38).GroELは基質タンパク質が結合した状態でATP加水分解が速くなる性質を有しており39),基質タンパク質存在下で効率的にフォールディングを助けることができる28).
・フットボールか弾丸か
GroELとGroESの複合体には弾丸型とフットボール型の2種類が存在しうる.上記の反応サイクルを含めて,GroELの総説の多くや教科書では弾丸型複合体のみを経由した反応機構が記載されている.フットボール型複合体は早くから電子顕微鏡観察にて確認されていたが32,40,41),1990年代のGroELの作用機構を巡る激しい競争の中で弾丸型複合体のみが重要な中間体であると認識されるようになっていた.しかし,2008年になって,我々は以前の弾丸型構造を前提にしていた生化学実験の不備を突きとめてフットボール型複合体の重要性を指摘する42)とともに,船津らは我々と独立にフットボール型複合体がGroELの反応サイクル中に蓄積すること43,44),さらには,基質タンパク質存在下ではフットボール型複合体が優先的に形成されること45)を見いだした(図7A).
フットボール型構造ではGroELの二つのリング間の相互作用が弾丸型構造と若干異なっている33,34)と同時に,リング間の接触部位が不安定になっており,ATP存在下でGroELの二層リングが一層に分割する四次構造変化(リング間のスプリット)が電気泳動で観察できる34).このスプリットは好熱菌Thermus thermophilusのシャペロニンGroEL–GroES複合体などで観察されている46,47).GroELの四次構造変化については細胞内も含めてまだ十分に理解されていない.GroELは基質タンパク質の有無によって,弾丸型とフットボール型の二つのサイクルを使い分けている可能性もあり,この二つのモデルは相互排他的ではないかもしれない.
2)細胞内でのGroELの役割
細胞内でのGroELの役割を解説する前に,翻訳途上の新生鎖がどのようなシャペロンに助けられているのかについて流布しているモデル(大腸菌の場合)を紹介する1).翻訳中のリボソームにはトリガー因子というリボソーム結合型シャペロンが新生鎖を待ち構えている.トリガー因子とDnaKは遺伝学的解析から機能が重複していることが知られており,トリガー因子とDnaKは翻訳途上の新生鎖が凝集体にならないよう防いでいると考えられている48,49).GroELとGroESは空洞を形成して基質タンパク質を格納するという点から翻訳終了後のポリペプチドに作用するというのが通説であり,その場合トリガー因子もしくはDnaKの後にGroELが関わるといわれている.しかし,我々は,GroELが細胞内,もしくは無細胞翻訳系にて新生鎖と相互作用することを見いだしている50,51).
では,実際の細胞内ではどのようなタンパク質がGroELに助けられているのであろうか.我々は細胞内でシャペロニンGroELがないとフォールディングできずに凝集したり,分解したりしてしまうタンパク質(細胞内GroEL基質)の研究に取り組み52,53),最終的に約60個の細胞内GroEL基質を同定した53).細胞内GroEL基質は次に示すような性質を有していた.
- ①前節で述べたシャペロンがない条件での可溶率データと照合すると,細胞内GroEL基質はほぼすべてが非常に凝集しやすいタンパク質であった(図7D).
- ②細胞内GroEL基質の分子量分布は40,000をピークとした正規分布で最大70,000弱であった.これはGroELと補助因子GroESが作る空洞にほぼ収まる分子量の範囲であり,シャペロン複合体が作る空洞が細胞内で非常に重要であることを示唆する.実際,GroELとGroESが形成する空洞は細胞にとって必須のフォールディング空間であることが変異体解析から示されている54).
- ③細胞内GroEL基質の7割ほどが代謝関連タンパク質であった.
もう一つの主要シャペロンであるDnaKについてもインタラクトーム解析から細胞内での基質タンパク質について報告22)があり,(GroELほど顕著ではないが)凝集性のタンパク質がDnaK基質となるようだ22).以上をまとめると,シャペロンは細胞内に存在する凝集しやすいタンパク質集団が凝集しないようにするのが第一の役割ということがあらためて確認されたといえる.
3)GroEL研究で残された課題
ここまでの説明でのGroELの作用機構の本質は,フォールディングのための安全な空間と時間を提供するものであって,フォールディングそのものには影響しない,というものである.しかし,GroELとGroESの空洞内で起こるフォールディングは自発的フォールディングよりも速く,フォールディング経路が溶液内での自発的なそれとは違うという報告が出てきている55–58).
大腸菌ではGroELの基質タンパク質の全容が解明されてきた.今後は,GroELがなければ天然構造に到達できえないタンパク質が細胞内に存在するのか,ということが焦点になろう.フォールディングが完全にGroELに依存してしまい自発的にフォールディングできなくなってしまったタンパク質が見つかれば,そのタンパク質では“Anfinsenのドグマ”は成立しないのかもしれない.
ここまで書いてきたように,フォールディングの観点からのタンパク質の理想はシャペロンの助けを借りずに自分でフォールディングすることだが,現実にはシャペロンなしでは凝集してしまうタンパク質が相当な割合で存在する.実際,GroELの細胞内基質タンパク質は凝集しやすい性質を持つ.
網羅的な凝集解析でわかった最も重要な知見は,タンパク質にはフォールディングしやすい集団と凝集しやすい集団の二つが存在していることであった19).この二峰性について興味深いのは,アミノ酸配列の相同性が高いタンパク質どうしで,一方は凝集性,もう一方は可溶性というホモログペアが多数存在することがわかってきたことである53).このようなペアが存在するということは,凝集性と可溶性は互いに相互変換可能であることを示唆する(図8).つまり,理想フォールディングをするタンパク質を凝集性にする,もしくはその逆で,現実には凝集しやすいタンパク質を理想的なタンパク質にすることは可能だろうか.
1)GroEL非依存(可溶性)からGroEL依存(凝集性)タンパク質への変換
GroELは生育に必須のシャペロンであり,ごく一部の例外を除いてすべての細胞に存在する.その例外が,ゲノムサイズが小さいことで知られるマイコプラズマ/ウレアプラズマの一部で,ゲノム解析からgroE遺伝子を持たないマイコプラズマ/ウレアプラズマが見つかっている59,60).我々はgroE遺伝子を持たないUreaplasma urealyticumのゲノム内に大腸菌GroELの細胞内基質タンパク質のオーソログが複数あることを見つけた.そこで,そのオーソログをクローニングし,大腸菌内でのフォールディングが大腸菌GroELに依存しているかどうかを調べた.その結果,ウレアプラズマの大腸菌GroEL基質オーソログは試したすべて(MetK, DeoA, YfcH)が大腸菌GroELがなくてもフォールディングできる,すなわちGroEL非依存であることを見いだした53).
その中の一つMetKはウレアプラズマと大腸菌でアミノ酸配列の43%が同一と非常によく似ているにも関わらず,GroEの依存性が異なるので,GroELの依存性の変換が可能だと考えた.詳細な変異体解析,ランダム変異によるスクリーニングなどの結果,ウレアプラズマMetKは点変異レベルで細胞内で大腸菌GroEL依存性になることが判明した61).しかも,その点変異は独立に複数存在する.興味深いことに,GroEL依存性に変換したウレアプラズマMetK変異体はフォールディング途上で凝集しやすくなっていた61).以上から,GroEL依存性と凝集性には正の相関があることがあらためてわかった.
2)GroEL依存(凝集性)からGroEL非依存(可溶性)タンパク質への変換
タンパク質にランダムに変異が入ったとき,ほとんどの変異は(点変異であっても)タンパク質の不安定化につながること62)が知られている.不安定化は結果として凝集性につながることが多いので,上記のウレアプラズマMetKが凝集性に変換するのは驚くべきことではない.では,GroELに依存している凝集性の基質タンパク質をGroEL非依存(可溶性)に変換することは可能だろうか.
我々は,GroEL基質タンパク質がGroEL欠乏下で可溶に変換したものを見つけだすスクリーニング系を確立した.GroEL基質タンパク質の一つにランダム変異を導入して,そのスクリーニング系で可溶になった変異体を探したところ,~10種類程度の点変異レベルのGroEL非依存変異体が取得できた(白濱ら,投稿準備中).
以上の結果をまとめると,GroELの依存性,ひいてはタンパク質の凝集性はかなり微妙な差異だけで決まりうること,しかも,一つのタンパク質に複数のホットスポットがあることが明らかとなった.まだごくわずかのモデルタンパク質だけの結果ではあるが,拡張すると,タンパク質の可溶性と凝集性は相互に変換可能であることが示唆された.
本稿のここまでの流れを整理してみる.①プロテオームレベルでの凝集体解析から,細胞内タンパク質は可溶性と凝集性の二つの集団に分かれる.30%ほどのタンパク質は理想的にはフォールディングできず凝集になりやすい.②GroELやDnaKといった主要シャペロンはグローバルに凝集を防ぐ.③細胞内でGroELの世話になっている基質タンパク質は実際に凝集になりやすい.④凝集性と可溶性の差異はごくわずかで,相互変換可能らしい.
以上をまとめて,タンパク質フォールディングの「理想」と「現実」について考察してみる.
なぜ理想タンパク質だけにならないのか?
凝集性と可溶性の差異がわずかであれば,タンパク質の分子進化の過程でいずれは凝集性の性質はなくなって,可溶性になってもよさそうなものである.シャペロンの多くはATPを使うので,本来必要のないエネルギーコストを使ってまで凝集性を残しておくのはアドバンテージがあるのではなかろうか.
ここで,シャペロンによるタンパク質の分子進化の緩衝作用について紹介する.分子進化の中立説を元にすると,酵素機能の向上や新規の機能の獲得などタンパク質が進化するためには,多様な変異体を準備しておく必要がある.タンパク質の安定性はぎりぎりに保たれているのがふつうなので,タンパク質変異のほとんどは不安定化につながる63).不安定化は凝集につながるので,せっかく変異が中立に導入されても,細胞内でフォールディングできなければ多様性を生み出せない.つまり,新規機能の獲得などタンパク質の進化は不安定化とのトレードオフの関係にある.そこで,シャペロンがタンパク質の凝集を防ぐことで,本来なら不安定になる変異を許容する,つまり緩衝作用によって多様性が保証される,という説が提案された64).この進化におけるシャペロンの緩衝作用はLindquistらがショウジョウバエのHsp90の研究をきっかけに提案した仮説であるが,GroELにも拡張されてきている63,65,66).
このシャペロンの緩衝作用を考慮すると,凝集しやすいタンパク質は「あえて」凝集性のままなのではないかという考え方になる.凝集性の集団が明確に残っていて,シャペロンを必要とするのはその方が進化的な観点からは有利ということだ.細胞内GroEL基質の7割ほどが代謝酵素であるが53),細胞内GroEL基質タンパク質は代謝ネットワークの中心経路から離れた周縁に存在するものが多いらしい67).代謝ネットワークの外側に位置するほど進化的に新しい酵素が多いらしく,シャペロンの進化における役割に合致する知見である.
フォールディングの「現実」問題はさらに拡張している.本稿ではほとんどふれなかったアミロイドやプリオンは病気に関係するタンパク質以外でも続々と見つかってきている.たとえば,出芽酵母では少なくとも10種類以上のプリオンが見つかってきている68).しかも,出芽酵母のプリオンは細胞にとって有害ではなく,生育環境によってはプリオンになった方が有利に働くケースが多々あることがわかってきている69,70).
また,単独では立体構造をとることができない天然変性タンパク質という概念も多くのフォールディング研究者が参入し,定着しつつある71).
冒頭にて,「これまでのフォールディング研究は,フォールディングしやすい「理想」的な挙動を示すタンパク質の研究に終始してきた」のではないかと書いた.本稿では,フォールディングと凝集を理想と現実の対比で考えてきたが,アミロイド/プリオンや天然変性タンパク質を考えると,実際のタンパク質ワールドは我々が考える以上に奥深いものではないだろうか.
現代物理学の未解明の大問題として,暗黒物質/暗黒エネルギーがある.いわく,我々が知る通常の物質は宇宙全体ではほんの数%を占めるにすぎず,残りの90%以上は正体不明の暗黒物質/暗黒エネルギーが占めているらしい.細胞という小宇宙の中のタンパク質ワールドにも暗黒物質的な状態がまだまだ潜んでいるかもしれない,というと大げさに聞こえるかもしれない.しかし,これまでのフォールディング研究はタンパク質ワールドの「氷山の一角」しか対象としてこなかったというのは間違いのないところである.多くのタンパク質の立体構造が続々と決定している今,これまで見過ごされてきたフォールディングが困難な現実のタンパク質の研究に多面的に取り組むのは喫緊の課題であろう.
謝辞Acknowledgments
本稿で紹介したシャペロン研究は,筆者が大学院入学時に吉田賢右教授(東京工業大学名誉教授)にきっかけをもらって始めたものである.ここに心より感謝申し上げます.また,ここで示したシャペロンや凝集体の研究は非常に多くの共同研究者,大学院生,学部生との相互作用による新しいアイディア,材料,手法の賜です.この場を借りて御礼申し上げます.
引用文献References
1) Hartl, F.U., Bracher, A., & Hayer-Hartl, M. (2011) Nature, 475, 324–332.
2) Anfinsen, C.B. (1973) Science, 181, 223–230.
3) Powers, E.T., Morimoto, R.I., Dillin, A., Kelly, J.W., & Balch, W.E. (2009) Annu. Rev. Biochem., 78, 959–991.
4) Dobson, C.M. (2003) Nature, 426, 884–890.
5) Knowles, T.P., Vendruscolo, M., & Dobson, C.M. (2014) Nat. Rev. Mol. Cell Biol., 15, 384–396.
6) Prusiner, S.B. (1998) Proc. Natl. Acad. Sci. USA, 95, 13363–13383.
7) Prusiner, S.B. (2013) Annu. Rev. Genet., 47, 601–623.
8) Tuite, M.F. & Serio, T.R. (2010) Nat. Rev. Mol. Cell Biol., 11, 823–833.
9) Taguchi, H. (2006)生化学,78, 1088–1091.
10) Taguchi, H. & Kawai-Noma, S. (2010) FEBS J., 277, 1359–1368.
11) Liebman, S.W. & Chernoff, Y.O. (2012) Genetics, 191, 1041–1072.
12) Newby, G.A. & Lindquist, S. (2013) Trends Cell Biol., 23, 251–259.
13) Ito, K. & Chiba, S. (2013) Annu. Rev. Biochem., 82, 171–202.
14) Pechmann, S., Willmund, F., & Frydman, J. (2013) Mol. Cell, 49, 411–421.
15) Shimizu, Y., Inoue, A., Tomari, Y., Suzuki, T., Yokogawa, T., Nishikawa, K., & Ueda, T. (2001) Nat. Biotechnol., 19, 751–755.
16) Shimizu, Y., Kuruma, Y., Kanamori, T., & Ueda, T. (2014) Methods Mol. Biol., 1118, 275–284.
17) Ying, B.W., Taguchi, H., Ueda, H., & Ueda, T. (2004) Biochem. Biophys. Res. Commun., 320, 1359–1364.
18) Kitagawa, M., Ara, T., Arifuzzaman, M., Ioka-Nakamichi, T., Inamoto, E., Toyonaga, H., & Mori, H. (2005) DNA Res., 12, 291–299.
19) Niwa, T., Ying, B.W., Saito, K., Jin, W., Takada, S., Ueda, T., & Taguchi, H. (2009) Proc. Natl. Acad. Sci. USA, 106, 4201–4206.
20) Castillo, V., Grana-Montes, R., & Ventura, S. (2011) Biotechnol. J., 6, 752–760.
21) Agostini, F., Vendruscolo, M., & Tartaglia, G.G. (2012) J. Mol. Biol., 421, 237–241.
22) Calloni, G., Chen, T., Schermann, S.M., Chang, H.C., Genevaux, P., Agostini, F., Tartaglia, G.G., Hayer-Hartl, M., & Hartl, F.U. (2012) Cell Reports, 1, 251–264.
23) Fang, Y. & Fang, J. (2013) Mol. Biosyst., 9, 806–811.
24) Ellis, J. (1987) Nature, 328, 378–379.
25) Richter, K., Haslbeck, M., & Buchner, J. (2010) Mol. Cell, 40, 253–266.
26) Hoffmann, A., Bukau, B., & Kramer, G. (2010) Biochim. Biophys. Acta, 1803, 650–661.
27) Ishida, Y. & Nagata, K. (2011) Methods Enzymol., 499, 167–182.
28) Taguchi, H. (2005) J. Biochem., 137, 543–549.
29) Niwa, T., Kanamori, T., Ueda, T., & Taguchi, H. (2012) Proc. Natl. Acad. Sci. USA, 109, 8937–8942.
30) Xu, Z., Horwich, A.L., & Sigler, P.B. (1997) Nature, 388, 741–750.
31) Sakikawa, C., Taguchi, H., Makino, Y., & Yoshida, M. (1999) J. Biol. Chem., 274, 21251–21256.
32) Taguchi, H., Konishi, J., Ishii, N., & Yoshida, M. (1991) J. Biol. Chem., 266, 22411–22418.
33) Fei, X., Ye, X., LaRonde, N.A., & Lorimer, G.H. (2014) Proc. Natl. Acad. Sci. USA, 111, 12775–12780.
34) Koike-Takeshita, A., Arakawa, T., Taguchi, H., & Shimamura, T. (2014) J. Mol. Biol., 426, 3634–3641.
35) Weissman, J.S., Hohl, C.M., Kovalenko, O., Kashi, Y., Chen, S., Braig, K., Saibil, H.R., Fenton, W.A., & Horwich, A.L. (1995) Cell, 83, 577–587.
36) Mayhew, M., da Silva, A.C., Martin, J., Erdjument-Bromage, H., Tempst, P., & Hartl, F.U. (1996) Nature, 379, 420–426.
37) Taguchi, H., Ueno, T., Tadakuma, H., Yoshida, M., & Funatsu, T. (2001) Nat. Biotechnol., 19, 861–865.
38) Ueno, T., Taguchi, H., Tadakuma, H., Yoshida, M., & Funatsu, T. (2004) Mol. Cell, 14, 423–434.
39) Aoki, K., Motojima, F., Taguchi, H., Yomo, T., & Yoshida, M. (2000) J. Biol. Chem., 275, 13755–13758.
40) Azem, A., Kessel, M., & Goloubinoff, P. (1994) Science, 265, 653–656.
41) Schmidt, M., Rutkat, K., Rachel, R., Pfeifer, G., Jaenicke, R., Viitanen, P., Lorimer, G., & Buchner, J. (1994) Science, 265, 656–659.
42) Koike-Takeshita, A., Yoshida, M., & Taguchi, H. (2008) J. Biol. Chem., 283, 23774–23781.
43) Sameshima, T., Ueno, T., Iizuka, R., Ishii, N., Terada, N., Okabe, K., & Funatsu, T. (2008) J. Biol. Chem., 283, 23765–23773.
44) Sameshima, T., Iizuka, R., Ueno, T., Wada, J., Aoki, M., Shimamoto, N., Ohdomari, I., Tanii, T., & Funatsu, T. (2010) J. Biol. Chem., 285, 23159–23164.
45) Sameshima, T., Iizuka, R., Ueno, T., & Funatsu, T. (2010) Biochem. J., 427, 247–254.
46) Ishii, N., Taguchi, H., Sasabe, H., & Yoshida, M. (1995) FEBS Lett., 362, 121–125.
47) Taguchi, H., Amada, K., Murai, N., Yamakoshi, M., & Yoshida, M. (1997) J. Biol. Chem., 272, 18155–18160.
48) Deuerling, E., Schulze-Specking, A., Tomoyasu, T., Mogk, A., & Bukau, B. (1999) Nature, 400, 693–696.
49) Teter, S.A., Houry, W.A., Ang, D., Tradler, T., Rockabrand, D., Fischer, G., Blum, P., Georgopoulos, C., & Hartl, F.U. (1999) Cell, 97, 755–765.
50) Ying, B.W., Taguchi, H., Kondo, M., & Ueda, T. (2005) J. Biol. Chem., 280, 12035–12040.
51) Ying, B.W., Taguchi, H., & Ueda, T. (2006) J. Biol. Chem., 281, 21813–21819.
52) Fujiwara, K. & Taguchi, H. (2007) J. Bacteriol., 189, 5860–5866.
53) Fujiwara, K., Ishihama, Y., Nakahigashi, K., Soga, T., & Taguchi, H. (2010) EMBO J., 29, 1552–1564.
54) Koike-Takeshita, A., Shimamura, T., Yokoyama, K., Yoshida, M., & Taguchi, H. (2006) J. Biol. Chem., 281, 962–967.
55) Brinker, A., Pfeifer, G., Kerner, M.J., Naylor, D.J., Hartl, F.U., & Hayer-Hartl, M. (2001) Cell, 107, 223–233.
56) Tang, Y.C., Chang, H.C., Roeben, A., Wischnewski, D., Wischnewski, N., Kerner, M.J., Hartl, F.U., & Hayer-Hartl, M. (2006) Cell, 125, 903–914.
57) Chakraborty, K., Chatila, M., Sinha, J., Shi, Q., Poschner, B.C., Sikor, M., Jiang, G., Lamb, D.C., Hartl, F.U., & Hayer-Hartl, M. (2010) Cell, 142, 112–122.
58) Georgescauld, F., Popova, K., Gupta, A.J., Bracher, A., Engen, J.R., Hayer-Hartl, M., & Hartl, F.U. (2014) Cell, 157, 922–934.
59) Glass, J.I., Lefkowitz, E.J., Glass, J.S., Heiner, C.R., Chen, E.Y., & Cassell, G.H. (2000) Nature, 407, 757–762.
60) Clark, G.W. & Tillier, E.R. (2010) Biochem. Cell Biol., 88, 185–194.
61) Ishimoto, T., Fujiwara, K., Niwa, T., & Taguchi, H. (2014) J. Biol. Chem., 289, 32073–32080.
62) Tokuriki, N., Stricher, F., Serrano, L., & Tawfik, D.S. (2008) PLOS Comput. Biol., 4, e1000002.
63) Tokuriki, N. & Tawfik, D.S. (2009) Nature, 459, 668–673.
64) Rutherford, S.L. & Lindquist, S. (1998) Nature, 396, 336–342.
65) Fares, M.A., Ruiz-Gonzalez, M.X., Moya, A., Elena, S.F., & Barrio, E. (2002) Nature, 417, 398.
66) Bogumil, D. & Dagan, T. (2012) Biochemistry, 51, 9941–9953.
67) Takemoto, K., Niwa, T., & Taguchi, H. (2011) BMC Syst. Biol., 5, 98.
68) Alberti, S., Halfmann, R., King, O., Kapila, A., & Lindquist, S. (2009) Cell, 137, 146–158.
69) Suzuki, G., Shimazu, N., & Tanaka, M. (2012) Science, 336, 355–359.
70) Halfmann, R., Jarosz, D.F., Jones, S.K., Chang, A., Lancaster, A.K., & Lindquist, S. (2012) Nature, 482, 363–368.
71) Dyson, H.J. & Wright, P.E. (2005) Nat. Rev. Mol. Cell Biol., 6, 197–208.
72) Kanno, R., Koike-Takeshita, A., Yokoyama, K., Taguchi, H., & Mitsuoka, K. (2009) Structure, 17, 287–293.
73) Koike-Takeshita, A., Mitsuoka, K., & Taguchi, H. (2014) J. Biol. Chem., 289, 30005–30011.
著者紹介Author Profile
 田口 英樹(たぐち ひでき)
田口 英樹(たぐち ひでき)東京工業大学大学院生命理工学研究科教授.博士(理学).
略歴1967年兵庫県に生る.89年東京工業大学工学部卒業.93年同大学院総合理工学研究科博士課程修了.東京工業大学資源化学研究所助手,東京大学新領域創成科学研究科助教授(准教授)を経て,2010年より現職.02~06年JSTさきがけ研究者を兼任.
研究テーマと抱負シャペロンの作用機構,プリオンやアミロイドを含むタンパク質凝集体の形成機構,細胞内タンパク質動態,新生鎖の生物学.
ウェブサイトhttp://www.taguchi.bio.titech.ac.jp(研究室ウェブサイト),http://taguchi-hideki.blogspot.com(ブログ「田口英樹のサプリメント」)
趣味街の散策.