DNA複製の際,DNAポリメラーゼが誤った残基を取り込むことがある.これらの誤りがDNAポリメラーゼ自身の持つ校正機構をすり抜けて残ると,対合する塩基を持たないアンペア塩基あるいは通常とは異なる塩基どうしが対合したミスペア塩基を生じる.両者はまとめてミスマッチ塩基と呼ばれる.ミスマッチ塩基を形成する二本鎖のうち,新生鎖側の配列は次のDNA複製を経て変異として固定され,その蓄積は細胞の老化やがん化につながる可能性がある.細菌からヒトまで,ほとんどの生物ではDNAミスマッチ修復系(mismatch repair: MMR)と呼ばれる反応機構が存在し,DNA複製直後に生じたミスマッチ塩基を修復することで,複製の忠実度を1000倍近く高めている1).ヒトではMMR関連遺伝子の変異やエピジェネティックなサイレンシングがリンチ症候群の主要な原因である2).リンチ症候群は大腸,子宮内膜,胃,卵巣などにおけるがんの若年性発症を特徴とし,全大腸がんの約5%程度に関与していると推測されている.MMR関連タンパク質群の機能解析によりリンチ症候群発症メカニズムの分子レベルでの理解が進むと期待される.
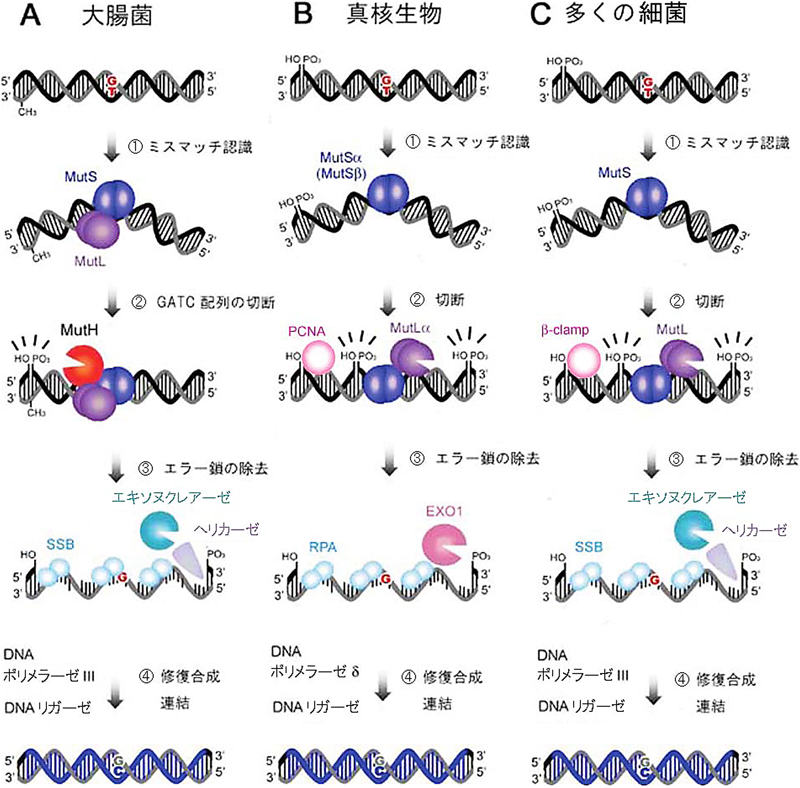
MMRはヌクレオチド除去反応を伴い,エラーを含むヌクレオチド鎖をいったん除去した後,再合成を行うことで修復する.他のDNA傷害と異なり,ミスマッチ塩基そのものは正常な塩基によって形成されており,二本鎖DNAのうちどちらの鎖を除去するべきであるかという情報を与えない.そのため細胞は,複製ミスを含むはずの新生鎖側を除去する仕組みを備えている.現在までに知られているMMRは,ヒト型のものと大腸菌型のものに大別される3,4).両者の最も大きな違いは新旧鎖を識別する戦略にある.真核生物型と大腸菌型のどちらにおいても新旧鎖識別の中心を担うのはエンドヌクレアーゼであり,この酵素がニックを入れた鎖が除去反応の対象となる(図1).
大腸菌型MMR(図1A)では,MutSがミスマッチ塩基対を認識し(①のステップ),MutLと相互作用する.MutS–MutL複合体は制限酵素であるMutHを活性化し,その認識部位であるGATC配列が切断される(②のステップ).鋳型鎖側のGATC配列は修飾酵素Damによるメチル化を受けて切断を逃れるが,新生鎖はまだ修飾を受けておらず,MutHは新生鎖特異的にニックを入れることになる.MutHが導入したニックを足がかりに,ヘリカーゼ,エキソヌクレアーゼがエラーを含むヌクレオチドを除去し(③のステップ),修復合成が行われる(④のステップ).
真核生物においても,ミスマッチ塩基はMutSホモログ(MutSαやMutSβ)によって認識され,MutLホモログ(MutLαやMutLβ)が相互作用することがわかっていた.しかし,制限酵素MutHは大腸菌およびその近縁種にしか存在せず,ヒトを含めた多くの生物ではMMRの概要が長らく未解明であった.
真核生物MMRの研究は,ミスマッチを導入したプラスミドをモデル基質とし,核抽出液中のミスマッチ修復活性を調べることで進められてきた.まず,プラスミドにあらかじめニックやギャップを持たせておくことで,除去反応が起こる鎖を指定できることが示された.つまり,DNA鎖の末端が新生鎖のシグナルとして機能したのである.細胞内のDNA複製では新生鎖にリーディング鎖やラギング鎖の末端が豊富に存在することから,これらをシグナルとして利用している可能性が考えられる.
ミスマッチ含有プラスミドの修復では,あらかじめ存在するニックからミスマッチまでのヌクレオチド鎖が除去されるが,その際,ニックはミスマッチの5′側と3′側のどちらに存在してもよく,ニックからみてミスマッチに近い方の経路がヌクレオチド除去反応の対象となる.これは,MMRが5′MMR(ミスマッチの5′側にニックがある際の修復)と3′MMRという二つの反応経路を持ち,除去反応が5′方向にも3′方向にも進むことができるためであると推測された.ところが奇妙なことに,5′と3′MMRの両方でEXO1の5′-3′エキソヌクレアーゼ活性が必要であることが明らかとなった.大腸菌型MMRにおいても,GATC配列とミスマッチの位置関係に対応して5′または3′のどちらか一方向のヌクレオチド除去反応が選択されるが,これは,大腸菌MMRにおいては方向性の異なる複数のエキソヌクレアーゼが機能するためであるとわかっている.真核生物ではなぜ1種類のエキソヌクレアーゼによって両方向の除去が可能なのか.
ブレークスルーとなる発見は,ヒト由来のタンパク質群を用いた部分再構成系の研究によりもたらされた.ヒトMutLがエンドヌクレアーゼ活性を持ち,MutSホモログ,PCNA,RFC,アデノシン5′-三リン酸(adenosine 5′-triphosphate: ATP)などに依存して,ミスマッチ塩基の両側を切断したのである5).これによってミスマッチ部位を含む一本鎖DNAのパッチが生じるため,5′と3′MMRの除去反応は共通の反応となり,1種類のエキソヌクレアーゼにより遂行できる.加えて,MutLによる切断は,二本鎖DNAのうち,ニックなど不連続性を持つ鎖に導入されていた.不連続鎖特異的な切断にはDNAにロードされた状態のPCNAが必要であることも示され6),ラギング鎖やリーディング鎖の3′末端を新生鎖識別シグナルとしてPCNAがMutLに伝達していると考えられた.平行して,不連続性のシグナルであるニックがミスマッチのどちら側に位置するかによってMutLの貢献度が異なることが示され,ニックがミスマッチの5′側に存在する場合にはMutLに依存しないMMR経路も存在すると考えられている.
MutLはN末端側のATP加水分解ドメインとC末端側の二量体形成ドメインからなる.N末端ドメインはヒトMutLと大腸菌MutL間で非常に相同性が高く,ほぼ同じといってよい.一方,C末端ドメインは両者で大きく異なる.ヒトMutL C末端ドメインには,大腸菌MutLにはみられないDQHA(X)2E(X)4モチーフが存在し,二価金属イオンを配位してヌクレアーゼ活性に関わる.大多数の真核生物および細菌MutLのC末端ドメインにもこのヌクレアーゼモチーフが保存されている(表1).
表1 ヒト,高度好熱菌,大腸菌MutLの比較 | 会合体 | C末端ドメインのエンドヌクレアーゼモチーフ | N末端ドメインのATPaseモチーフ | C末端ドメインのPCNAまたはβ-クランプ相互作用モチーフ |
|---|
| ヒトMutLα | ヘテロ二量体 | ○ | ○ | ○ |
| 高度好熱菌MutL | ホモ二量体 | ○ | ○ | × |
| 大腸菌MutL | ホモ二量体 | × | ○ | ○ |
酵母やさまざまな細菌由来MutLがエンドヌクレアーゼ活性を持つこと7),また,細菌におけるPCNAのカウンターパートであるβ-クランプがMutLのエンドヌクレアーゼドメインと相互作用することも実験的に確かめられ,大腸菌型MMRはまれであり,ヒト型MMRが生物界で圧倒的に広く採用されていると考えられるようになった.以下では,特に述べない限りエンドヌクレアーゼ活性を持つヒト型のMutLを単にMutLと呼ぶ.
制限酵素MutHと異なり,MutLは,単独では何ら基質特異性を示さない.精製したMutLを試験管内でDNAと反応させるとDNAを細切れにしてしまう.この活性をミスマッチ特異的なものとするために,細胞内では厳密な制御機構が存在すると考えられた.我々は,そのDNA切断活性の制御機構を明らかにするために,安定性が高く生化学的解析に適した高度好熱菌や超好熱菌由来のMutLを用いた.以下に示す研究は,高度好熱菌丸ごと一匹プロジェクト8)の一環として行われた.
MutLはGHKL ATPアーゼスーパーファミリーに属する.このファミリーに属するタンパク質には,Hsp90,細菌由来ヒスチジンキナーゼ,大腸菌MutLなどがあり,これらはすべて,ATP結合と加水分解のサイクルに依存して大きな構造機能変化を生じる.大腸菌MutLではATP結合に伴ってN末端ドメインが二量体化すること,DNAへの親和性が向上することが報告されていたが,ヒト型のMutLに関しては,ATPの効果は未知であった.そこで,ATP存在下でのMutLのDNA結合能,DNA切断活性を調べたところ,ATPによってMutLのDNA結合能およびDNA切断活性が抑制されることを見いだした.2 mMのATPはMutLのDNA切断活性の速度を最大で10倍以上低下させた.ATPをATPの非加水分解アナログに代えた場合にも同様の結果が得られたが,アデノシン5′-二リン酸は何の効果も示さなかった.以上の結果から,ATP結合によってMutLのヌクレアーゼ活性が抑制されることがわかった.MutLのATPに対する解離定数は約20 μMであることから,細胞内で遊離のMutLはATPを結合し,非特異的なDNA切断が起こらないように不活性化された状態にあると考えられる7).
すでに述べたとおり,MutLはN末端のATP加水分解ドメインとC末端のヌクレアーゼドメインからなる.DNA結合は主にN末端ドメインが担っており,C末端ドメインは補助的に働く.興味深いことに,ATPによるDNA結合能の抑制は,N末端ドメイン単独では観察されず,MutL全長においてのみ観察された9).ATP結合に伴う機能変化には,N末端ドメインとC末端ドメインの協調的な働きが必要であるといえる.
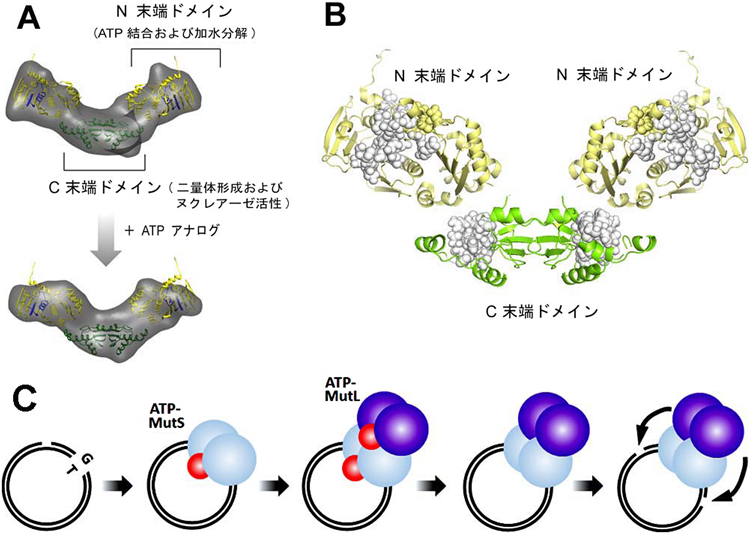
それではATPが結合した際,MutLにはどのような構造変化が起きているのだろうか.大腸菌MutLも含め,これまで知られているGHKLファミリータンパク質では,例外なくATP結合にはN末端ドメインの二量体化を伴うことがわかっている.ATP結合による構造変化を調べるために,X線小角散乱を用いてMutLの構造解析を行った.一般に,単離されたMutLは凝集する傾向が強く,これまでに全長構造が信頼性高く解析されたことはなかった.我々は,非常に安定で溶解度も高い超好熱菌由来MutLを利用することで全長タンパク質の構造解析に成功した.驚いたことに,ATP結合時にもN末端ドメインは二量体化しておらず,大腸菌MutLでみられたような大きな構造変化は起こっていないことがわかった(図2A).しかしながら,ATP結合に伴ってN末端ドメインだけでなく,C末端ドメインにおいても形状に明らかな変化が認められた.
次に,ATP結合時の構造変化を重水素交換質量分析法により調べた.この手法は,微細な構造変化に由来するポリペプチド主鎖の重水素交換速度の変化を質量分析計によって測定するもので,NMRによる測定と異なり巨大な分子の全体あるいはタンパク質複合体なども対象にできる.その結果,ATP結合に伴って,ヌクレアーゼ活性モチーフ周辺に明確な重水交換速度の低下が検出された10).つまり,N末端ドメインへのATP結合によってC末端ドメインのヌクレアーゼ活性部位の自由度が制限され,その結果,ヌクレアーゼ活性が低下することが示唆された(図2B).さらに,ATP依存的な重水交換速度の低下は,C末端ドメインの,それまで注目されていなかった領域にも及んでいた.この領域には細菌からヒトMutLの間で完全に保存されたアルギニン残基が存在する(ヒトMutLのサブユニットhMLH1ではArg-755にあたる)が,これを部位特異的変異導入によってアラニンに置換するとヌクレアーゼ活性が消失した.
このように,重水素交換速度の測定によって,これまで知られていなかった触媒に必須の残基を同定することに成功した.さらに重要なことに,hMLH1のArg-755には,リンチ症候群の家系で報告されているミスセンス変異が存在した.近年,シークエンス技術の向上によって,がん細胞・組織における遺伝子変異のデータが蓄積されているが,それらが有害な変異であるか,中立の変異なのかを判断する実験的証拠の不足が問題となっている.hMLH1 Arg-755に関しては,ミスセンス変異によってヒトMutLのヌクレアーゼ活性が消失し,MMRが機能不全に陥ることでリンチ症候群の発症につながることが強く示された.
これまでに,非特異的切断を防ぐためにATP結合によってMutLが不活性化される機構を述べてきた.では,MutLにかけられた安全装置を解除する因子は何か.プラスミドを基質とし,精製タンパク質による部分再構成系を利用することで,ATP結合型MutLを活性化する条件を探索した.その結果,基質プラスミドにミスマッチが含まれる場合に,MutSとATP依存的にMutLのエンドヌクレアーゼ活性が促進されることを見いだした11).さらに,MutSとMutLのATP加水分解活性欠失変異体を用いた実験から,MutLの効率的な活性化には,MutSとMutLの両者によるATP加水分解が必要であることがわかった.面白いことに,基質プラスミドからミスマッチをなくし,別の分子である直鎖DNAにミスマッチを持たせて混合した場合にはプラスミドの切断は起こらなかった.つまり,ミスマッチ,MutS,MutLの三者が同じDNA鎖上に存在することがMutL活性化の条件であるといえる.同一DNA上においてMutS–MutL複合体が形成されることでMutSとMutLのATP加水分解が起こり,MutLの不活性化が解除されると考えられる(図2C).
これまで述べたとおり,MutLのヌクレアーゼ活性に「ミスマッチ特異性」を持たせる分子機構を試験管の中で再構成することに成功した.しかし,「新生鎖特異性」を付与する機構については我々が用いた好熱菌由来の系では再構成できていない.ニックを導入したミスマッチプラスミドを基質とし,PCNAのカウンターパートであるβ-クランプをクランプローダーとともに混合し,β-クランプがDNAにロードされていることを確認の上,MutLによるDNA切断活性の鎖特異性を調べたが,好熱菌由来MutLは両方の鎖を切断していた11).好熱菌におけるMMRではクランプがシグナルを伝達するためにさらに別の因子が必要とされる可能性もある.あるいは,MutLが利用する新旧鎖識別シグナルが鎖の不連続性ではないのかもしれない.多くの生物ではMutLのC末端ドメインにPCNAあるいはβ-クランプ相互作用モチーフが存在するが,好熱菌由来MutLにはそのモチーフが見当たらず(表1)12),このことも好熱菌MMRの特殊性を示唆している.
この10年で,MutHを持たない生物におけるMMRの理解は急速に進んだ.特に真核生物ではMutLエンドヌクレアーゼがPCNA依存的に新生鎖特異的な切断活性を示すことがわかり,修復機構の全容がみえてきた.しかしながら,新生鎖のシグナルとなる鎖の不連続性とMutLの切断部位は遠く離れており,PCNAがどのようにMutLにシグナルを伝達するのかを明らかにする必要がある.また,MutHを持たない細菌においては,娘鎖の不連続性が新旧鎖識別シグナルとして働くことを示す直接的な証拠がまだなく,試験管内再構成での鎖特異性の再現が待たれる.古細菌では,mutSやmutLホモログと予想される遺伝子を持つものも多いが,大腸菌型あるいはヒト型のMMRが機能していることは証明されていない.古細菌のDNAポリメラーゼが高い正確性を示すせいでMMRが不要になったのではないか,という指摘もある.しかし,MMRの存在意義はゲノムの安定性維持に限らない.たとえば細菌では,MMRが複製の忠実度に大きく貢献しているために,MMRの活性を調節することでゲノムの改変速度を容易に操ることができ,環境変化への迅速な適応を可能にしている.古細菌におけるMMRについては慎重に検証される必要がある.
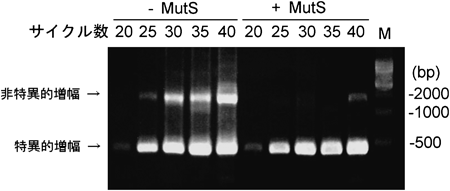
MMRに関する研究は,基本的な生命現象の理解と病態の解明に貢献するだけでなく,MMRタンパク質の工業的な応用にもつながる可能性がある.たとえば,MMRを試験管内で機能させることでPCRにおける変異を抑制するというアイデアが古くから存在する.しかしながら,試験管内でのDNA複製ではMMRに認識される新旧鎖識別シグナルを持たせることができないため,複製ミスを修復することは困難であると予想される.そこで我々は,複製ミスによって生じるミスマッチ塩基に好熱性細菌由来の耐熱性MutSを結合させ,次サイクルでの複製を阻害する手法を考案した.この手法により,PCR由来の変異が3分の1程度に抑えられることがわかった.さらに,予期していなかったことであるが,好熱菌由来MutSは変異抑制効果だけでなく非特異的増幅抑制効果も示した(図3).これは,プライマーが鋳型鎖と非特異的に対合した際に生じるミスマッチ塩基対に対してMutSが結合し,DNAポリメラーゼの接近を阻害するためであると考えられた13).MMRタンパク質の機能解明が進むことでこれらの技術がさらに発展し,PCRに基づく診断技術や研究手法の精度改善に貢献すると期待される.
謝辞Acknowledgments
本研究は,大阪大学倉光成紀教授の指導の下,理化学研究所SPring-8センターにおいて行いました.理化学研究所飯野均博士(現河合塾),大阪大学島田敦広博士(現兵庫県立大学),増井良治准教授(現大阪市立大学教授),高橋達郎助教,慶應大学山本竜也助教の多大な貢献に感謝いたします.
引用文献References
1) Kunkel, T.A. & Erie, D.A. (2005) Annu. Rev. Biochem., 74, 681–710.
2) Lynch, H.T., Kimberling, W., Albano, W.A., Lynch, J.F., Biscone, K., Schuelke, G.S., Sandberg, A.A., Lipkin, M., Deschner, E.E., Mikol, Y.B., Elston, R.C., Bailey-Wilson, J.E., & Danes, B.S. (1985) Cancer, 56, 934–938.
3) Fukui, K. (2010) J. Nucleic Acids, 2010, 260512.
4) Modrich, P. (2006) J. Biol. Chem., 281, 30305–30309.
5) Kadyrov, F.A., Dzantiev, L., Constantin, N., & Modrich, P. (2006) Cell, 126, 297–308.
6) Pluciennik, A., Dzantiev, L., Iyer, R.R., Constantin, N., Kadyrov, F.A., & Modrich, P. (2010) Proc. Natl. Acad. Sci. USA, 107, 16066–16071.
7) Fukui, K., Nishida, M., Nakagawa, N., Masui, R., & Kuramitsu, S. (2008) J. Biol. Chem., 283, 12136–12145.
8) Yokoyama, S., Hirota, H., Kigawa, T., Yabuki, T., Shirouzu, M., Terada, T., Ito, Y., Matsuo, Y., Kuroda, Y., Nishimura, Y., Kyogoku, Y., Miki, K., Masui, R., & Kuramitsu, S. (2000) Nat. Struct. Biol., 7 (Suppl), 943–945.
9) Iino, H., Kim, K., Shimada, A., Masui, R., Kuramitsu, S., & Fukui, K. (2011) Biosci. Rep., 31, 309–322.
10) Yamamoto, T., Iino, H., Kim, K., Kuramitsu, S., & Fukui, K. (2011) J. Biol. Chem., 286, 42337–42348.
11) Shimada, A., Kawasoe, Y., Hata, Y., Takahashi, T.S., Masui, R., Kuramitsu, S., & Fukui, K. (2013) FEBS J., 280, 3467–3479.
12) Fukui, K., Shimada, A., Iino, H., Masui, R., & Kuramitsu, S.(2011) in DNA Repair - on the Pathway to Fixing DNA Damage and Errors (Storici, F. eds.), pp. 123–142, Intech, Rijeka.
13) Fukui, K., Bessho, Y., Shimada, A., Yokoyama, S., & Kuramitsu, S. (2013) Int. J. Mol. Sci., 14, 6436–6453.
14) Shimada, A., Masui, R., Nakagawa, N., Takahata, Y., Kim, K., Kuramitsu, S., & Fukui, K. (2010) Nucleic Acids Res., 38, 5692–5705.
著者紹介Author Profile
 福井 健二(ふくい けんじ)
福井 健二(ふくい けんじ)大阪医科大学医学部生化学教室助教.理学博士.
略歴1977年奈良県に生る.2002年大阪大学理学部卒業.07年同大学院理学研究科博士課程修了.07年理化学研究所播磨研究所研究員.12年大阪大学産学連携本部特任助教.13年株式会社トランスジェニック研究員.14年より現職.
研究テーマと抱負タンパク質の構造機能解析とその応用に関する研究.これまでに,「ミスマッチ」をキーワードに様々なDNA結合タンパク質の機能を解明してきた.今後,治療や診断など医療に役立つ可能性のある基礎研究を推進していきたい.
ウェブサイトhttp://www.osaka-med.ac.jp/deps/med/
趣味バスケットボール,昆虫の観察.