生物種の全遺伝情報をゲノムと呼ぶように,脳の神経回路接続の総体のことをコネクトームと呼ぶ.この言葉ができたのは比較的最近のことであるが,近年,コネクトームを「解読」することで脳を理解しようという機運が高まっている.哺乳類のコネクトーム研究に関しては,現在三つの異なるスケールにおいて,それぞれ異なるアプローチが提案されている.マクロスケールのコネクトーム解析では,水分子の拡散異方性を測定することで神経線維を可視化する拡散テンソル画像(diffusion tensor imaging: DTI)計測など,主にMRIが用いられており,ヒトを含む動物における神経束のトレーシングに威力を発揮している.現時点での分解能は1 mm程度である.メゾスケールのコネクトーム解析では光学顕微鏡(特に蛍光顕微鏡)を用いた解析が主流である.特に遺伝学やウイルスベクター,蛍光タンパク質を組み合わせることで,特定の回路だけを標識,追跡するのに大変優れている.しかしながら,光学顕微鏡は分解能の限界が200 nm程度とされており,厳密にシナプスの同定を行うことはできないという欠点がある.ミクロスケール,すなわちシナプスレベルでのコネクトーム解析を行うには分解能に優れた電子顕微鏡を用いたアプローチが必須であり,すでに電子顕微鏡を用いたコネクトーム解析のパイプラインが提案されている.しかしながら,大容量のデータ取得や解析における困難から,大きなサンプルの解析にはまだ課題が多い.
従来,電子顕微鏡および光学顕微鏡においては,3次元構造を解析するには,連続切片の撮影と再構築が必須であった.しかしながら,切片作製時における歪みや位置合わせの困難から,この作業には大変な労力を要する.近年,こうした問題を解決する方法として,サンプル表面を少しずつ削りながら連続ブロック表面をイメージングするタイプの電子顕微鏡(serial block-face scanning electron microscopy: SBF-SEMなど)および光学顕微鏡(serial two-photon tomographyなど)が開発され,すでに市販もされている.たとえば,アレン脳科学研究所のグループは,マウス脳に緑色蛍光タンパク質(GFP)を発現するアデノ随伴ウイルス(AAV)ベクターを導入し,これを市販のserial two-photon tomographyシステムで全脳イメージングすることでメゾスケールのコネクトームを解析している.
しかしながら,最近になってさまざまな組織透明化の手法が開発され,「脳は切って解析する」という常識は変わりつつある.また,組織透明化によって,光学顕微鏡がマクロスケールからミクロスケールまでをカバーできる可能性が出てきている.本稿では組織透明化法の開発に関する現状と今後の展望を述べるとともに,実際にこれらの方法を取り入れるにあたって注意すべき点についても述べたい.また,「次々に新しい方法が出てくるけど,結局どの方法がよいの?」というよくある質問にもお答えすることにしたい.
組織透明化は,現在コネクトーム研究への応用に対する期待から注目されており,過大な宣伝も時折目にするが,決して新しい方法ではない.生体組織を透明化する試みは100年以上も前からなされている.多くは芳香族有機溶媒を用いたもので,サリチル酸メチルやBABB(ベンジルアルコールおよび安息香酸ベンジルの混合物)などがよく知られている.このほか,ペプチド結合を適度に化学切断したのちグリセリン置換を行う透明化法は古くから透明骨格標本の作製に用いられている.
組織透明化の原理に関しては,サンプルと溶媒の屈折率が鍵であるということがすでによく知られている.通常,生体組織は屈折率1.33の水に浸されているが,生体組織は線維状タンパク質や脂質などにより屈折率が1.5程度の構造を有する.このため,組織の構造物と溶媒の間には屈折率の差が生じ,反射,屈折,光散乱の原因となりうる.特に光散乱については,近年着目されている補償光学系など光学系の工夫を施しても取り除くことは困難である.したがって,生体組織を透明にするには,組織から高屈折率成分を取り除いたり,溶媒を高屈折率液体に置き換えたり,この両者を組み合わせることで,組織中の屈折率を均一にすることが必要となる.
従来の組織透明化法は適当な染色法と組み合わせることが前提であったが,これらの方法をそのまま現在の生命科学研究に用いようとするとさまざまな制約があった.そこで,最新の蛍光顕微鏡技術や蛍光タンパク質の利用に最適な組織透明化法の開発が注目されているというのが現状である.
BABB法は古くから共焦点顕微鏡観察に用いられてきたが,最近になって光シート型蛍光顕微鏡の一種,ultramicroscopyが開発されたことで着目された1).この方法ではGFPなどの蛍光タンパク質が短時間で褪色してしまうという問題はあるものの,簡便に良好な透明度を得ることができ,マウスの脳を数mmの深さまでイメージングすることができる.その後,BABB法の改良型として報告された3DISCO法では,脳における光散乱の要因である脂質をテトラヒドロフラン(tetrahydrofuran: THF)で効果的に除くとともに,高屈折率液体にジベンジルエーテル(dibenzylether: DBE)を用いることで蛍光タンパク質の褪色を軽減している2).3DISCO法によってマウス全脳の蛍光イメージングが可能となったが,蛍光タンパク質が著しく減弱してしまうという問題は依然として残されている.BABB法やiDISCO法は蛍光タンパク質の褪色以外にも,脱水・透明化プロセスにおけるサンプルの収縮が問題点としてあげられる.最近になって,より蛍光タンパク質の蛍光を保持するため,組織をレジンに包埋した上で3DISCO法によって透明化する方法も提案されている3).
BABB法や3DISCO法は蛍光タンパク質の蛍光を保持するという観点からは難があるものの,透明度は大変優れており,抗体染色と組み合わせて深部観察することは十分可能である.最近,凍結融解,メタノール処理,プロテアーゼ処理などを施すことで,マウス全脳を抗体染色できると報告されている4).メタノール処理を用いた方法は別のグループからもiDISCO法として報告されている5).
2,2′-チオジエタノール(2,2′-thiodiethanol: TDE)はもともと高屈折率のマウント剤として報告された溶媒であるが,組織透明化試薬としても優れている6).TDEは水溶性のため,透明化に際して脱水プロセスを必要とせず,組織の収縮はそれほど顕著ではない.ただし,TDEも蛍光タンパク質を褪色させてしまうため,抗体染色などと組み合わせることが必要である.
ホルムアミドを主成分とするClearTもサンプルの伸縮をあまり伴わずに透明化が可能であるが,やはり蛍光タンパク質の褪色が問題である.褪色を抑えたClearT2も提案されている7).
有機溶媒を用いた組織透明化法では蛍光タンパク質の褪色が大きな問題であったが,これは蛍光タンパク質の発色団が蛍光を発するのに水分子が必要なためである.Scale法は水溶液ベースであり,蛍光タンパク質を褪色させることなく組織透明化を達成した初めての方法である8).尿素とグリセリン,界面活性剤を組み合わせることで光散乱を減らし,透明度を上げている.2光子励起顕微鏡と組み合わせることで,脳表面から深さ4 mmまで蛍光イメージングを行うことに成功している.難点としては,透明化に時間がかかること,組織が膨潤してもろくなることがあげられる.
CUBIC法は,Scale法で用いている三つの試薬を再検討することで開発された,改良版Scale法ともいうべき方法である9).CUBIC法ではScale法で用いていたグリセリンをアミノアルコールに代えることでさらに透明度を上げることに成功している.これにより,蛍光タンパク質をある程度保持したまま,全脳規模のultramicroscopyが可能となっている.また,一部の組織ではヘムの光吸収も透明度を損なう原因となることが知られているが,CUBIC法ではヘムを溶出できるため,血液を多く含む臓器の深部イメージングにも有効である10).
6. CLARITY, Advanced CLARITY, PACT
脳組織の透明化において最大の困難はミエリン鞘などに多く含まれている脂質の除去である.3DISCO法ではTHFによって除き,CUBIC法では高濃度のTriton-X100で除去しているが,CLARITY法ではタンパク質をポリアクリルアミドゲル担体に架橋した上で,SDSを含むバッファー中で電流を流して脂質を除去するという方法をとっている11).最初に発表されたCLARITY法ではパラメータの設定が難しく,再現性に乏しいというのがもっぱらの評判であったが,その後発表された改良法(Advanced CLARITY, PACT)ではSDSを含むバッファー中でサンプルを撹拌するという簡便な方法も提案されている12,13).しかしながら,マウス全脳のような大きなサンプルでは透明化に数週間を要し,迅速にというわけにはいかない.
CLARITY法は細胞膜成分を積極的に除去しているため,抗体の浸透性に優れているというのが利点である.また,優れた透明度を活かして全脳蛍光イメージングするための光シート型顕微鏡の作製も試みられている(CLARITY-optimized light-sheet microscopy: COLM)12).
これまでに報告されている組織透明化法は非常に時間がかかったり,操作が煩雑であったりするという問題点があった.また,透明化の過程で,3DISCOではサンプルの収縮,CUBICやCLARITYではサンプルの一時的な膨潤を伴うため,この過程で微細な組織形態が損なわれるといった問題点があった.
筆者らが開発したSeeDB法は,組織を3日間かけて高濃度のフルクトース溶液に浸けていくという簡便な操作で,組織の微細な形態にも影響を与えることなく透明化することができる14).溶媒を脂質などの屈折率に近づけることで透明化を達成しているため,脂質を除去する必要がなく,細胞膜さえも保持している.透明度ではCLARITYやCUBICには及ばないため,光シート型顕微鏡を用いたイメージングには向かないものの,簡便で再現性が高く,微細な構造の定量解析に優れている.共焦点顕微鏡や二光子励起顕微鏡との相性がよいため,一般的な研究者には使い勝手がよい方法といえよう.プロトコルの詳細やトラブルシューティングに関しては,筆者が管理するウェブサイトSeeDB Resourcesを参照されたい15).
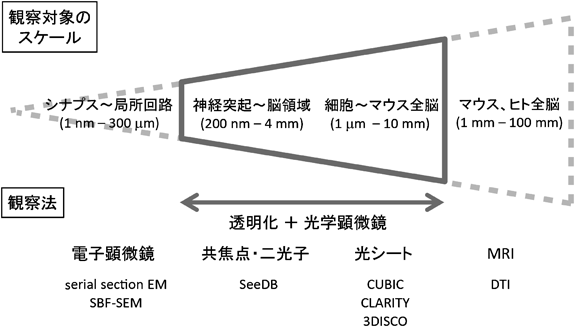
組織透明化を用いて3次元蛍光イメージングを行うにあたっては,観察の対象に応じて最適な透明化法を選択することが重要である(図1,表1).万能の方法というのは存在しない.とりあえず全脳にわたって微細な構造まで見る,というのは困難であるばかりか時間とエフォートの無駄である.研究の目的が明確であれば見るべきポイント,すなわち用途も明確であろう.
表1 各種透明化法の比較 | 3DISCO2),iDISCO5) | TDE6) | CUBIC9, 10) | Advanced CLARITY12) | SeeDB14) |
|---|
| 透明度 | 非常に高い | 高い | 非常に高い | 非常に高い | 高い |
| 組織の伸縮 | かなり縮む | 若干縮む | 一時的にかなり膨張 | 一時的にかなり膨張 | 伸縮しない |
| 蛍光タンパク質の安定性 | かなり褪色 | かなり褪色 | やや褪色 | やや褪色 | 数日間は安定 |
| 自家蛍光 | 青~緑は自家蛍光が強い | 出にくい | 出にくい | 出やすい | 1週間以上経つと出やすい |
| 深部抗体染色 | 優れている | 可能 | 優れている | 優れている | 可能 |
| 操作時間 | 2~3日 | 2~3日 | 3~10日 | 1~数週間 | 2~3日 |
| 操作の簡便さ | やや煩雑 | 簡便 | 簡便 | 非常に煩雑 | 簡便 |
| 類似技術がある場合には最新のバージョンもしくは最も使い勝手の良い方法を記載している.筆者の研究室における検討結果に基づく(iDISCOを除く).操作時間は脳スライスを用いるか全脳を用いるかによっても変わる. |
全脳のイメージングが必須な実験としては,たとえばあるトランスジーンの発現領域を全脳にわたって確認したいとか,神経活動に伴って発現するレポーターの局在を全脳でマッピングしたいとか,脳の領域対領域の結合関係を見たい,といったものが考えられる.こうした場合にはCUBIC法が最も簡便で再現性に優れる.CUBIC法で透明化すると脆弱になるサンプルや透明度が不十分なサンプルに関しては,アクリルアミドゲルに架橋するCLARITY法も有効であると考えられる(ただし,CLARITY法もサンプルの膨潤は避けられない).また,免疫染色が必須な実験ではメタノール処理と3DISCOの組合わせも比較的簡便で検討に値する.これらの方法を用いて全脳イメージングを行う場合には,使用する顕微鏡についてもよく検討する必要がある.というのも,通常の共焦点顕微鏡のようなポイントスキャン型の顕微鏡を用いる場合,全脳にわたってイメージングしようとすると膨大な時間がかかり,高解像度のイメージングは事実上不可能だからである(図1).ultramicroscopyや光シート型顕微鏡を使えばより高速にイメージングすることが可能であるが,市販の顕微鏡では屈折率の高い透明化サンプルの観察時に球面収差が生じるため,実は十分な分解能は得られない.また,そもそもCUBIC法やCLARITY法,3DISCO法では透明化ステップでサンプルの伸縮を伴うため,必ずしも微細な構造が保持されておらず,微細な構造を観察する意味は小さい.これらのことから,現時点ではCUBIC法やCLARITY法は細胞レベルや軸索束レベルの分解能(数µm)でイメージングする方法と割り切って使うべきであろう.
もう少し細かく(0.5~数µm),一つ一つの軸索や樹状突起の走行まで定量的に解析したいという場合には,透明化の過程で形態変化を伴わないSeeDBが最もよいであろう.顕微鏡に関しては光シート型顕微鏡よりもむしろポイントスキャン型の共焦点顕微鏡や二光子励起顕微鏡の方がよい.光シート型顕微鏡は励起光がカメラに捉えられるまでの間に光散乱で像が劣化するのに対して,ポイントスキャンでは原理的にそのような影響を受けにくいためである.また,SeeDBなどの透明化液の屈折率に対応した対物レンズがすでに市販されていることから,これらを使えば深部でも球面収差の影響なく,高分解能の画像を得ることができる.とはいえ,深さ3 mm,4 mmを超えると単一の軸索を追跡することは難しい.適切な向きの脳スライスを作製する,神経細胞をまばらに標識する,といった工夫を組み合わせる必要がある.
さらに局所的に細かい構造(0.2~1 µm),たとえば樹状突起のスパインの定量解析を行いたいという場合には二光子励起顕微鏡ではなく,一光子励起の共焦点顕微鏡を使うのが有利である.顕微鏡における分解能は光の波長とレンズの開口数(numerical appature: NA)によって決まり,光の波長が短いほど,またNAが大きいほど分解能がよい.共焦点顕微鏡を使うと,励起波長が二光子励起顕微鏡の半分になるため,分解能も半分になる.NAを大きくするためには原理的に作動距離を小さくしなければならないため,分解能を向上させるためには深さを犠牲にせざるをえない.したがって,細かい構造を観察したければ,脳をスライスにしてSeeDBで透明化し,NAの大きな油浸レンズを使って共焦点顕微鏡で観察するのがよい.将来的には超解像顕微鏡を組み合わせてさらに細かい構造を可視化することも可能になるであろう(筆者ら,未発表データ).しかしながら,いずれの場合も,作動距離の制約と撮影に要する時間の両方の理由から,全脳の高分解能イメージングは現実的ではない.
本稿では神経科学への応用を前提として解説したが,神経科学においてのみならず,現代生物学のあらゆる分野で,生き物の構造を3次元的かつ定量的に捉えることは必須になりつつある.したがって,組織透明化法は今後もさまざまな改良法や応用が出てくるものと期待される.今後の開発の方向性としては,1)より簡便により透明に,2)さらに深く,3)さらに高分解能で,最終的には4)in vivoで見る,といったものがあげられるだろう.こうした新しい技術によって3次元的な画像情報の取得が容易となり,新しい生物学が発展することを期待したい.
謝辞Acknowledgments
本研究は科学研究費補助金若手研究費(A),JSTさきがけ,三菱財団,住友財団,中島記念国際交流財団,理化学研究所の助成を受けて行われた.共同研究者の柯孟岑博士および研究室メンバーに感謝いたします.
引用文献References
1) Dodt, H.U., Leischner, U., Schierloh, A., Jahrling, N., Mauch, C.P., Deininger, K., Deussing, J.M., Eder, M., Zieglgansberger, W., & Becker, K. (2007) Nat. Methods, 4, 331–336.
2) Erturk, A., Becker, K., Jahrling, N., Mauch, C.P., Hojer, C.D., Egen, J.G., Hellal, F., Bradke, F., Sheng, M., & Dodt, H.U. (2012) Nat. Protoc., 7, 1983–1995.
3) Becker, K., Hahn, C.M., Saghafi, S., Jahrling, N., Wanis, M., & Dodt, H.U. (2014) PLoS ONE, 9, e114149.
4) Gleave, J.A., Lerch, J.P., Henkelman, R.M., & Nieman, B.J. (2013) PLoS ONE, 8, e72039.
5) Renier, N., Wu, Z., Simon, D.J., Yang, J., Ariel, P., & Tessier-Lavigne, M. (2014) Cell, 159, 896–910.
6) Gonzalez-Bellido, P.T. & Wardill, T.J.(2012) Cold Spring Harb. Protoc., http://dx.doi.org/doi:10.1101/pdb.prot069625.
7) Kuwajima, T., Sitko, A.A., Bhansali, P., Jurgens, C., Guido, W., & Mason, C. (2013) Development, 140, 1364–1368.
8) Hama, H., Kurokawa, H., Kawano, H., Ando, R., Shimogori, T., Noda, H., Fukami, K., Sakaue-Sawano, A., & Miyawaki, A. (2011) Nat. Neurosci., 14, 1481–1488.
9) Susaki, E.A., Tainaka, K., Perrin, D., Kishino, F., Tawara, T., Watanabe, T.M., Yokoyama, C., Onoe, H., Eguchi, M., Yamaguchi, S., Abe, T., Kiyonari, H., Shimizu, Y., Miyawaki, A., Yokota, H., & Ueda, H.R. (2014) Cell, 157, 726–739.
10) Tainaka, K., Kubota, S.I., Suyama, T.Q., Susaki, E.A., Perrin, D., Ukai-Tadenuma, M., Ukai, H., & Ueda, H.R. (2014) Cell, 159, 911–924.
11) Chung, K., Wallace, J., Kim, S.Y., Kalyanasundaram, S., Andalman, A.S., Davidson, T.J., Mirzabekov, J.J., Zalocusky, K.A., Mattis, J., Denisin, A.K., Pak, S., Bernstein, H., Ramakrishnan, C., Grosenick, L., Gradinaru, V., & Deisseroth, K. (2013) Nature, 497, 332–337.
12) Tomer, R., Ye, L., Hsueh, B., & Deisseroth, K. (2014) Nat. Protoc., 9, 1682–1697.
13) Yang, B., Treweek, J.B., Kulkarni, R.P., Deverman, B.E., Chen, C.K., Lubeck, E., Shah, S., Cai, L., & Gradinaru, V. (2014) Cell, 158, 945–958.
14) Ke, M.T., Fujimoto, S., & Imai, T. (2013) Nat. Neurosci., 16, 1154–1161.
15) SeeDB Resources, https://sites.google.com/site/seedbresources/
著者紹介Author Profile
 今井 猛(いまい たけし)
今井 猛(いまい たけし)理化学研究所多細胞システム形成研究センター(理研CDB)チームリーダー.理学博士(東京大学).
略歴1978年東京都生まれ,長野県出身.2006年東京大学大学院理学系研究科生物化学専攻修了.同博士研究員を経て,10年より現職.京都大学大学院生命科学研究科客員准教授.
研究テーマと抱負大学院時代から一貫して,哺乳類脳の神経回路形成のロジックを研究.独立後は,生理学のアプローチも取り入れながら,ダイナミックな神経活動パターンや機能的な神経回路が生後発達の過程でどのようにして作られるのかについて研究している.
ウェブサイトhttp://imai.uijin.com/
趣味育児,実験.