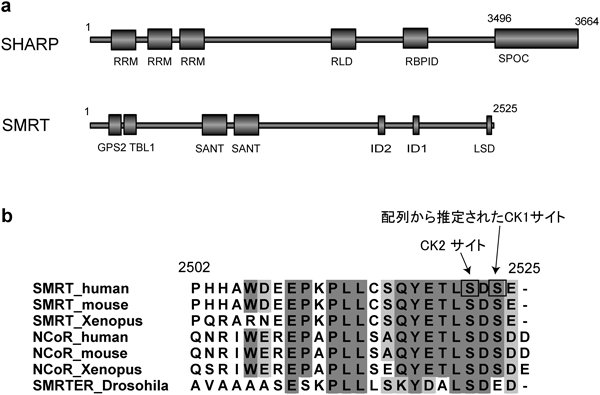
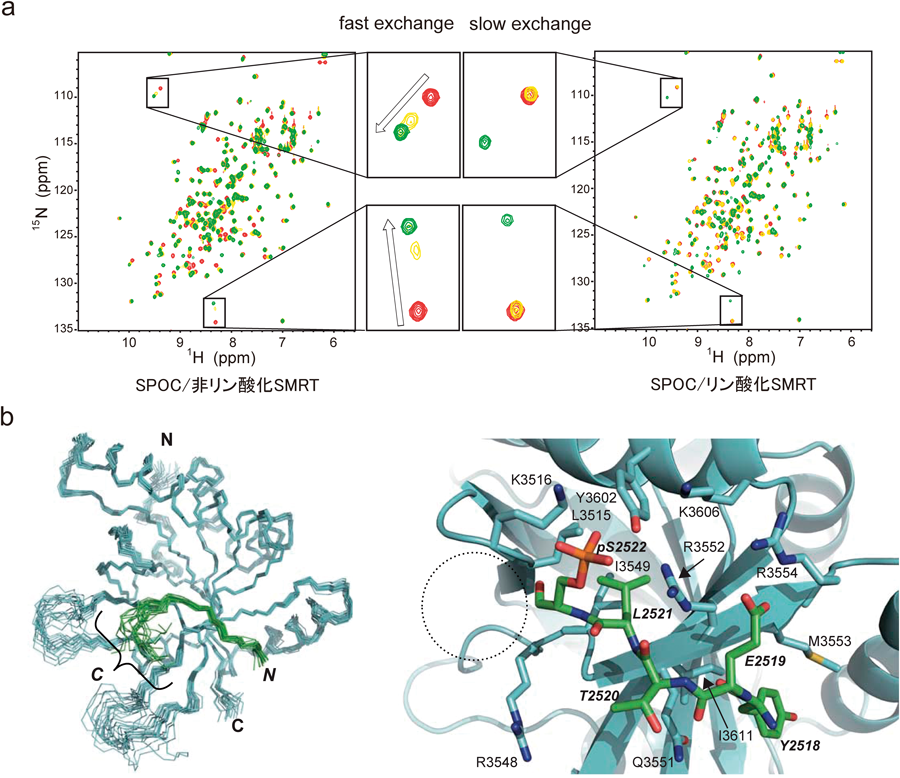
転写抑制補因子の複合体形成におけるCK2によるリン酸化の分子スイッチとしての役割Phosphorylation of the transcriptional co-repressor complex by CK2 as a molecular switch
首都大学東京大学院理工学研究科Graduate School of Science and Engineering, Tokyo Metropolitan University ◇ 〒192-0397 東京都八王子市南大沢1-11-1 Minami-Osawa, Hachioji-shi, Tokyo 192-0397, Japan