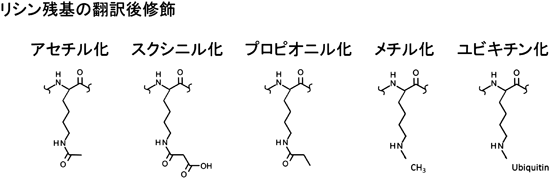
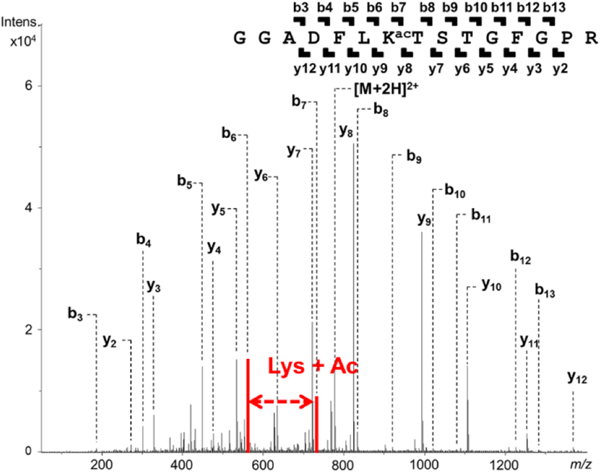
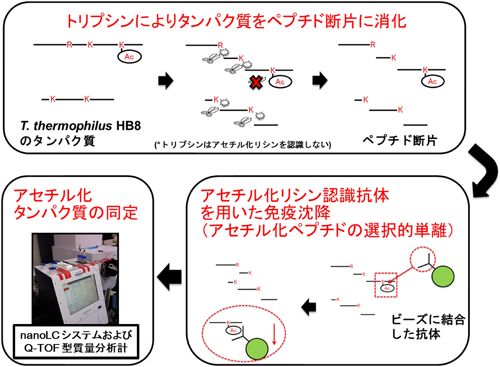
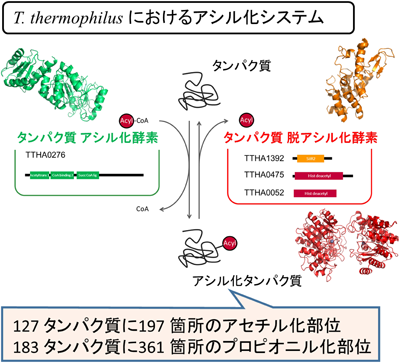
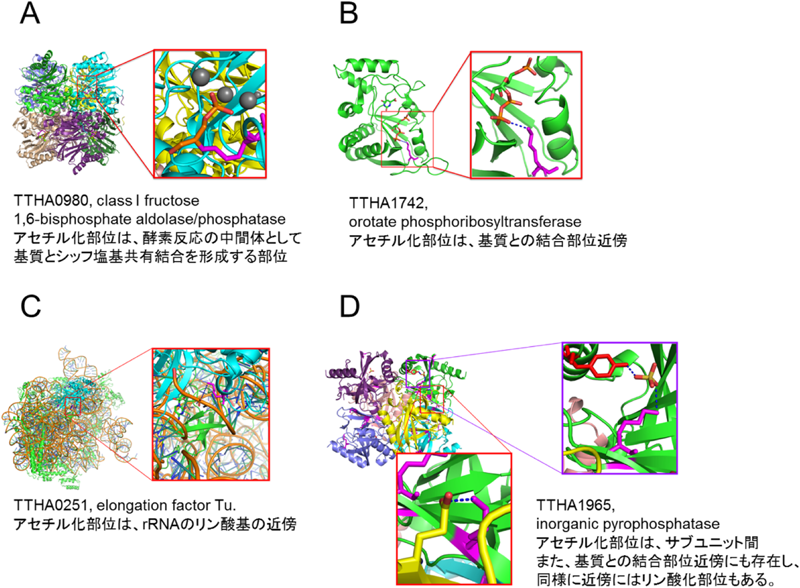
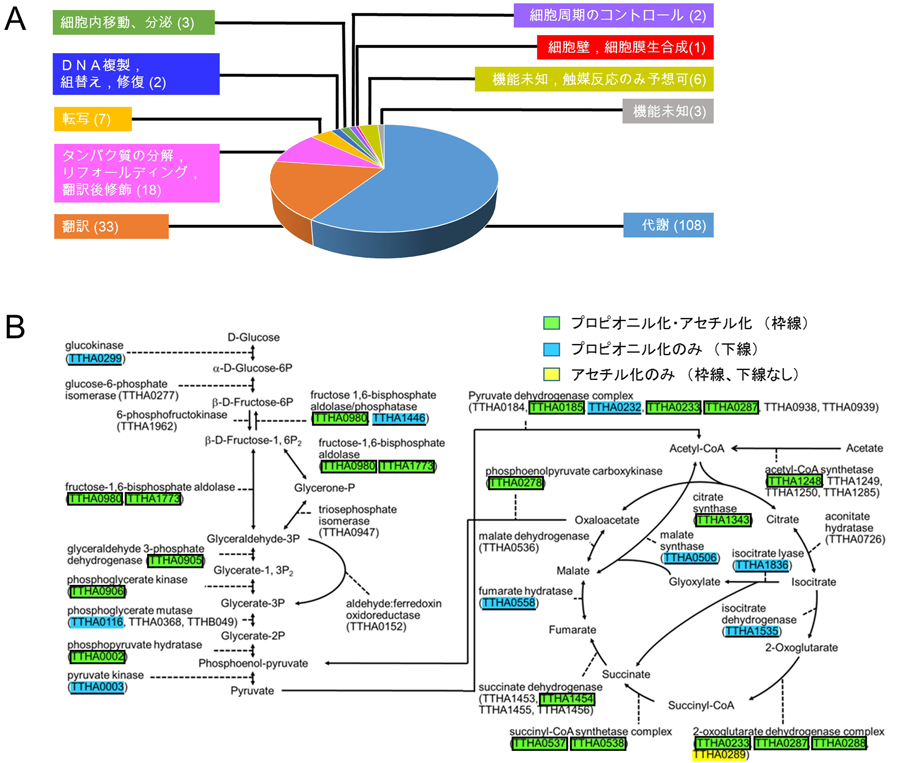
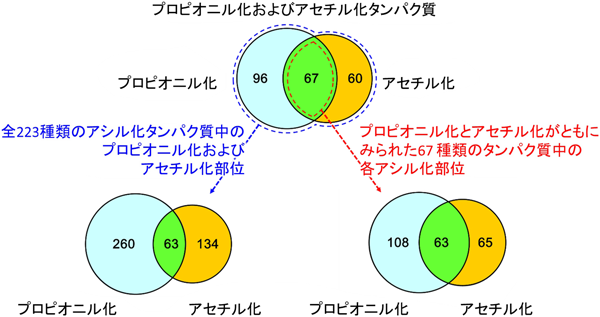
翻訳後修飾による酵素の多機能性を探る―質量分析技術Functional discovery of functionally unknown proteins by mass spectrometry
大阪大学大学院理学研究科生物科学専攻Department of Biological Sciences, Graduate School of Science, Osaka University ◇ 〒560-0043 大阪府豊中市待兼山町1番1号1-1 Machikaneyama-cho, Toyonaka-shi, Osaka 560-0043, Japan