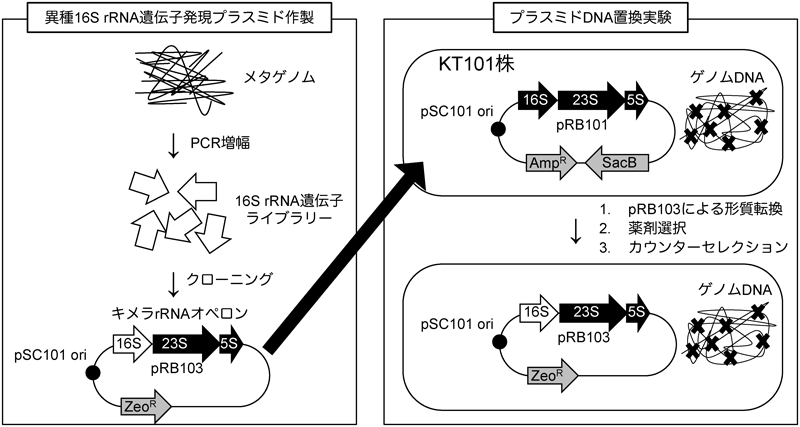
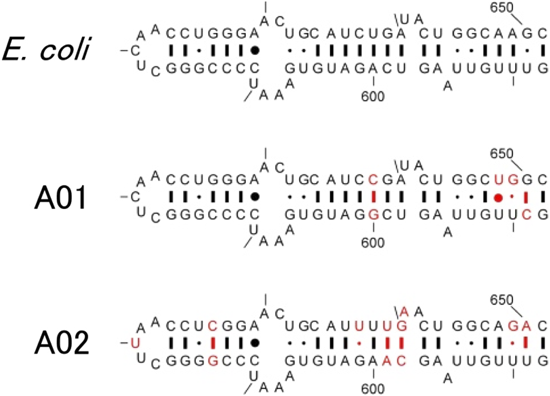
16SリボソームRNAの水平伝播実験からみえてくるリボソームの可塑性Functional plasticity of bacterial ribosome: Experimental horizontal gene transfer of 16S rRNA genes in Escherichia coli
1 国立研究開発法人産業技術総合研究所生命工学領域生物プロセス研究部門合成生物工学研究グループSynthetic Bioengneering Research Group, Bioproduction Research Institute, Department of Life Science and Biotechnology, National Institute of Advanced Industrial Science and Technology (AIST), Japan ◇ 〒305-8566 茨城県つくば市東一丁目1番1号1-1-1 Higashi, Tsukuba-shi, Ibaraki 305-8566, Japan
2 東京大学大学院新領域創成科学研究科生命科学研究系メディカル情報生命専攻Department of Computational Biology and Medical Sciences, Division of Biosciences, Graduate School of Frontier Sciences, The University of Tokyo ◇ 〒277-8561 千葉県柏市柏の葉五丁目1番5号5-1-5 Kashiwanoha, Kashiwa-shi, Chiba 277-8561, Japan
3 東京大学大学院新領域創成科学研究科Graduate School of Frontier Sciences, The University of Tokyo ◇ 〒277-8561 千葉県柏市柏の葉五丁目1番5号5-1-5 Kashiwanoha, Kashiwa-shi, Chiba 277-8561, Japan