ホタルに代表される発光生物は我々の目を楽しませてくれるが,科学の進展にも大きく寄与してきた.蛍光タンパク質(GFPなど),発光酵素(ルシフェラーゼ),発光基質(ルシフェリン)などの言葉はライフサイエンスの研究者にとって今やきわめてなじみ深いものとなっている.本節では,生物の発光現象としての生物発光をあらためて整理し,この現象を支える生化学,そして応用技術の基盤となる光の定量測定について解説する.
1)生物による発光現象の生化学
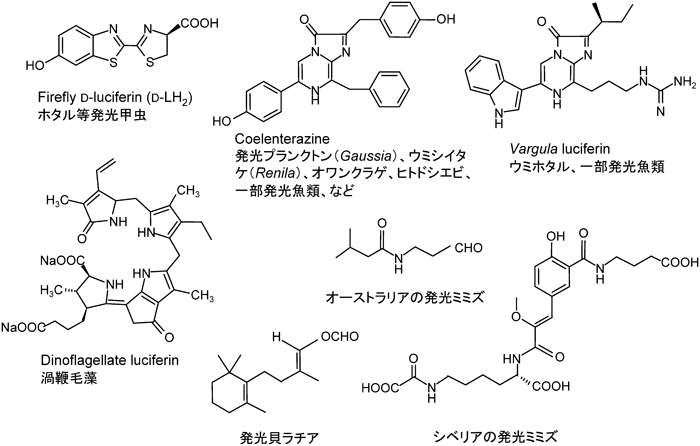
我々に身近なホタルやホタルイカ以外にも発光細菌,発光キノコ,オワンクラゲ,ヒオドシエビ,そしてウミホタルや夜光虫のようなプランクトンなど多くの発光生物が知られている.これまでに多くの発光生物からルシフェリンが単離構造決定され(図1),発光反応を触媒するルシフェラーゼも多数クローニングされている1–3).特に2014年にロシアのグループにより30年ぶりに新規のルシフェリンが発光ミミズから同定された4).しかし発光笠貝ラチアやミミズなどはルシフェラーゼのクローニングが未完であり,発光キノコのように基質の構造も明らかにされていないものもある.
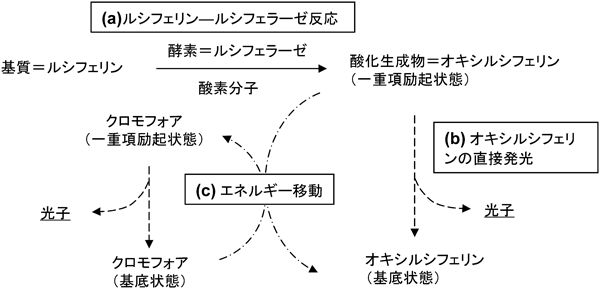
生物発光を担う化学反応はルシフェリン–ルシフェラーゼ反応(L–L反応)として広く知られ,光を発する機構は主に二つ考えられる(図2).一つ目はホタルやウミホタルのように,酸化生成物が蛍光性のクロモフォアを有しているため,L–L反応のみで蛍光放射すなわち発光が起こるものである.二つ目は,L–L反応生成物が蛍光放射を行うのではなく,クロモフォアを持つ蛍光タンパク質などに励起エネルギーを移動させ,蛍光放射により発光を行う機構である.代表例はオワンクラゲなど腔腸動物の蛍光タンパク質である.発光貝ラチアあるいは渦鞭毛藻などもL–L反応生成物が蛍光性のクロモフォアを持たないため,励起エネルギーの分子内あるいは分子間移動が想定されるが,詳細は不明である.
近年では「生物発光(bioluminescence)」という言葉がL–L反応とほぼ同義で用いられ,蛍光タンパク質とは別のものとして位置づけられているが,生物発光とは本来,発光生物が光る現象そのものを意味しており,発光生物であるオワンクラゲから単離された蛍光タンパク質GFP(緑色蛍光タンパク質)も生物発光のためのタンパク質の一つである.しかしサンゴなどの発光生物ではない腔腸動物からも蛍光タンパク質は単離されており,生物界では蛍光を発する生物の方が,自発的に発光する発光生物より広く分散している.
2)生物発光反応の酵素
発光生物は図1に示した多様なルシフェリンの酸化反応により発光しており,その反応はルシフェラーゼによって触媒される.すでに多くの発光生物のルシフェラーゼ遺伝子がクローニングされ,大腸菌等で合成した酵素の発光活性が確認されている.このようなルシフェラーゼ遺伝子は遺伝子発現のレポータ酵素等のバイオ研究用ツールとして,広く活用されている5–7).表1に,代表的なルシフェラーゼに関する情報をまとめた.
表1 発光酵素ルシフェラーゼ| 生物種 | 主な製品(購入先) | その他 |
|---|
| ホタルルシフェリンを基質とするもの | Photinus pyralis(北米産ホタル) | ルシフェラーゼレポータシステム(プロメガ) | 天然精製物がシグマアルドリッチ社より購入可能 |
| Pyrearinus termitilluminans(ブラジル産ヒカリコメツキムシ) | ELuc(東洋紡) | 量子収率が最大(0.61)pH非感受性 |
| Rhagophthalmus ohbai(イリオモテボタル) | SLG(東洋紡) | 3色ルシフェラーゼシステムの緑色,pH非感受性 |
| SLO(東洋紡) | 3色ルシフェラーゼシステムの橙色,pH非感受性 |
| Phrixothrix hirtus(鉄道虫) | SLR(東洋紡) | 3色ルシフェラーゼシステムの赤色,pH非感受性 |
| Pyrophorus plagiophthalamus(ジャマイカ産ヒカリコメツキ) | CBG,CBR(プロメガ) | 2色ルシフェラーゼシステム |
| Luciola cruciata(ゲンジボタル) | | リコンビナント酵素が和光純薬より入手可能 |
| Luciola mingrelica(欧州産ホタル) | | リコンビナント酵素が和光純薬より入手可能 |
| セレンテラジンを基質とするもの | Renilla reniformis(ウミシイタケ) | レニラルシフェラーゼ(プロメガ) | |
| Gaussia princeps(カイアシ類) | ガウシアシフェラーゼ(New England Biolabs) | 分泌型 |
| Oplophorus gracilirostris(トゲオキヒオドシエビ) | NanoLuc(プロメガ) | 市販品はfurimazineを基質として使用 |
| Metridia longa(カイアシ類) | MetLuc(タカラバイオ) | 分泌型 |
| ウミホタルルシフェリン(Cypridina luciferin)を基質とするもの | Vargula hilgendorfii(ウミホタル) | Cypridina Luciferase (New England Biolabs) | 分泌型 |
ルシフェラーゼは細胞内にとどまるタイプ(非分泌型)と分泌されるタイプ(分泌型)に分類される.ホタルやウミシイタケルシフェラーゼは非分泌型であり,最終的に細胞内で分解されるが,その分解速度は酵素によって異なっている.よって,細胞内イメージングに用いる場合には,その酵素の寿命も考慮しなくてはいけない.また,レポータアッセイで用いる場合も薬剤等の処理時間や発光を測定するタイミングについて最適化する必要がある.
一方,分泌型のルシフェラーゼは細胞内イメージングのようなツールには適さないが,たとえばウミホタルルシフェラーゼをレポータアッセイに用いれば,タンパク質は即座に細胞外に分泌されるため培地内のルシフェラーゼの活性を測るだけで対象遺伝子の発現レベルを評価できる8,9).また発光生物の体外に分泌されて発光するウミホタルルシフェラーゼは非分泌型に比べ酵素としての安定性が高いという特長があり,ラベル化試薬として有用である.
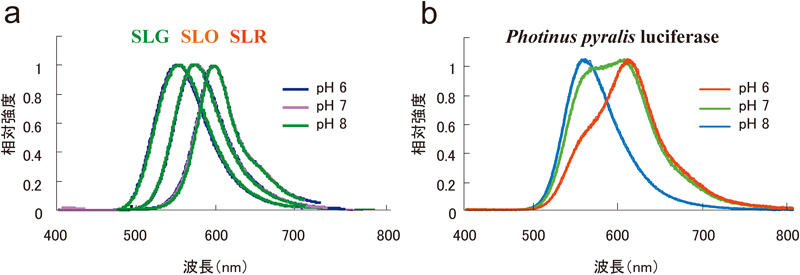
ホタルなど発光甲虫のルシフェラーゼは,溶液pHに対する感受性で生化学的に大きく二つに大別される(図3)10).最初にクローニングされた北米産ホタルのルシフェラーゼはpH感受性であり,細胞内のpHに依存し発光色および発光強度が大きく変化し,定量的な評価に影響を及ぼす.これに対し近年クローニングされたELucあるいは3色ルシフェラーゼのSLG,SLO,SLRのスペクトルはpHが変化しても一定のままであり,発光色の違いを利用した定量解析に適している.
3)生物発光反応の発光強度評価
明るく光る発光反応は,たとえばイメージングに応用すればより鮮明な画像が得られるなど,有用性はきわめて高い.そのため,より明るく光る発光基質あるいは酵素の開発は発光反応における重要な研究テーマの一つであり,発光強度の客観的評価はきわめて重要である.しかし発光強度の評価は原理的にも技術的にも煩雑なため,学術論文においても実験に基づく客観的な評価データを求められることはほとんどなかった.そこで,我々は発光反応における発光強度の評価について検討した.以下に,絶対光子数計測に基づく定量的な評価について説明する.
発光強度は全光子束Ilmすなわち反応溶液から放出される全光子数の時間密度が最も客観的な尺度といえる.Ilmは酵素反応速度論的にミカエリス・メンテンの式から以下のように表記することができる.ここで,QYは発光反応量子収率,kcatは酵素1分子あたりの反応速度,[E]は酵素の濃度,Kmはミカエリス定数,[S]は基質濃度である.QYは,基質1分子が反応を経て1光子を放出する確率と定義されており,反応により消費した分子数をS,生成した光子数をPhotonsとするとと表せる.通常の酵素反応では[S]はKmに比べて十分に大きいので,(1)式は次のように単純化することができる.
この式の意味するところは,生物発光反応の明るさは反応系に固有の活性パラメータであるQYとkcatによって明確に規定でき,活性状態にある酵素の濃度[E]に比例する,ということである.それでは,明るい反応系を構築するためには,QY,kcatあるいは[E]のどのパラメータを改善すればよいのだろうか.
古くからホタル生物発光反応の効率はきわめて高く,発光にあたり熱をほとんど放出しない冷光といわれてきたが,これは1959年に報告されたQY=0.88±0.25という数字に起因する11).しかしながら,この値には少なからず疑問があった.たとえば,当時は天然のルシフェリンが実験に用いられたはずだが,天然のルシフェリンは精製の過程でラセミ化するので純度は100%ではない.また,全光子束を測定することは現在でも決して容易ではない.我々は2008年に東京大学の秋山らのグループと共同でこの再実験に取り組み,QYが約0.4という結果を得た12).現在,多くの論文,教科書でもこの値が使われている.さらに他の発光甲虫や変異体のルシフェラーゼのQYを求めるため,そしてより簡便な方法で測定するため,新たに光放射計測の国家標準に準じた絶対測定系を構築し,発光甲虫ルシフェラーゼ群の酵素活性パラメータ解析を実施した.
表2に,ホタルのルシフェリンを基質とする代表的な酵素のQYとkcatの値を示す13).我々が測定したルシフェラーゼのうち,量子収率が最も高いのはブラジル産ヒカリコメツキムシ由来のルシフェラーゼELucで,その値は約0.6である.これに対し大部分の発光甲虫ルシフェラーゼは0.4~0.5,天然で赤色に光る鉄道虫の頭部由来のルシフェラーゼが最も小さく0.2弱である.このように量子収率は酵素の種類によって異なるが,発光色(発光極大波長)に依存し,発光極大波長が短いほどQYが大きくなるという相関が認められた.また酵素間のQYの違いは数倍程度でしかなかった.同様にkcatは天然型のルシフェラーゼではいずれもほぼ同じ桁数であった.
表2 発光甲虫ルシフェラーゼによるL–L反応の発光極大波長λMax,量子収率QYおよびkcat | ルシフェラーゼ | λMax (nm) | QY | kcat (s−1) |
|---|
| Photinus pyralis(北米産ホタル) | リコンビナント | 560 | 0.45 | |
| Photinus pyralis(北米産ホタル) | 天然精製物リコンビナント | 562 | 0.48 | 4.3×10−2 |
Pyrearinus termitilluminans
(ブラジル産ヒカリコメツキムシ) | | 539 | 0.61 | |
| Phrixothrix hirtus(鉄道虫) | リコンビナント | 625 | 0.15 | |
| Luciola cruciata(ゲンジボタル) | リコンビナント | 565 | 0.43 | |
| Luciola mingrelica(欧州産ホタル) | リコンビナント | 571 | 0.43 | |
| Pyrocoelia miyako(ミヤコマドボタル) |
| 野生型 | リコンビナント | 554 | 0.45 | 4.4×10−2 |
| N230S変異体 | リコンビナント | 606 | 0.21 | 1.5×10−2 |
| S199T変異体 | リコンビナント | 559 | 0.48 | 0.3×10−2 |
| S200A変異体 | リコンビナント | 556 | 0.46 | 0.3×10−2 |
以上の結果を(3)式に当てはめてみると,Ilmに関する支配的要因は[E],すなわち酵素濃度であることがわかる.逆に,酵素そのものには特別にQYやkcatが大きいものが存在するわけではなく,明るい発光反応系を得るためには[E]を大きくすることが重要となる.ここで注意する必要があるのは,[E]が単なる酵素濃度ではなく活性状態にある酵素の濃度であることである.したがって,明るい発光シグナルを得るためには,細胞内での発現量を向上させ酵素の絶対量を増加させるだけでなく,酵素の安定性を向上させるなどして[E]を高め,発光量Ilmそのものを増大させる必要がある.ただし,変異体ルシフェラーゼにおいてkcatが著しく低下するものがある.これは変異導入により活性が喪失していると解釈できる.変異導入によってルシフェラーゼの改善を試みる場合は,kcatが低下しないように留意する必要がある.
以上の結果はホタルルシフェリンを基質とする甲虫ルシフェラーゼの話であるが,セレテラジンなど他のルシフェリンを基質とするルシフェラーゼでは違った結果になる可能性もあり,今後の研究課題である.
4)発光の絶対計測とスペクトル測定
前項で述べたルシフェラーゼの酵素活性パラメータQYおよびkcatを決定するためには,全光子束Ilmを実験的に測定する必要がある.kcatはラインウィーバー・バークプロットから求められる最大反応速度Vmaxと[E]から求められる.反応速度Vは反応系全体の基質消費速度と同義であり,全光子束とQYから導くことができる.
(2)式で示すPhotonsは,全光子束を時間積分することにより測定する.Sは基質濃度より比較的容易に決定できるが,反応によって消費された基質分子数を求める必要がある.我々は5分程度の測定時間内にすべての基質分子の反応が完了するよう基質分子数をきわめて低く設定し,反応開始前から計測を行うことで,全光子数を実測している.100 µLの反応液中の基質分子は10−13 mol(6.02×1010分子)であり,通常の酵素発光反応溶液での基質濃度と比べて4~5桁程度低い.
ここで,光の検出器について少し掘り下げて説明する.光の検出器はバイオイメージングなどでも多くのものが使われているが,発光反応の微弱な光放射を検出するためには,感度の高い光電子増倍管(PMT)が用いられる.アバランシェ・フォトダイオードなどの半導体検出器もPMTより量子効率(1光子が電気シグナルを生む確率)が高く高感度であるが,きわめて微弱な光を測定するためにはノイズ(ダークカウント,暗電流値)が低いことが重要であり,この点ではPMTの方が優れている.実際,ルミノメータと呼ばれる汎用型の測定装置ではPMTが主流である.一方で,スペクトルの測定で用いられるマルチチャンネル分光計ではCCDのような半導体検出器が使われる.発光強度が時間変化しやすい発光反応の場合は,すべての波長のシグナルを同時に測定できるマルチチャンネル式が有効であるが,マルチ計測のためには検出器のアレイ化が必要であり,これには半導体検出器が適している.また検出器は種類を問わず分光応答度(いわゆる波長感度)特性があり,これにも留意する必要がある.たとえばPMTは青色~緑色領域の応答度は高いが,赤色~赤外域での応答度はほぼゼロである.このためルミノメータを用いて測定した赤色ルシフェラーゼの発光シグナルが緑色に比べて小さいからといって,必ずしも赤色の方が暗いわけではない.
ルミノメータは通常,PMT内部に生成する光電子の数が表示される(カウント値).ルミノメータで全光子束の絶対計測を行うためには,カウント値と発光反応溶液サンプルの全光子束との相関,すなわち全光子束応答度の校正が必要となる.この校正は全光子束が既知の参照用発光反応溶液サンプルを用いて実施する.参照用発光反応溶液サンプルの全光子束は,積分球式マルチチャンネル分光測定装置を用いて決定する14).この装置は光放射に関する国家標準に準じており,積分球を用いることで,球の外部に設置した標準電球から導入された放射束と,球の内部に設置するサンプルからの全放射束を,マルチチャンネル分光放射計により同等に測定することができる.装置の分光応答度は分光放射照度標準電球を用いて校正することで,他の国家標準にトレーサブルな全光子束測定系となっている.不確かさは±10~15%(包含係数k=2)程度である.この積分球式マルチチャンネル分光測定装置は残念ながら感度が低いため,QYの測定にはルミノメータを用いる.ただしルミノメータには波長感度特性があるので,QY測定の対象となる発光反応溶液サンプルのスペクトルごとに参照溶液を調製し,全光子束応答度の校正を行う.
ところで,分光計測において,以下の点を留意する必要がある.たとえば入射スリットを広くすると,測定されるスペクトルは実際よりも幅が広くなるが,バイオ分野では概して微弱な光を効率よく測定するため,スリット幅を大きくしていることが多い.また光検出器の波長感度特性,そして応答度非直線性などのため,スペクトルの生データは歪んでいる.発光反応を測定するためには分光器の特性を理解したうえで妥当性評価を行い,標準光源を用いた適切な校正が欠かせない.
実験動物を用いた薬効評価や安全評価試験は3Rの理念のもと細胞を用いたセルベースアッセイに代替されつつある.初期段階における薬効や安全評価のために行われるセルベースアッセイは細胞数や細胞形状の変化を指標にするものであったが,これでは判定性があいまいかつスループット性に問題があった.そこで,これらを評価する分子マーカーの遺伝子発現を指標とするレポータアッセイが行われるようになった.第1世代では対象となる遺伝子のプロモータ配列の下流にたとえばβ-ガラクトシダーゼ遺伝子を挿入したベクターを構築,これを細胞に導入する.対象遺伝子の発現が活性化された場合,本タンパク質が生産されるので,呈色反応により発色させ,発色性によって遺伝子発現量を推定した.他にクロラムフェニコールアセチルトランスフェラーゼ遺伝子を用いたCATアッセイなどもあるが,定量性や簡便性が低いため,第2世代としてルシフェラーゼや蛍光タンパク質GFPを用いたレポータアッセイが行わるようになった.二つのレポータタンパク質は簡便性には優れているが,定量性という意味では発光量という数値で評価できるルシフェラーゼを用いたものが,より精度の高いセルベースアッセイとして普及した.本節では表1に取り上げるルシフェラーゼ群を中心に説明する.
ルシフェラーゼを用いたセルベースアッセイでは,細胞それ自体の変化などを評価できる恒常的に発現するコントロール遺伝子を併せて比較することで,より再現性を高めることができる.つまり二つの遺伝子の発現を指標にセルベースアッセイを行う方法である.プロメガ社はホタルルシフェラーゼとレニラルシフェラーゼを併せたデュアルレポータアッセイを商品化した.しかしながら,この方法では二種類のルシフェリンを用いるため発光測定が煩雑であり,かつ,二つのルシフェラーゼの細胞内半減期が著しく異なる点,また発光反応のバックグラウンドが異なる点が問題となっていた.そこで,我々は同じホタルルシフェリンを共通の発光基質としながらも発光色の異なる発光甲虫ルシフェラーゼに着目し,各々の発光色を分離することでレポータアッセイを行うマルチカラールシフェラーゼアッセイを開発した15).
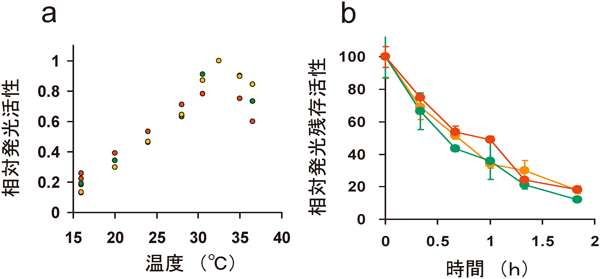
前述したようにホタルルシフェリンに対して,溶液のpHに依存して発光スペクトルが変化するもの(主にホタルルシフェラーゼ)と,変化しないもの(主に発光甲虫由来ルシフェラーゼ)がある.マルチカラールシフェラーゼアッセイは後者を利用したもので,3色(緑,橙,赤色)のルシフェラーゼ群を用いたものである.本ルシフェラーゼ群は,1)最大発光波長が30 nm程度異なり,色フィルターで分離計測可能,2)従来のデュアルレポータと異なり,ルシフェリンが1種類,3)三つのルシフェラーゼ活性の温度依存性,半減期がほぼ同様であるという特徴を持つ(図4).
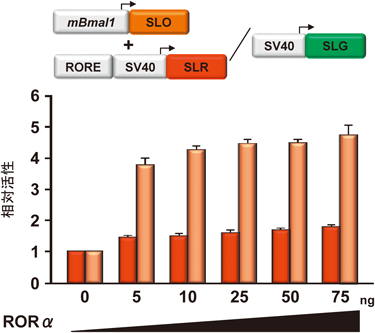
2004年,北山らは我々がクローニングした赤,緑色発光甲虫ルシフェラーゼを用いてシアノバクテリアの二つの遺伝子発現を同時計測した16).計測には2本の光電子増倍管を用いて,二つの遺伝子の発現を連続的に測定した.同年,我々は哺乳類細胞内の体内時計に関して,時計遺伝子Bmal1プロモータとBmal1プロモータ内のRev-Erb and ROR response element(RORE)の配列を上流に導入したSLR,SLGを用いて,転写因子RORαに対する応答性が異なることを明らかにした17).この際,内部標準としてThymidine kinase(TK)プロモータ下流にレニラルシフェラーゼを導入したベクターを用いた.翌年,さらに内部標準としてSLOを用いることで,三つの遺伝子発現を3発光色のルシフェラーゼで同時に計測することにも成功した(図5)15).2005年以降,BranchiniらやOguraらはホタルルシフェラーゼの変異体を用いることで2発光色による2遺伝子発現計測を行った18,19).
セルベースアッセイの大きな目的は創薬や毒性スクリーニングにあるが,重要な点はハイスループット性と再現性である.前者を満足するためにはできるだけシンプルな方法とし,計測装置とのマッチングが重要である.一方,後者には細胞の安定性を担保し,再現性を確保するための適切な内部標準が必要となる.創薬スクリーニングを行った例としてDavisらは,nuclear factor-κB(NF-κB)シグナル伝達経路の活性化を調整するinhibitor of NF-κB(IκBα)の細胞内安定性を高める化合物を同定するためCBGをIκBαと融合し,一方,内部標準としてCBRを用い,二つのバンドパスフィルターを組み合わせたCCDカメラを検出系として用いることで1536穴プレートを対象としたスクリーニング系を構築した20).一方,我々の開発したマルチカラーレポータアッセイを用いて,皮膚感作性の分子マーカーであるinterleukin 8(IL-8)の遺伝子発現をSLO,内部標準としてglyceraldehyde 3-phosphate dehydrogenase(G3PDH)のプロモータの発現をSLRで評価する細胞が樹立された.本細胞を用いることでヨーロッパ実験動物代替法センターが規定した22化合物に対して82%の再現性,88%の毒性評価信頼度で解析できた21).本マルチカラールシフェラーゼアッセイを用いた実験動物代替法は現在,OECDのテストガイドラインに申請準備中である.
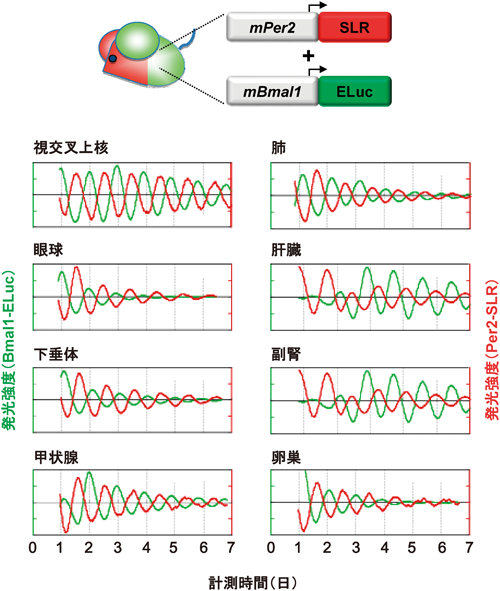
これまで紹介したルシフェラーゼアッセイでは,最初のシアノバクテリアの例を除いて細胞に刺激を加えたのち,ある一定時間後,細胞を破砕し,その段階までに合成され,細胞内で分解されなかったルシフェラーゼの発光量からプロモータの活性を評価した.しかしながら,ホタル発光系のユニークさは,他の発光系のルシフェリンと異なり,ホタルルシフェリンが培養液中で高い安定性を示し,かつ,生きた細胞内に徐々に浸透し細胞内で生物発光反応を起こすことができる点である.具体的には測定開始1週間後でも半減することはなく,一定のシグナルを得ることができる.一方,セレンテラジンやウミホタルルシフェリンの培地内や生体内での寿命は短い.よってホタルルシフェリンを用いれば,生きた細胞内の遺伝子発現を長時間にわたり解析できる.我々は逆位相の関係で発現することが知られている時計遺伝子Bmal1とPer2をそれぞれELuc,SLRによってリアルタイムに解析する方法を開発した.具体的には,これらの組み合わせを持ったトランスジェニックマウスを作製し,世界で初めて2色の発光で体内時計を知らせるマウスの作製に成功,本マウスの各組織におけるPer2とBmal1発現の周期と位相は組織ごとに異なるものの,正確な逆位相を示すことを明らかにした(図6)22).また,Bmal1プロモータの制御下でELucが発現するトランスジェニックマウスとSLRが発現するRat-1線維芽細胞を個別に作製し,トランスジェニックマウスより単離した組織とRat-1細胞の共培養を行い,両者のリズム発現を同一ディッシュ内でリアルタイムに計測し,組織-細胞間の相互作用解析に成功した23).このように,マルチカラールシフェラーゼアッセイは細胞,組織レベルで複数の遺伝子発現を明らかにできるユニークな方法として活用され続けている.
光を用いた細胞機能解析は顕微鏡の機能の向上とGFPや量子ドットなどの蛍光プローブ群の開発により進展した.生物発光もまた,発光に特化した顕微鏡を用いることで細胞内の機能解析が十分可能である.蛍光と発光の違いは明確であり,蛍光は励起光が必要であるが,発光は化学反応であり,励起光を必要としない.蛍光では,励起によって強いシグナルを出すことから細胞内の局所における短い時間の可視化(イメージング)が可能である.一方,生物発光では生体内の光が届きにくい領域での変化をとらえることができるもののシグナルが弱く,光を積算する必要があるため,蓄積された画像をパラパラ漫画でみるようなイメージとなる.ただし,励起光照射による細胞の損傷を考慮する必要がなく,数日以上にわたる長時間の観察が可能である.深さ方向の優位さの一例として,我々はホタルルシフェラーゼおよびGFPをそれぞれ定常的に発現するマウスを用い,それらをドナーとして骨髄移植を行った24).次に移植マウスの脳内にリポ多糖(LPS)を加え損傷を起こし,骨髄細胞からの浸潤現象をイメージングした.ルシフェラーゼを用いた場合,損傷後,時間経過とともに修復が行われることを光量の変化で可視化することができたが,この変化は蛍光では観察できなかった.多くの研究においてルシフェラーゼを導入したがん細胞をマウス等に移植し,in vivo発光イメージングによって薬剤効果などを検証する方法が一般的であるのは,まさにこの深さ方向の優位性による25).
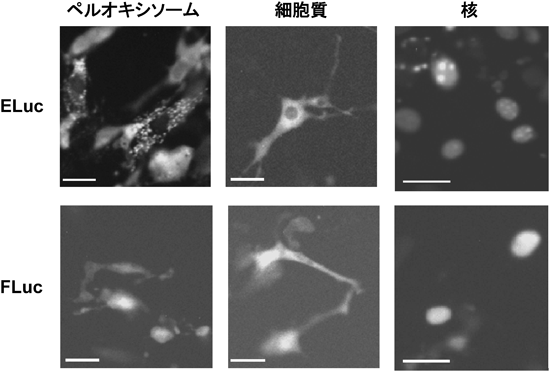
さらに表2において溶液中で高い安定性と高い量子収率を持つことが示されたELucが細胞レベルでのイメージングでも有用であることを確認した26).前述したように,発光甲虫由来のルシフェラーゼのスペクトルは反応場のpHや温度に依存しない.ELucはホタル由来のルシフェラーゼに比べて約10倍以上の発光強度が得られ,露光時間を大幅に減らすとともに高い空間分解を示すことに成功した(図7).また,細胞内の局在シグナルをつけてもシグナルが減ずることはなく,細胞小器官の観察も可能である.これを用いれば容易に細胞の動的な変化を1週間程度連続的に可視化できる.また,1細胞レベルでの遺伝子発現も定量的に解析することが可能となる.
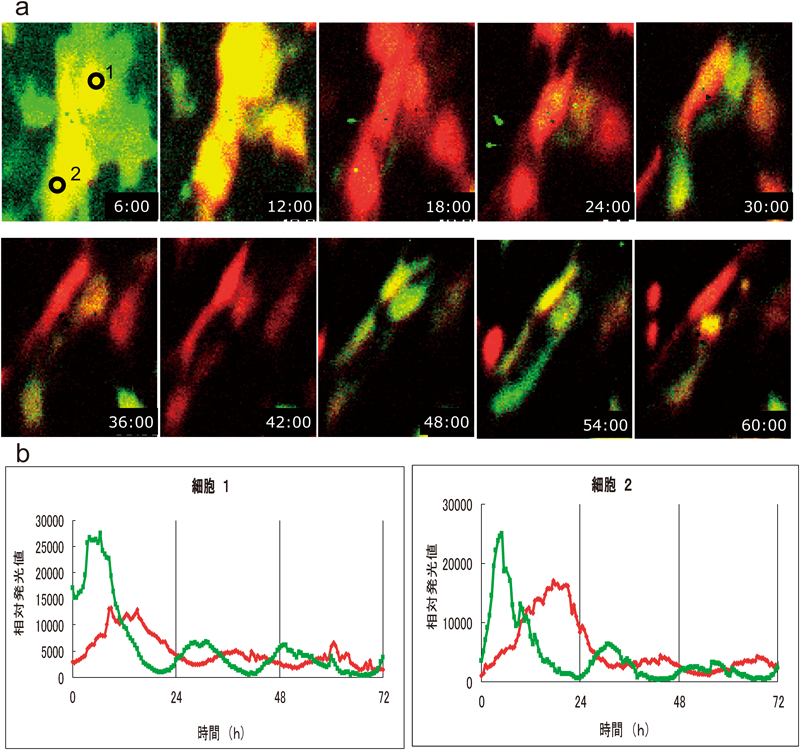
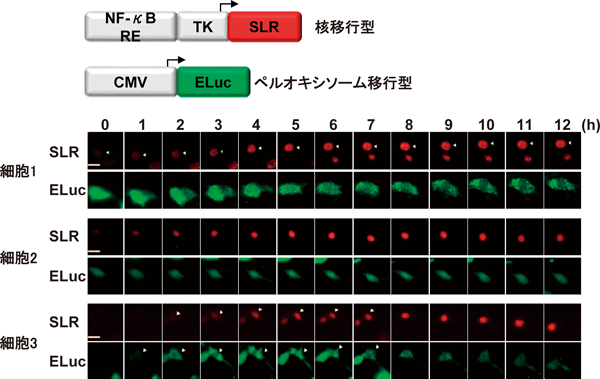
発光に特化した顕微鏡でも異なる発光色の光を色フィルターにより分離することでマルチカラーイメージングが可能である.我々は細胞集団レベルで観察した時計遺伝子Bmal1とPer2の動きを二つの発光色で解析する手法を1細胞観察に適用した.図8は発光細胞の画像(a)と1細胞ごと(b)に解析した例である.1個の細胞の中の二つの対象遺伝子の発現パターンを1週間程度観察することができた27).しかしながら,二つの発光色の強度レベルが近すぎたため,色分離が難しい時間帯が存在した.そこで,空間的にも分離することで,より詳細な1細胞2遺伝子変動の解析を行った.NF-κB応答配列(NF-κB RE)の下流に核内に移行するSLR,内部標準としてCytomegalovirus(CMV)プロモータの下流にペルオキシソーム内に移行するELucを用いた.tumor necrosis factorによりNF-κBを活性化すると核内の赤色発光は増加するが,内部標準レポータであるペルオキシソーム内の緑色発光はほぼ一定であり,1細胞内の細胞小器官レベルで二つの遺伝子発現の変化を同時に可視化できた(図9)28).細胞小器官の違いでルシフェラーゼの寿命が異なり,遺伝子発現情報が異なるのか,ルシフェラーゼを入れ替えて検証したが違いはなく,小器官の局在,発光色の異なるルシフェラーゼで細胞情報の可視化が異なることはなかった.
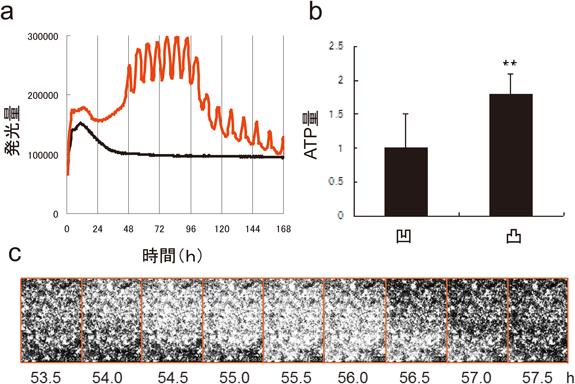
生物発光を用いて細胞機能を解析する際に考慮すべき点は,発する光の意味を十分に理解することである.たとえば発光甲虫の光で遺伝子発現の変化を可視化するにはルシフェリン,ATP,酸素が化学反応を律速させないような十分量が必要である.たとえば,酸素濃度が低下するがん細胞では,発現の低下によるルシフェラーゼ量の減少ではなく,酸素量の減少で発光強度が低下することがある29).我々が観測した例では,ATDC5細胞をインスリンで分化させる段階の遺伝子発現を可視化しようと試みたが,すべてのプロモータ配列のレポータベクターで,インスリン添加2,3日後に4時間周期の発光振動が数日間みられた(図10a).当初は遺伝子発現がみな同調すると考えたが,ATP量の変動に目を向けたところ,急激な軟骨分化過程においてATPが4時間周期で変動,そのために発光が変化したことが明らかになった(図10b)30).この際,細胞間に光のウェーブが観察され(図10c),軟骨分化過程において,細胞間コミュニケーションが存在する可能性が示唆された.この場合はルシフェリン,ルシフェラーゼ,酸素量は十分であるが,ATPが変化することによる発光強度の変化となる.ここでヒントを得て,我々はセファロース表面にルシフェラーゼを結合させ,それをマウス皮下に注入し,炎症に伴いデンジャーシグナルとしてATPが放出される過程の可視化に成功した31).いずれにしても光シグナルの変化が何に起因するか十分に考慮しながら生物発光を用いることが肝要である.
引用文献References
1) Shimomura, O. (2006) Bioluminescence: Chemical Principles And Methods, World Scientific Pub Co Inc.
2) Hastings, J.W. (2013) Bioluminescence: Living Lights, Lights for Living, Harvard University Press.
3) 今井一洋,近江谷克裕(2006)バイオ・ケミルミネッセンスハンドブック,丸善.
4) Petushkov, V.N., Dubinnyi, M.A., Tsarkova, A.S., Rodionova, N.S., Baranov, M.S., Kublitski, V.S., Shimomura, O., & Yampolsky, I.V. (2014) Angew. Chem. Int. Ed. Engl., 53, 5566–5568.
5) Roda, A., Guardigli, M., Pasini, P., & Mirasoli, M. (2013) Anal. Bioanal. Chem., 377, 826–833.
6) Frank, L.A. & Krastiskaya, V.V. (2014) Adv. Biochem. Eng. Biotechnol., 144, 175–197.
7) Roura, S., Gálvez-Montón, C., & Bayes-Genis, A. (2013) J. Cell. Mol. Med., 17, 693–703.
8) Nakajima, Y., Kobayashi, K., Yamagishi, K., Enomoto, T., & Ohmiya, Y. (2004) Biosci. Biotechnol. Biochem., 68, 565–570.
9) Wu, C., Suzuki-Ogoh, C., & Ohmiya, Y. (2007) Biotechniques, 42, 290–292.
10) Ohmiya, Y. (2005) Jpn. J. Appl. Phys., 44(9A), 6368–6792.
11) Seliger, M.H. & McElroy, W.D. (1960) Arch. Biochem. Biophys., 88, 136–141.
12) Ando, Y., Niwa, K., Yamada, N., Irie, T., Enomoto, T., Kubota, H., Ohmiya, Y., & Akiyama, H. (2008) Nat. Photonics, 2, 44–47.
13) Niwa, K., Ichino, Y., Kumata, S., Nakajima, Y., Hiraishi, Y., Kato, D., Viviani, V.R., & Ohmiya, Y. (2010) Photochem. Photobiol., 86, 1046–1049.
14) Niwa, K., Ichino, Y., & Ohmiya, Y. (2010) Chem. Lett., 39, 291–293.
15) Nakajima, Y., Kimura, T., Sugata, K., Enomoto, T., Asakawa, T., Kubota, H., Ikeda, M., & Ohmiya, Y. (2005) Biotechniques, 38, 891–894.
16) Kitayama, Y., Kondo, T., Nakahira, Y., Nishimura, H., Ohmiya, Y., & Oyama, T. (2004) Plant Cell Physiol., 45, 109–113.
17) Nakajima, Y., Ikeda, M., Kimura, T., Honma, S., Ohmiya, Y., & Honma, K. (2004) FEBS Lett., 565, 122–126.
18) Branchini, B.R., Southworth, T.L., Khattak, N.F., Michelini, E., & Roda, A. (2005) Anal. Biochem., 345, 140–148.
19) Ogura, R., Matsuo, N., Wako, N., Tanaka, T., Ono, S., & Hiratsuka, K. (2005) Plant Biotechnol., 22, 151–155.
20) Davis, R.E., Zhang, Y.-Q., Southall, N., Staudt, L.M., Austin, C.P., Inglese, J., & Auld, D.S. (2007) Assay Drug Dev. Technol., 5, 85–103.
21) Takahashi, T., Kimura, Y., Saito, R., Nakajima, Y., Ohmiya, Y., Yamasaki, K., & Aiba, S. (2011) Toxicol. Sci., 124, 359–369.
22) Noguchi, T., Michihata, T., Nakamura, W., Takumi, T., Shimizu, R., Yamamoto, M., Ikeda, M., Ohmiya, Y., & Nakajima, Y. (2010) Biochemistry, 49, 8053–8061.
23) Noguchi, T., Ikeda, M., Ohmiya, Y., & Nakajima, Y. (2012) PLoS ONE, 7, e37093.
24) Akimoto, H., Kwon, H.J., Ozaki, M., Yasuda, K., Honma, K., & Ohmiya, Y. (2009) Biochem. Biophys. Res. Commun., 380, 844–849.
25) Müller, J., Wunder, A., & Licha, K. (2013) Recent Results Cancer Res., 187, 221–246.
26) Nakajima, Y., Yamazaki, T., Nishii, S., Noguchi, T., Hoshino, H., Niwa, K., Viviani, V.R., & Ohmiya, Y. (2010) PLoS ONE, 5, e10011.
27) Kwon, H.J., Enomoto, T., Shimogawara, M., Yasuda, K., Nakajima, Y., & Ohmiya, Y. (2010) Biotechniques, 48, 460–462.
28) Yasunaga, M., Nakajima, Y., & Ohmiya, Y. (2014) Anal. Bioanal. Chem., 406, 5735–5742.
29) Khalil, A.A., Jameson, M.J., Broaddus, W.C., Lin, P.S., Dever, S.M., Golding, S.E., Rosenberg, E., Valerie, K., & Chung, T.D. (2013) Int. J. Mol. Imaging, 2013, 287697.
30) Kwon, H.J., Ohmiya, Y., Honma, K.I., Honma, S., Nagai, T., Saito, K., & Yasuda, K. (2012) Cell Death Dis., 3, e278.
31) Takahashi, T., Kimura, Y., Niwa, K., Ohmiya, Y., Fujimura, T., Yamasaki, K., & Aiba, S. (2013) J. Invest. Dermatol., 133, 2407–2415.
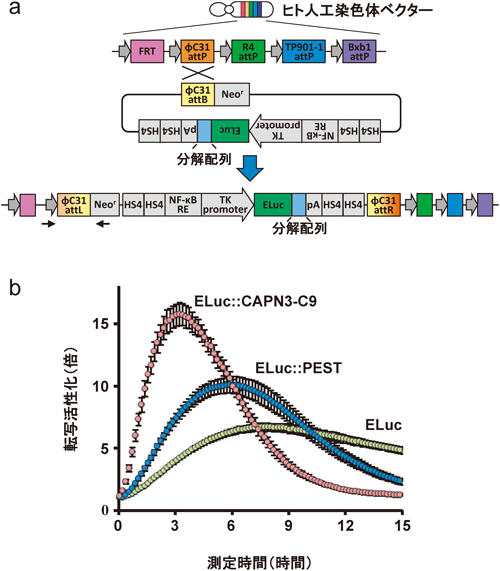
32) Yasunaga, M., Murotomi, K., Abe, H., Yamazaki, T., Nishii, S., Ohbayashi, T., Oshimura, M., Noguchi, T., Ohmiya, Y., & Nakajima, Y. (2015) J. Biotechnol., 94, 115–123.