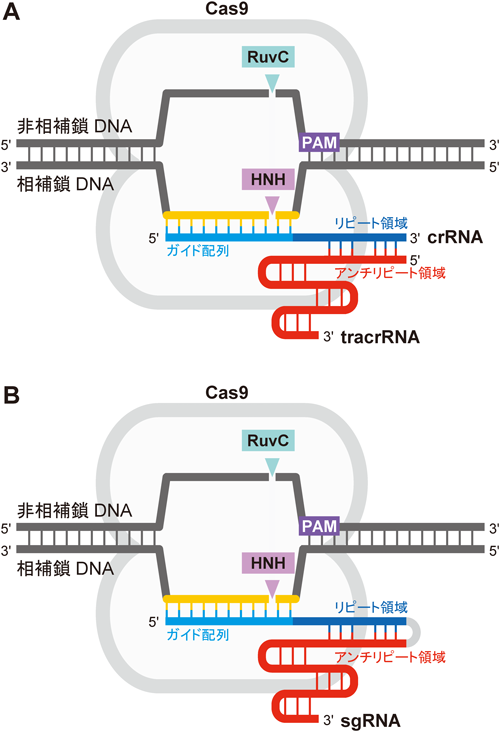
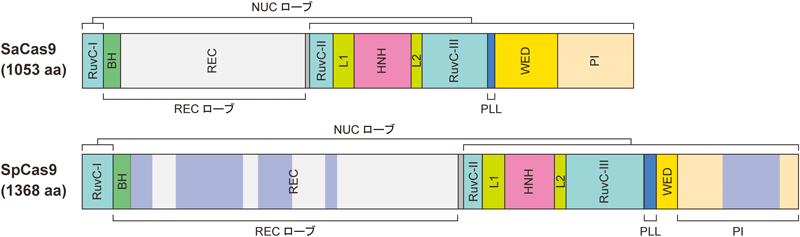
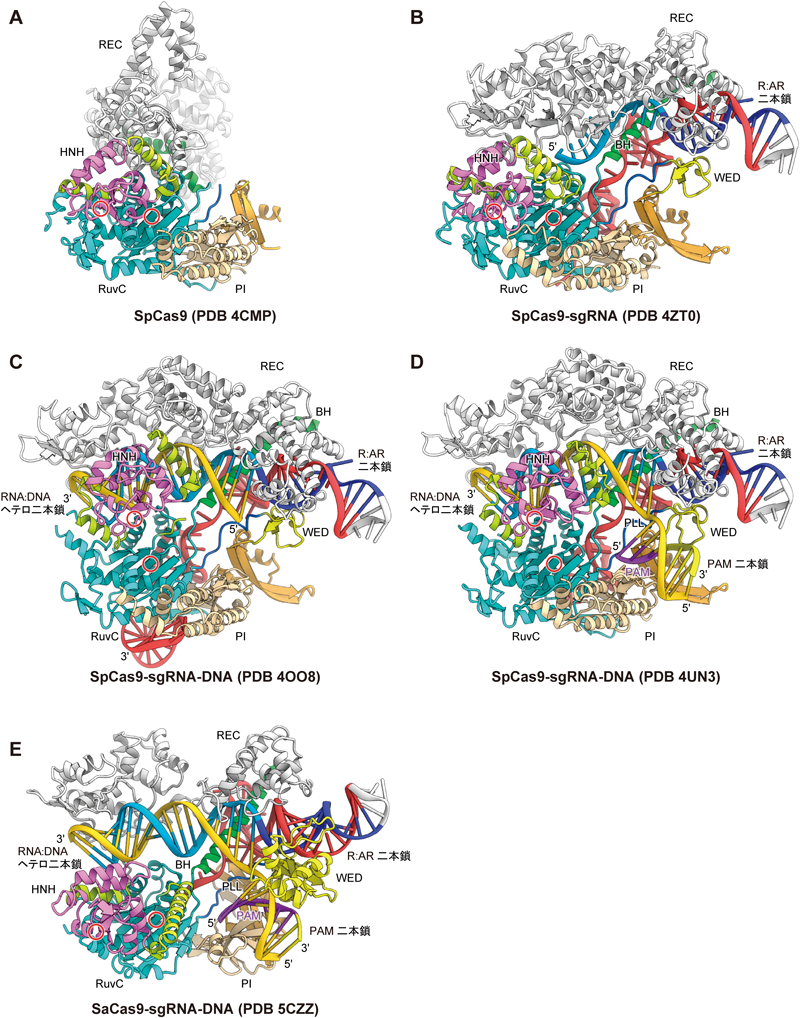
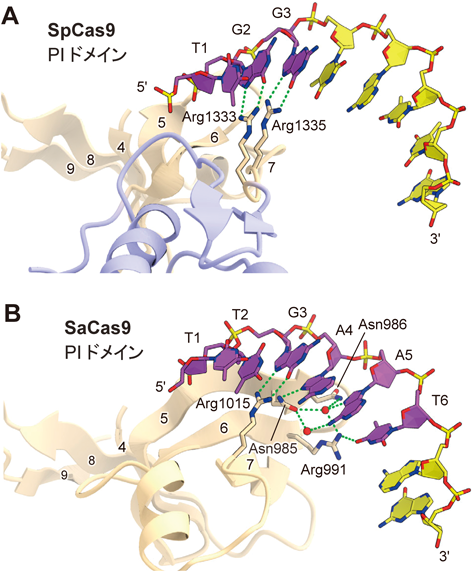
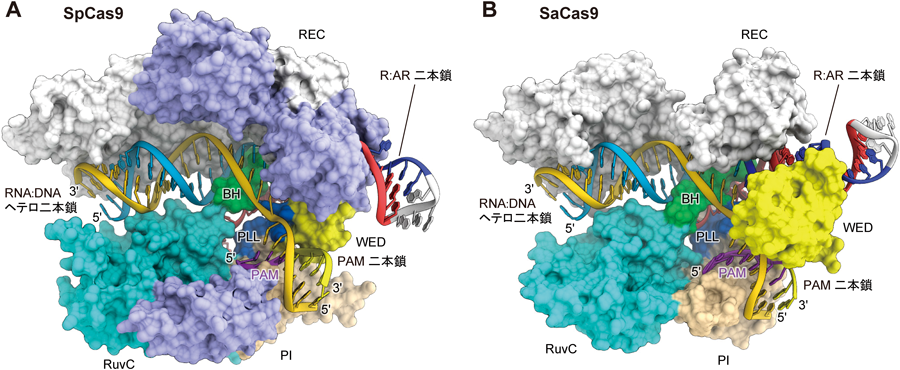
CRISPR-Cas9の構造と機能Structure and function of CRISPR-Cas9
1 東京大学大学院理学系研究科Graduate School of Science, The University of Tokyo ◇ 〒113-0033 東京都文京区本郷七丁目3番1号Hongo 7-3-1, Bunkyo-ku, Tokyo 113-0033, Japan
2 国立研究開発法人科学技術振興機構戦略的創造研究推進事業さきがけ「ライフサイエンスの革新を目指した構造生命科学と先端的基盤技術」研究領域Research Area “Structural Life Science and Advanced Core Technologies for Innovative Life Science Research”, Precursory Research for Embryonic Science and Technology (PRESTO), Japan Science and Technology Agency (JST)