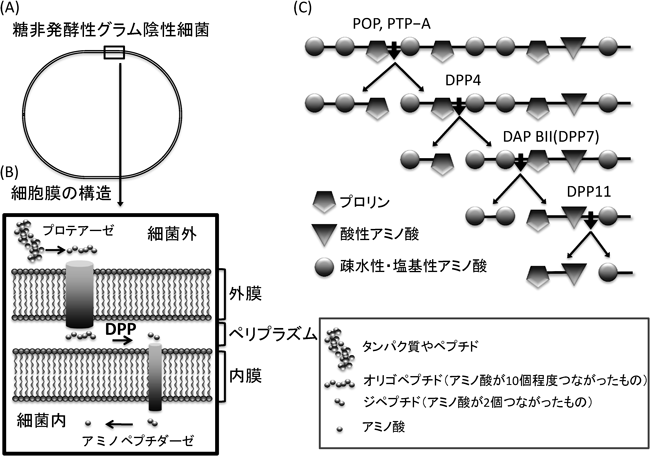
多剤耐性菌のStenotrophomonas maltophiliaや歯周病原菌のPorphyromonas gingivalis,Porphyromonas endodontalisなどの糖非発酵性グラム陰性細菌は,糖や炭水化物ではなくタンパク質やペプチドを炭素源および栄養源とする細菌として知られている1).これらの細菌の細胞壁は外膜,ペリプラズム,内膜の三層構造により構成されている(図1A, B).これらの細菌では細胞外にエンドペプチダーゼなどを分泌しており,外部のタンパク質やペプチドはそれらの酵素の働きでオリゴペプチドにまで分解されたのち,外膜を通ってペリプラズムに達する.そして,オリゴペプチドはペリプラズムに局在するジペプチジルペプチダーゼ(dipeptidyl peptidase: DPP)群によってジペプチドあるいはトリペプチドに分解される.これらのジペプチドやトリペプチドはペプチドトランスポーターによりアミノ酸単体よりも優先的に細菌内に輸送されることから,DPP群は細菌の生育や増殖に特に重要な役割を持つことが知られている2).微生物DPP群の酵素は,その基質特異性に応じてペプチドを認識し,さまざまなジペプチドを産生する3–5)(図1C).一般に,DPPといえば糖尿病の標的分子として知られる“DPP4”を指すことが多いが,本稿では,糖非発酵性グラム陰性細菌のペプチド代謝系に関与するDPP群の中のS46ファミリーと呼ばれる新規DPPについて主に紹介する.
糖非発酵性グラム陰性細菌のペプチド代謝に関係するDPPは主にペリプラズムに局在している6).歯周病菌はDPP4,DPP5,DPP7,DPP11,PTP(proryl tripeptidyl aminopeptidase,トリペプチドを産生する)を持つ.これらのDPPは,プロテアーゼデータベースMEROPS7)で触媒残基の順番や基質特異性およびアミノ酸配列相同性などに基づいて,DPP4,DPP5,PTP(POP(prolyl oligopeptidase))はClan SC S9ファミリー,DPP7,DPP11はClan PA S46ファミリーの二つのファミリーに分類される.Clan SC S9ファミリーのDPPは,主に基質切断部位から基質アミノ末端側の最初のアミノ酸であるP1位のプロリンあるいはアラニンに基質特異性を持ち,βプロペラ構造の制御ドメインとα/β構造の触媒ドメインにより構成される(図2D).S9ファミリーにはヒトDPP4も属していて,S9ファミリー内の酵素は,立体構造上も高い相同性があることが知られている8).一方,Clan PA S46ファミリーは,P1位が塩基性,酸性,疎水性のアミノ酸に対する広い基質特異性を持つことから,多様なオリゴペプチドを分解する酵素であると考えられる3).また,キモトリプシンをはじめとする他のClan PA Sファミリーの酵素がすべて基質の内部から切断するエンド型の酵素であるのに対してS46ファミリーの酵素のみが基質の端から切断するエキソ型である.
これまで,S9ファミリーに属するプロリン特異性DPPであるヒトDPP4やPTPに関して生化学的解析や立体構造解析などの詳細な研究が行われ,その成果が創薬分野にも利用されている.S46ファミリーのDPPに関しては,ヒトに類縁酵素がないこともあって顧みられることはあまりなかった.筆者らは,2014年にこれまで立体構造が明らかにされていなかったS46ファミリーの酵素であり疎水性アミノ酸および塩基性アミノ酸を認識するDAP BIIの触媒残基と立体構造の決定に成功し9),さらに2015年には酸性アミノ酸を認識するDPP11の立体構造解析に成功した.その結果,基質特異性の異なる二つのS46ファミリー酵素のサブサイトと基質P1位の側鎖間の相互作用様式の違いを分子レベルで明らかにすることができた10).
1996年に長岡技大の小笠原博士らは豆腐工場廃液よりジペプチド高生産菌であるPseudoxanthomonas mexicana WO24というグラム陰性菌を見いだした.この細菌は,DAP BI,DAP BII,DAP BIII,DAP IV,POPの5種のDPPを有していた3).これらのDPPは歯周病菌のDPP群と同様に二つのファミリーに分けられ,DAP BII以外は生化学的解析およびアミノ酸配列解析によってMEROPS Clan SC S9ファミリーに属していた(図2D).一方,DAP BIIは他のDPPに比べて基質特異性が広く,X-Pro結合をも切断するエンド活性を示さない新規のDPPだった.2001年にジョージア大のBanbula博士らもDAP BIIに類似の酵素を歯周病菌DPP7として発見し4),プロテアーゼデータベースであるMEROPSにClan PA S46ファミリーが新たに登録された.さらに,2011年には,長崎大の根本博士と岩手医大の原賀博士らにより酸性アミノ酸を主に認識するDPP11が歯周病菌から同定された5).これらS46ファミリーの酵素は塩基性あるいは疎水性のアミノ酸を認識するDAP BIIおよびDPP7タイプのものと酸性アミノ酸のみを認識するDPP11タイプのものに分けられる.他のClan PA Sファミリーの酵素は200残基程度のキモトリプシンフォールドを持つエンド型の酵素であるが,S46ファミリーの酵素は他のClan PA Sの酵素と異なり,触媒残基が500残基程度の範囲に広く分布するエキソ型の酵素であり,どのような触媒ドメイン構造を持つのかが不明であった(図2E).
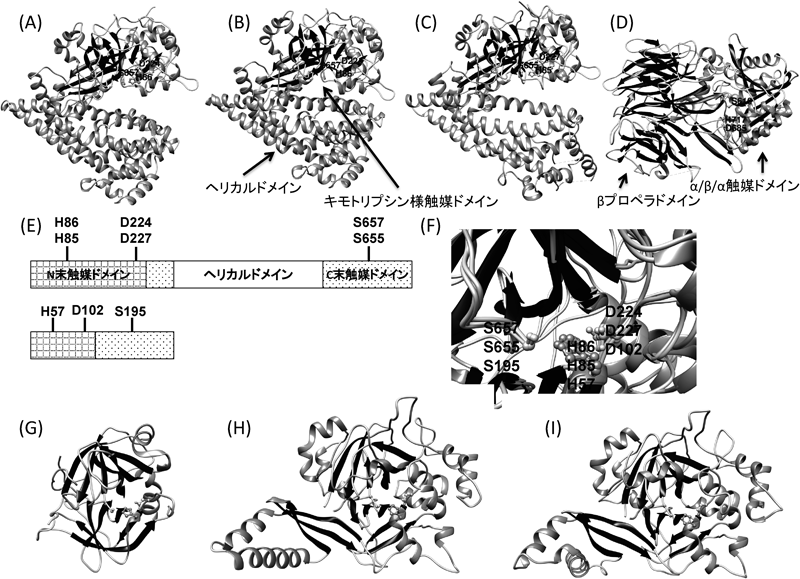
2014年に筆者らはClan PA S46に属する酵素として初めてDAP BIIの結晶構造を決定した.得られたDAP BIIの全体構造は球状のようにもみえたが,キモトリプシン様ドメインと多くのヘリックスを持つαヘリカルドメインともいうべき二つのドメインが上下に配置され,片側が蝶番のように結合しゲームキャラクターの“パックマン”のような構造をとっていた(図2A).そして,キモトリプシン様の触媒ドメインは,N末端側とC末端側の二つの領域に分断され,触媒ドメインのN末端側のβバレル側に触媒3残基11)のうちH86およびD224の二つが,もう一つのC末端側のβバレル側にもう一つのS657が属し,二つのβバレルの間には新規構造のαヘリカルドメインが挿入されていた(図2E).DAP BIIのトリプシン様ドメインと芳香族アミノ酸に基質特異性のあるキモトリプシンとのアミノ酸配列の相同性は15%と非常に低いものの,立体構造の重ね合わせによる平均二乗偏差は1.84 Åと小さく,触媒3残基の位置は完全に保存されていた(図2F~H).二つに分断された領域が一つの触媒ドメインを形成し,しかもキモトリプシンとの立体構造上の相同性が保たれていることは,分子進化を考える上で非常に興味深い.
最初に構造決定したDAP BIIのアポ体では,基質がどのように結合しているかがわからず,DAP BIIの触媒ドメインはエンド型のキモトリプシンとほぼ同じなのに,エキソ型である理由は説明できなかった.そこで,筆者らはDAP BIIとジペプチドとの共結晶化を試み,ペプチドとの複合体の構造解析に成功した.その結果,アポ体では“パックマン”のように触媒ドメインとヘリカルドメインが開いていたが,ジペプチドとの複合体では閉じて球状になっていた(図2B).また,ジペプチド単位での切断は,基質N末端アミノ酸のアミノ基とカルボキシ基とヘリカルドメインのN330による水素結合と触媒ドメインのN215とN674による水素結合ネットワークとW216によるペプチド基質N末端側のブロックによって担われていた.また,DAP BIIとオクタペプチドとの複合体の構造解析から,長すぎるペプチドではDAP BIIが閉じた構造をとることができず,その結果として長いペプチドは切断されにくいことが示唆された.新規のαヘリカルドメインは,その構造のユニークさという点だけでなく,DAP BIIのエキソ活性とオリゴペプチド認識機構に重要であることが明らかとなった.
S46ファミリーの酵素には,主に疎水性・塩基性アミノ酸を認識するDAP BIIおよびDPP7タイプと,酸性アミノ酸を認識するDPP11タイプという基質特異性のまったく異なる二つのタイプの酵素が存在する12).我々は,DAP BIIの構造解析に続いて,歯周病菌DPP11の構造解析にも成功した.DPP11はDAP BIIとほぼ同じ構造を持っていた(図2A, C, H, I)が,DAP BIIのS1ポケットがD651,I652,G654,A672,G675,S679,M693の主に疎水性のアミノ酸により構成されている(図3A)のに対して,DPP11のS1ポケットはH649,T650,G652,N670,R673,G677,S691と主に中性と塩基性のアミノ酸により構成されていた(図3B).このうち,酸性アミノ酸を認識するのは,R673だと考えられた.さらに,R337がDPP11の基質結合時に相互作用しうる位置に存在していた.基質との複合体の構造が得られなかったこともあり,このR337とR673に関するDPP11変異体6種(R337A, R673A, R337A-R673A, R337N, R673G, R337N-R673G)を作製し,活性測定を行った.その結果,R673Aでは活性が消失したが,R337Aでは,活性が上昇した.つまり,酸性アミノ酸を主に認識する残基はR673であることが示唆された.R337Aでは,活性が上昇することから主に認識する残基ではないものの酸性アミノ酸の認識に影響することが考えられる.この時点でも,DPP11とペプチドとの複合体の構造は得られていなかったので,DAP BIIをDPP11化(G675R)してその複合体の構造を得ることとした.得られたDAP BII(G675R)はDPP11様の活性を持ち,Leu-Gluとの複合体の構造解析に成功した.これまでにDPP11とペプチドとの複合体の構造は得られていないものの,DAP BII(G675R)複合体の立体構造におけるペプチドとの相互作用様式は,DPP11におけるペプチドとの相互作用様式をミミックしていると考えられ,その結果に基づいてS46ファミリーのサブサイトS1における模式図を示した(図3C).
近年,抗菌薬の不適切な使用による短期間での耐性菌の出現が大きな社会問題となっている.製薬会社にとっては,新規抗菌薬の開発コストを回収する前に耐性菌が出現することから,魅力的な市場ではない.そのため,1983~1987年には16もの新規抗菌薬が承認されていたが,2008~2012年にはわずか2と大幅に減少している13).一方,既存抗菌薬の作用機序は主に細胞壁合成阻害,タンパク質合成阻害,DNA合成阻害の三つに分類され,長期的には耐性菌の出現を避けることはできない.そのため,既存の抗菌薬とは異なる作用機序を有する抗菌薬の開発が重要である.WHOは,この危機的状況を打開するために昨年,新規抗菌薬開発に向けた提言を行い,米国では2020年までに新規抗菌薬を10種開発することを目指している13,14).また,国内関連6学会からも新規抗菌薬開発の方向性が示されている15).耐性菌は,広域スペクトルを持つ万能抗菌薬の使用によるとも考えられていることから,今後は,起因菌の特徴に応じた狭域抗菌薬が求められる.S46ファミリーの酵素を標的分子とする阻害薬やプロドラッグは,“S46酵素を持つグラム陰性糖非発酵性病原菌”に対する狭域スペクトルの抗菌薬となると期待している.微生物に特有な分子機構や分子構造を見いだすような基礎研究こそ,アカデミアならではの重要な役目だと考えている.今後,産学官によるオールジャパンでの新規抗菌薬開発が進むことを願ってならない.
謝辞Acknowledgments
本研究の共同研究者である長岡技術科学大学の小笠原渉准教授,鈴木義之博士,昭和大学の田中信忠准教授,宇宙航空研究開発機構の太田和敬主任開発員,山田貢主任開発員に深く感謝いたします.DAP BIIおよびDPP11の高分解能の立体構造は,国際宇宙ステーション「きぼう」における微小重力下結晶化実験により得られた結晶を用いた高エネルギー加速器研究機構PF(2011G090, 2013G138)とSPring-8での実験成果である.また,本研究の一部は科研費(25462872),私立大学戦略的研究基盤形成事業および創薬等支援技術基盤プラットフォーム事業,大阪大学蛋白質研究所共同研究員制度(超分子構造解析学研究室)の支援により実施しました.
引用文献References
1) Bostanci, N. & Belibasakis, G.N. (2012) FEMS Microbiol. Lett., 333, 1–9.
2) Takahashi, N. & Sato, T. (2001) J. Dent. Res., 80, 1425–1429.
3) Ogasawara, W., Kobayashi, G., Okada, H., & Morikawa, Y. (1996) J. Bacteriol., 178, 6288–6295.
4) Banbula, A., Yen, J., Olesky, A., Mak, P., Nugno, M., Travis, J., & Potempa, J. (2001) J. Biol. Chem., 276, 6299–6305.
5) Ohara-Nemoto, Y., Shimoyama, Y., Kimura, S., Kon, A., Haraga, H., Ono, T., & Nemoto, T.K. (2011) J. Biol. Chem., 286, 38115–38127.
6) Nishimata, H., Ohara-Nemoto, Y., Baba, T.T., Hoshino, T., Fujiwara, T., Shimoyama, Y., Kimura, S., & Nemoto, T.K. (2014) PLoS ONE, 9, e114221.
7) Rawlings, N.D., Barrett, A.J., & Bateman, A. (2012) Nucleic Acids Res., 40(D1), D343–D350.
8) Nakajima, Y., Ito, K., Toshima, T., Egawa, T., Zheng, H., Oyama, H., Wu, Y., Takahashi, E., Kyono, K., & Yoshimoto, T. (2008) J. Bacteriol., 190, 7819–7829.
9) Sakamoto, Y., Suzuki, Y., Iizuka, I., Tateoka, C., Roppongi, S., Fujimoto, M., Inaka, K., Tanaka, H., Masaki, M., Ohta, L., Okada, H., Nonaka, T., Morikawa, Y., Nakamura, K.T., Ogasawara, W., & Tanaka, N. (2014) Sci. Rep., 4, 4977.
10) Sakamoto, Y., Suzuki, Y., Iizuka, C., Roppongi, S., Fujimoto, M., Inaka, K., Tanaka, H., Yamada, M., Ohta, K., Gouda, K., Nonaka, T., Ogasawara, W., & Tanaka, N. (2015) Sci. Rep., 5, 11151.
11) Suzuki, Y., Sakamoto, Y., Tanaka, N., Okada, H., Morikawa, Y., & Ogasawara, W. (2014) Sci. Rep., 4, 4292.
12) Rouf, S.M., Ohara-Nemoto, Y., Hoshino, T., Fujiwara, T., Ono, T., & Nemoto, T.K. (2013) Biochimie, 95, 824–832.
13) Infectious Diseases Society of America (2013) Fact Sheet.
14) WHO (2014) Fact Sheet.
15) 新規抗菌薬の開発に向けた6学会提言(2014).
著者紹介Author Profile
 阪本 泰光(さかもと やすみつ)
阪本 泰光(さかもと やすみつ)岩手医科大学薬学部助教.博士(薬学).
略歴1971年熊本に生まる.2002年長岡技術科学大学大学院博士後期課程単位取得退学,同年昭和大学保健医療学部助手,昭和大学診療放射線専門学校講師,07年昭和大学保健医療学部講師,08年岩手医科大学薬学部助手,09年より現職.
研究テーマと抱負現在は,主にジペプチジルアミノペプチダーゼの構造生物学的研究を行っている.一人ではなし得ないことも,様々な研究者,学生,企業,機関等と協力することで可能になると信じています.共同研究募集中です!
ウェブサイトhttp://gaia-sb.iwate-med.ac.jp/pharm/
趣味旅行.
 野中 孝昌(のなか たかまさ)
野中 孝昌(のなか たかまさ)岩手医科大学薬学部教授,日本薬学会理事.博士(工学).
略歴1963年埼玉県に生る.87年東京大学薬学部卒業.89年同大学院薬学系研究科修士課程修了.92年長岡技術科学大学大学院工学研究科博士後期課程修了.92年同大学講師.05年同大学教授.07年より現職.
研究テーマと抱負土の中から見つけてきた薬が億の単位の人々を救い,億の単位の研究費を生み出している.私たちは,X線結晶構造解析を駆使して,シリコンの中から薬を見つけ出すことを目標としている.
ウェブサイトhttp://bit.ly/imu-sb, http://bit.ly/imu-fb
趣味日本一広い岩手県のドライブ.