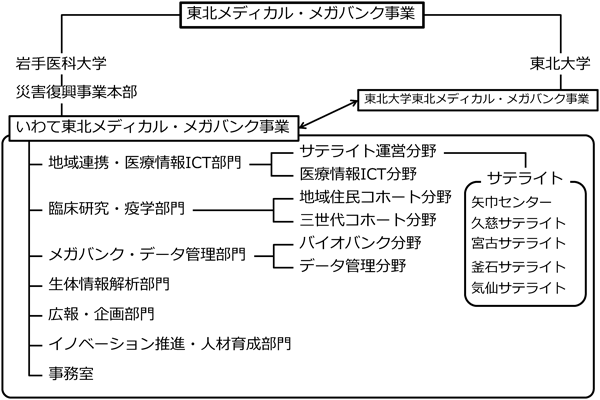
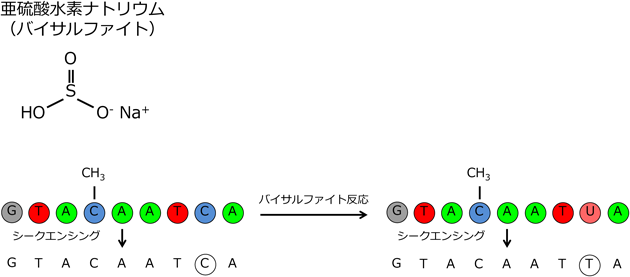
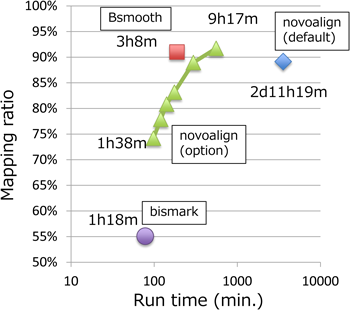
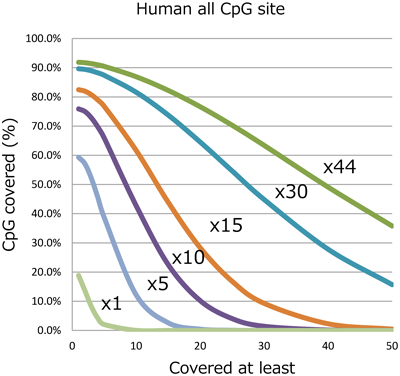
1) Nagasaki, M., Yasuda, J., Katsuoka, F., Nariai, N., Kojima, K., Kawai, Y., Yamaguchi-Kabata, Y., Yokozawa, J., Danjoh, I., Saito, S., Sato, Y., Mimori, T., Tsuda, K., Saito, R., Pan, X., Nishikawa, S., Ito, S., Kuroki, Y., Tanabe, O., Fuse, N., Kuriyama, S., Kiyomoto, H., Hozawa, A., Minegishi, N., Engel, J.D., Kinoshita, K., Kure, S., & Yaegashi, N. (2015) Nat. Commun., 6, 8018.
2) 1000 Genomes Project Consortium, Abecasis, G.R., Altshuler, D., Auton, A., Brooks, L.D., Durbin, R.M., Gibbs, R.A., Hurles, M.E., & McVean, G.A. (2010) Nature, 467, 1061–1073.
3) Sudmant, P.H., Rausch, T., Gardner, E.J., Handsaker, R.E., Abyzov, A., Huddleston, J., Zhang, Y., Ye, K., Jun, G., Hsi-Yang Fritz, M., Konkel, M.K., Malhotra, A., Stütz, A.M., Shi, X., Paolo Casale, F., Chen, J., Hormozdiari, F., Dayama, G., Chen, K., Malig, M., Chaisson, M.J., Walter, K., Meiers, S., Kashin, S., Garrison, E., Auton, A., Lam, H.Y., Jasmine Mu, X., Alkan, C., Antaki, D., Bae, T., Cerveira, E., Chines, P., Chong, Z., Clarke, L., Dal, E., Ding, L., Emery, S., Fan, X., Gujral, M., Kahveci, F., Kidd, J.M., Kong, Y., Lameijer, E.W., McCarthy, S., Flicek, P., Gibbs, R.A., Marth, G., Mason, C.E., Menelaou, A., Muzny, D.M., Nelson, B.J., Noor, A., Parrish, N.F., Pendleton, M., Quitadamo, A., Raeder, B., Schadt, E.E., Romanovitch, M., Schlattl, A., Sebra, R., Shabalin, A.A., Untergasser, A., Walker, J.A., Wang, M., Yu, F., Zhang, C., Zhang, J., Zheng-Bradley, X., Zhou, W., Zichner, T., Sebat, J., Batzer, M.A., McCarroll, S.A., Mills, R.E., Gerstein, M.B., Bashir, A., Stegle, O., Devine, S.E., Lee, C., Eichler, E.E., & Korbel, J.O.; 1000 Genomes Project Consortium (2015) Nature, 526, 75–81.
4) Lappalainen, T., Sammeth, M., Friedländer, M.R., 't Hoen, P.A., Monlong, J., Rivas, M.A., Gonzàlez-Porta, M., Kurbatova, N., Griebel, T., Ferreira, P.G., Barann, M., Wieland, T., Greger, L., van Iterson, M., Almlöf, J., Ribeca, P., Pulyakhina, I., Esser, D., Giger, T., Tikhonov, A., Sultan, M., Bertier, G., MacArthur, D.G., Lek, M., Lizano, E., Buermans, H.P., Padioleau, I., Schwarzmayr, T., Karlberg, O., Ongen, H., Kilpinen, H., Beltran, S., Gut, M., Kahlem, K., Amstislavskiy, V., Stegle, O., Pirinen, M., Montgomery, S.B., Donnelly, P., McCarthy, M.I., Flicek, P., Strom, T.M., Lehrach, H., Schreiber, S., Sudbrak, R., Carracedo, A., Antonarakis, S.E., Häsler, R., Syvänen, A.C., van Ommen, G.J., Brazma, A., Meitinger, T., Rosenstiel, P., Guigó, R., Gut, I.G., Estivill, X., & Dermitzakis, E.T.; Geuvadis Consortium (2013) Nature, 501, 506–511.
7) Gudbjartsson, D.F., Helgason, H., Gudjonsson, S.A., Zink, F., Oddson, A., Gylfason, A., Besenbacher, S., Magnusson, G., Halldorsson, B.V., Hjartarson, E., Sigurdsson, G.T., Stacey, S.N., Frigge, M.L., Holm, H., Saemundsdottir, J., Helgadottir, H.T., Johannsdottir, H., Sigfusson, G., Thorgeirsson, G., Sverrisson, J.T., Gretarsdottir, S., Walters, G.B., Rafnar, T., Thjodleifsson, B., Bjornsson, E.S., Olafsson, S., Thorarinsdottir, H., Steingrimsdottir, T., Gudmundsdottir, T.S., Theodors, A., Jonasson, J.G., Sigurdsson, A., Bjornsdottir, G., Jonsson, J.J., Thorarensen, O., Ludvigsson, P., Gudbjartsson, H., Eyjolfsson, G.I., Sigurdardottir, O., Olafsson, I., Arnar, D.O., Magnusson, O.T., Kong, A., Masson, G., Thorsteinsdottir, U., Helgason, A., Sulem, P., & Stefansson, K. (2015) Nat. Genet., 47, 435–444.
10) Ohmomo, H., Hachiya, T., Shiwa, Y., Furukawa, R., Ono, K., Ito, I., Ishida, Y., Satoh, M., Hitomi, J., Sobue, K., & Shimizu, A. (2014) PLOS ONE, 9, e104283.