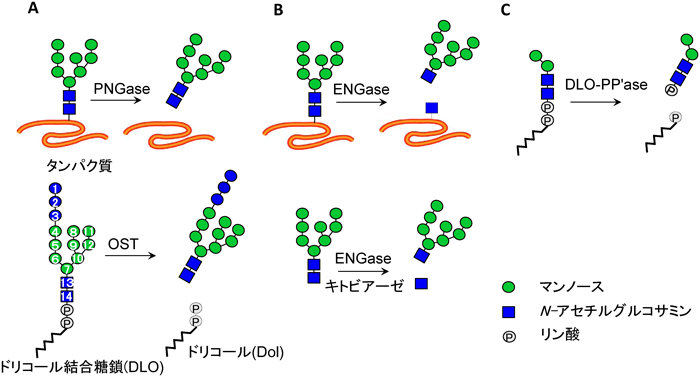
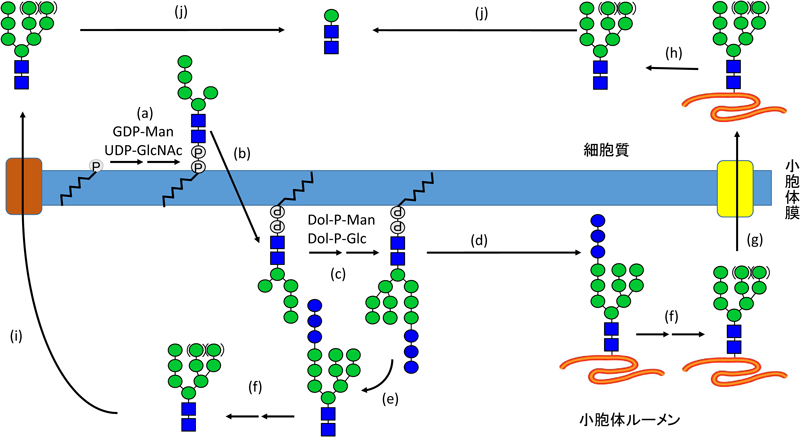
真核細胞の分泌経路を通るタンパク質のほとんどは,小胞体においてN型糖鎖の修飾を受ける.N型糖鎖の付加反応はオリゴ糖転移酵素(oligosaccharyltransferase:OST)と呼ばれる酵素複合体によって担われ,そのドナー基質は出芽酵母,および哺乳動物ではドリコール二リン酸上に合成された14糖(Glc3Man9GlcNAc2-PP-Dol)からなる(図2)3, 4, 20).その14糖はOSTの活性によってタンパク質上のN型糖鎖付加部位(Asn-Xaa-Ser/Thr, Xaa≠Pro)のAsn残基に転移される4, 20).OSTの活性サブユニットはStt3というタンパク質で,哺乳動物ではSTT3AとBという2種類のサブユニットが存在する21).Stt3は真核細胞だけでなく細菌(PglB)や古細菌(AglB)にもオルソログが観察されている22, 23).興味深いことに,真核細胞の中でもTrypanosoma bruceiやLeishmania majorなどの原生動物では,ゲノム上にOSTの他のサブユニットは見いだされず,代わりに複数のSTT3パラログを持つ24–26).
これまで哺乳動物細胞の膜(小胞体)画分からGn2型FNGが生成する現象は広く知られていた27–29).これらのFNGは膜の内腔で生じることが示されており28, 29),その遊離反応はOSTによって行われることが示唆されてきたが29, 30),最近まで実験的な証拠は得られていなかった.
我々は小胞体内のFNGの生成機構を調べるため,まず出芽酵母を用いて実験を行った31).出芽酵母の細胞質PNGase (PNG1)を欠損した酵母はFNGの95%以上が消失するが,ごく微量FNGが生じる32).このFNGの量をOST関連の変異株で測定したところ,そのFNG生成量とOST活性(タンパク質への糖転移活性)には正の相関が観察された31).さらに,OST酵素複合体を精製し,ドナー基質であるGlc3Man9GlcNAc2-PP-Dolと反応させたところ,Glc3Man9GlcNAc2が生成し,OSTの加水分解活性が生化学的に初めて示された31).この加水分解反応は糖転移反応のアクセプター基質(Nα-アセチル-Asn-Tyr-Thr)の存在下では抑制されることから,OSTの糖転移反応と加水分解反応は共通の基質(DLO)に競合的に作用することが示唆された31).
我々は哺乳動物においても小胞体におけるFNG生成反応の解析を行っている.驚いたことに,細胞質における糖タンパク質の脱糖鎖酵素であるPNGase, ENGaseをコードする遺伝子(Ngly1, Engase)の二重破壊株においても細胞内のFNGの総量はほとんど変化がなかった33).このことは,FNGの95%以上が細胞質PNGaseの活性によって作られる,という出芽酵母とは対照的であり,FNGの生成機構は生物種によって大きく異なることがわかる(ちなみに出芽酵母にはENGaseは存在しない34)).細胞膜透過処理をした細胞を用いて,このFNGは小胞体内腔で生じることが示された33).出芽酵母と同様,このFNG生成活性はOSTのアクセプター基質の存在下で抑制されることから,哺乳動物細胞においてもOSTがDol-PP-OSの加水分解反応を担っている可能性が強く示唆された33).
Stt3オルソログの加水分解反応は細菌であるCampylobacter jejuniでも観察されており,OST酵素に広くみられる反応のようである.C. jejuniにおいて,OSTのFNG生成活性は浸透圧の調整に重要な役割を果たしていると考えられている35).興味深いことに,出芽酵母においてOSTの加水分解活性はOST活性の約1/100程度の効率であるのに対し,哺乳動物細胞のOSTの加水分解活性はOST活性の約1/3程度と見積もられる31, 36).このことから哺乳動物細胞においてOSTの加水分解活性が格段に増強されていることがわかる.この生物学的意義は不明だが,これらの活性は細胞内のマンノース6-リン酸(M6P)によって制御されていることが示唆されている30, 37, 38).細胞内のM6P量は小胞体ストレスや単純ヘルペスウイルスの感染によっても上昇する37).GaoとLehrmanは,このM6Pに依存したDol-PP-OSの加水分解活性はウイルス感染に対する防御反応として進化的に獲得されてきたのではないか,という仮説を提唱している38).
ごく最近,細胞質のDNaseの一種であるTREX1タンパク質がOSTと結合することによってOSTの加水分解反応を抑制している,という興味深い結果が示されている39).これらはDNase活性には依存しないTREX1の生理活性であり,OSTと結合できないTREX1の変異体が自己免疫疾患発症の原因となることから,このTREX1によるOSTの活性制御の重要性が示唆されている39).
5. ドリコール結合型糖鎖–ピロホスファターゼによるリン酸化FNGの遊離
DLOからのFNGは,OSTによる加水分解の他,ドリコール結合型糖鎖:ピロホスファターゼ(DLO-PP´ase)によっても生じる27, 51–55).DLO-PP´aseの活性はヒト肝臓54),マウス肝臓56)の他,出芽酵母にも検出されている57).一方で,リン酸化FNGの存在は出芽酵母ではまだ実験的に示されていない.
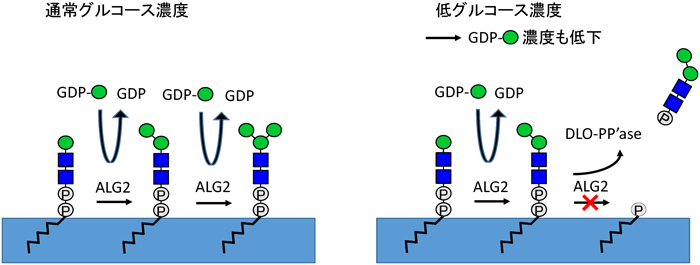
DLO-PP´aseをコードする遺伝子はまだ同定されておらず,その生理機能の詳細は不明である.Pericら,およびVluegelsらはDLO-PP´aseの反応産物であるリン酸化FNGがさまざまな1型先天性糖鎖形成不全症患者由来の細胞で蓄積していることを見いだしている53, 54).一方我々は最近,グルコース飢餓培地で哺乳動物細胞を培養する際,ドリコール糖鎖の生合成の中間体は一過的に生成するものの,ほとんど検出されなくなることを明らかにした55).詳細な解析の結果,そのDLOの消失はDLO-PP´aseの活性によるものであることが判明した55).グルコース飢餓はN型糖鎖の付加反応を低下させる58–63).これらの結果を考え合わせると,グルコース飢餓状態の細胞ではDLO-PP´aseの働きによってDLOの生合成中間体を蓄積させないことで,不完全な糖鎖がタンパク質に転移することを防いでいる可能性が考えられる.すなわち,DLO-PP´aseの反応はDLOの品質管理機構として機能している可能性が示された55).
興味深いことに,グルコース飢餓時に遊離してくる主要なリン酸化FNGはMan2GlcNAc2-Pである.Man2GlcNAc2-DolはALG2 (asparagine-linked glycosylation2)によって合成されるが,本酵素は二つ目と三つ目のManを転移する二重の機能を持ったα-マンノース転移酵素である(図4).このことから,ALG2の二つ目の反応がDLO合成の律速となって,GDP-Manの枯渇状態ではMan2GlcNAc2-Pが蓄積しやすい状態が作り出されているのかもしれない.今後ALG2の酵素学的解析が待たれるところである.
DLOはMan5GlcNAc2までが細胞質側で,その後はルーメン側で合成される(図2).一方,通常検出されるリン酸化FNG(Man0-7GlcNAc2-P)はほぼ細胞質で検出される53–55).細胞質側を向いているDLO (Man0-5GlcNAc2-PP-Dol)とルーメン側を向いているDLO (Man6/7GlcNAc2-PP-Dol)は別々のDLO-PP´aseの作用を受けるのか,またはDLOのフリッピングによって共通のDLO-PP´aseの作用を受けるのかは,今後明らかにされるべき問題である.いずれにしても完全長のリン酸化DLO (Glc3Man9GlcNAc2-P)はさまざまなグリコシダーゼ阻害剤存在下でも観察されないので53, 55),DLO-PP´aseの作用を受けない仕組みが存在すると考えられる.
6. 細胞質におけるFNGの生成:二つの脱N型糖鎖,PNGaseとENGase
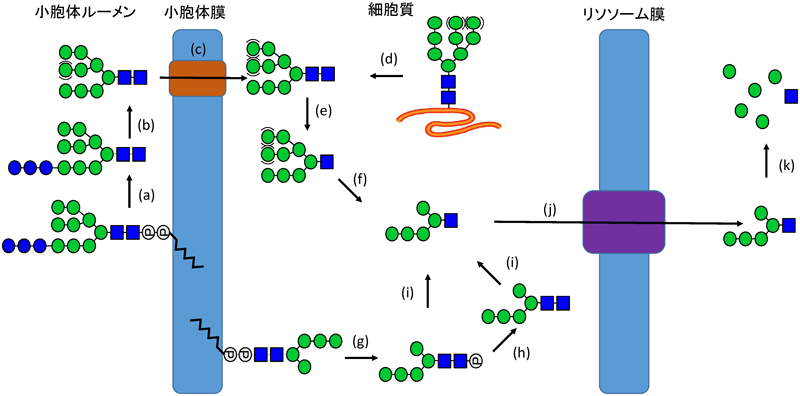
FNGはDLOからだけでなく,糖タンパク質の分解からも生じる.小胞体ではタンパク質のホメオスタシスや品質管理が行われており,機能的なフォールディングをとれないタンパク質は異常タンパク質として小胞体の内腔から細胞質に放出され,その後プロテアソームの活性で分解される.この分解機構は小胞体関連分解(endoplasmic reticulum-associated degradation:ERAD)と呼ばれ,出芽酵母から哺乳動物細胞に至るまで高度に保存されている64, 65).細胞質PNGaseはERADの過程で細胞質に放出された糖タンパク質のN型糖鎖を脱離する酵素である.本酵素の機能の詳細については別稿に譲る8, 11–13, 66–69).
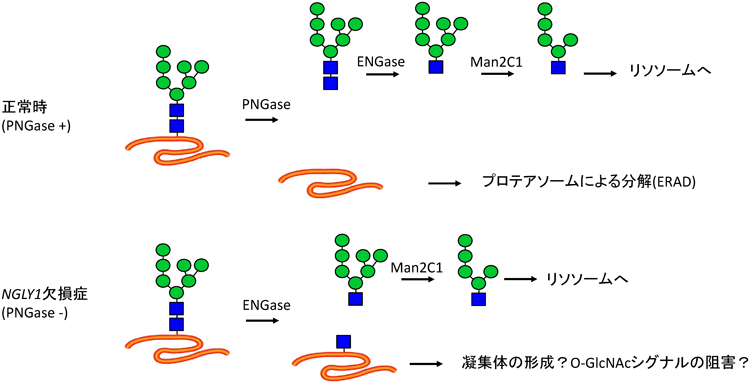
細胞質PNGase/Ngly1のERADにおける重要性は最近までほとんど不明なままであった.なぜなら,出芽酵母においてpng1欠損株はほとんど表現型を示さず,また哺乳動物細胞においてもこれまで調べられた範囲で,発現を抑制したり酵素活性を阻害したりしても,モデルERAD基質の分解の効率にはほとんど影響がなかったからである70–73).一方,最近我々は植物由来の毒素タンパク質,リシンA鎖の無毒化変異体(RTAΔm)を哺乳動物細胞に発現させたところ,Ngly1-ノックアウト(KO)マウス由来の胚繊維芽細胞でその分解が著しく遅延することを見いだした74).興味深いことに,Ngly1-KO細胞においてもRTAΔmのN型糖鎖は脱離されており,その反応は細胞質のもう一つのN型糖鎖脱離酵素,ENGaseによって行われることが判明した74).ENGaseはこれまでGn2型FNGの代謝に関わると考えられてきており,この結果は予想外のものであった.
ENGaseによる糖鎖脱離はタンパク質にGlcNAc 1残基を残す(図5).RTAΔmにおいてはENGaseの反応産物であるN-GlcNAcタンパク質が細胞内凝集体として蓄積し,結果分解が遅れることが明らかとなった74).一方ENGaseとNgly1の両方を欠損した細胞では,驚いたことにRTAΔmの分解は野生型由来の細胞とほぼ同等であった.この結果は細胞質ENGaseが特にNgly1が機能不全の状況で糖タンパク質に直接作用し,N-GlcNAcタンパク質を過剰に生成することがNGLY1欠損症の病態発現に関わる可能性を示唆している.ENGaseの糖タンパク質への反応性は基質によって異なる74).
N-GlcNAcタンパク質はこれまで哺乳動物細胞でもたびたび観察されている75–78).また植物では実際にN-GlcNAcタンパク質が細胞質ENGaseの作用によって生成することが示されている79).したがってN-GlcNAcの生成自体は正常細胞にも起こるであると考えられる.一方で,N-GlcNAcが過剰に細胞質内に生成することで,一部のN-GlcNAcタンパク質が細胞に何らかの悪影響をもたらす可能性は十分に考えられる.たとえば上述のように毒性を持つ凝集体を作る可能性の他,O-GlcNAcと構造上類似することから,O-GlcNAcのシグナル経路に異常をもたらす可能性も提唱されている(図5)74).細胞質や核におけるタンパク質のO-GlcNAc修飾はリン酸化と同様タンパク質の機能や局在性を調節しうる重要な機能を持つ翻訳後修飾である80, 81).
ENGaseは真核細胞に広く存在しているが,出芽酵母や分裂酵母(Schizosaccharomyces pombe)にはその遺伝子オルソログは存在しない34).またO-GlcNAc修飾も出芽酵母には存在しない.最近ヒトのPNGase遺伝子(NGLY1)の変異による遺伝病が発見された69).その症状は全身的な発育不全,運動障害,四肢の筋力低下,肝機能障害,無涙症など実に多岐にわたり,PNGaseの機能の重要性を如実に物語っている.対照的に出芽酵母ではPNG1の欠損株は顕著な表現型を示さない82).哺乳動物においてENGaseの存在,およびO-GlcNAcシグナル経路の存在の有無が出芽酵母とヒトにおけるPNGase欠損の表現型の差として現れている可能性は十分に考えられる.いずれにしても将来ENGaseの特異的な阻害剤が開発できれば,N-GlcNAcタンパク質の生成を抑え,NGLY1欠損症に対する治療薬の有力な候補となることが期待される.
由来がDLOであれ,糖タンパク質であれ,哺乳動物細胞において細胞質に放出されたGn2型FNGはENGaseによってGn1型FNGに変換される33, 83, 84).その後細胞質α-マンノシダーゼであるMan2C1によって特定のα-Man残基(図1の残基番号8, 9, 11, 12)が切断される(図2).Man2C1はFNGの代謝に関わることが示されている85–88).細胞質α-マンノシダーゼはGn2型FNGに比べてGn1型FNGをよい基質とする89, 90).実際Engaseを欠損したマウス細胞で蓄積する主な糖鎖はMan8–9GlcNAc2であり,Man2C1が効率よく作用するためにはENGaseの作用が必要であることが明らかとなった33).
ENGaseとMan2C1によって代謝された細胞質の糖鎖(主にMan5GlcNAc, 図3)はリソソーム膜上に存在する特異的な糖鎖輸送体によってリソソームに取り込まれる91, 92).リソソームに取り込まれたFNGは単糖まで分解される.リソソーム膜上のオリゴ糖輸送体の詳細は不明である.一方,細胞質中のFNGはオートファジーの誘導によっても減少することから,オートファジーはリソソームの輸送体非依存的なFNG代謝に関わりうる93).
ごく最近,我々は哺乳動物細胞においてリン酸化FNGの代謝にはENGaseが関与していることを明らかにした56).Man4GlcNAc2-Pより大きいリン酸化FNGはEngase欠損細胞において蓄積が観察される.このことから,Man0–3GlcNAc2-PはENGaseの基質特異性によりいい基質とならず,α1–2結合のMan(図1の残基番号5)がin vivoにおける効率よいリン酸化FNGへの反応に必須であることが示された56).Man0–3GlcNAc2-Pの代謝機構の詳細は現在のところ不明である.また,リン酸化FNGに働くホスファターゼの存在も示唆されている54).このホスファターゼについての詳細はまったく不明であり,少なくとも我々はこの酵素が存在する積極的な実験的証拠は持ち合わせていない.
前述のように出芽酵母ではENGaseは存在せず,FNGはもっぱらAms1という細胞質–液胞α-マンノシダーゼによって分解される31, 32, 94–97).Ams1による糖鎖分解は,定常期や細胞壁ストレス(細胞壁の合成が阻害される状態)において亢進し31, 32, 95–97),その最終産物はMan1GlcNAc2である31, 32, 94, 96, 97).Ams1は細胞質で合成され,小胞輸送を介さずCvt経路(cytosol-to-vacuole経路)を用いて液胞に運ばれるが,atg19変異株を用いてほとんどのAms1を細胞質に局在させるとFNGの代謝は亢進される31, 32).すなわちAms1は液胞よりも細胞質に存在する方がむしろFNG代謝の効率が上がる,ということになる.したがって,ことN型糖鎖の代謝に限っては,Ams1が液胞にターゲットされる必要性は不明であり,細胞質が主な糖鎖代謝の場所として機能しているようである.
9. 哺乳動物の細胞質にみられる,シアル酸を持つFNG(シアリルFNG)
最近高マンノース型の糖鎖の他に,シアル酸を持つような複合型FNGの存在が哺乳動物で観察されている.たとえば細胞内のシアリルFNGはマウス肝臓125),がん細胞由来培養細胞126),オートファジー欠損細胞93),およびさまざまながん組織で観察されている127–129).これらのほとんどはGn1型の糖鎖であり,リソソーム蓄積病であるシアリドーシス(シアリダーゼ欠損症),あるいはガラクトシアリドーシス(保護タンパク質/カプテシンA欠損症)で観察されるFNGと類似の構造をとっている102–104, 108).蓄積した糖鎖は主に細胞質に蓄積しているようである93, 125, 126).このことから,リソソームから何らかの理由で細胞内に蓄積したシアリルFNGが細胞質に漏出している可能性が考えられる.組織におけるシアリルFNGの存在はがん組織に特異的に観察されたことから,細胞内のシアリルFNGの蓄積は細胞がん化のマーカーになる可能性が提唱されている128, 129).
我々はオートファジー欠損細胞において,リソソーム上のシアル酸輸送体であるシアリンの発現を抑制すると,オートファジーの欠損を誘導した際に細胞質のシアリルFNGの蓄積が遅れることを見いだした93).さらにごく最近,オートファジーの欠損の誘導に伴い,シアリンのタンパク質のレベルが上昇していることが示された130).シアリンの発現上昇がシアリルFNGの増加とどのような関連があるのかは,今後明らかにされるべき問題である.
いずれにしてもこれらの結果を考え合わせると,哺乳動物におけるFNGは図3に示すような高マンノース型糖鎖にとどまらず,シアル酸を持つような複合型のFNGも存在することは明らかである.一方このシアリルFNGを含めた細胞内の複合型FNGの代謝機構はあまりよくわかっていない.オートファジーの誘導によってシアリルFNGの量は減少することから,オートファジーは少なくとも細胞質シアリルFNGの代謝に関わりうる93).
我々は先に細胞質シアリダーゼ(NEU2)を発現することで,シアリルFNGの分解が促進することを明らかにした126).またごく最近,細胞質に存在するグリコシダーゼであるGBA3をNEU2と共発現することによって,細胞内シアリルFNGの代謝が劇的に促進することがわかった131).GBA3は細胞質で働く基質特異性の広いβ-グリコシダーゼであるが,複合型FNGのβ-ガラクトシダーゼとして働く証拠は得られなかった.一方でGBA3はNEU2と物理的に相互作用し,NEU2を安定化させる131).したがって,GBA3は少なくともNEU2を安定化することでシアリルFNGの代謝に関与することが示された.
以上,FNGの生成,プロセシング機構に関わる最新の知見を概観してきた.あらためて明らかにすべきことの多さに圧倒される思いである.しかし謎はこれだけではない.何しろ糖鎖修飾のなかで最もよく研究されているN型糖鎖でさえこの状態である.他の糖鎖(O型糖鎖,GPI,糖脂質等)で非リソソーム型の代謝機構が存在するのかどうかの検討は,まったく手つかずといってよい.ましてやその重要性に思いを馳せることなど,ほとんど暴挙に近いかもしれない.しかし,“何もわかっていない”という状況が私にとってこれほどに魅力的に映るのは,歪んだ性格ゆえであろうか.
昨今は研究のツールが整ってきており,新たなプロジェクトに手を出すことが,容易とまではいかなくとも敷居がだいぶ低くなった,と感じる.ある遺伝子に興味を持ったら,これまでの研究の蓄積がなくとも発現系からKO細胞・マウスまで比較的短期間で手に入れることが不可能ではない時代である.これまで以上に研究の“切り口”が重要になってくるとともに,いわゆる“流行りの研究”に研究費も研究人口も集中しやすい状況が生み出されている,といえるのかもしれない.“大事な研究”に研究を集中すること自体は決して悪いことではないと思うが,反面研究者の間で研究テーマに対する“こだわり”が薄れている危惧を感じるのは私だけではないであろう.
ある会議でうかがった中川真一さん(理化学研究所)の発言が,今でも耳に残っている.“その研究分野を作り上げた人は、絶対ねつ造なんかしないんですよ。たいていねつ造する人は後追いでその分野に参加した人たちです”—私はその発言を詳細に検証する術を持たないが,けだし至言と感じた.確かにその分野を大事に育ててきた研究者は,たとえデータの解釈に対する誤りはありえても,決して恣意的にデータを操作したり不誠実にでっち上げたりはしないであろう.コダワリ,もっといえばそのテーマに対する“愛”が健全な研究の遂行には必須ではなかろうか.
私は幸いなことに,細胞質PNGaseとその機能の研究をこれまで一貫して続けることができた.この研究テーマに注ぐ愛情は人後に落ちない自負がある.とまれ,振り返ってもし“重要な”研究テーマだけが許される世の中であれば,酵素の遺伝子を見つけることも,NGLY1欠損症というヒトの遺伝疾患に出会うこともなかったであろう.なにせN型糖鎖の代謝といえばいまでも“リソソーム”というのが大方の常識であるし,出芽酵母のpng1欠損株はなんら重篤な表現型を示さないのだ.
PNGase/Ngly1が関与する“糖タンパク質の非リソソーム代謝”機構は,まだ遺伝子さえ解明されていない分子が山積,という状態であり,正直なところその“重要性”について云々することも難しい研究分野であることは率直に認めるところである.しかしその不明な分子の中から,“第2,第3のNGLY1”が見つからないとはどうしていえようか? 昨今(応用研究に比べその重要性がわかりにくい)基礎研究をとりまく環境は決してバラ色ではないものの,私自身は今後も“研究者としての知的好奇心”にしっかり軸足を据えて,現在向き合っている疑問に“コダワって”研究を続けていけることを願っている(願えば叶うわけではないが,願わないことには叶わない.世の理である.).
引用文献References
1) Schwarz, F. & Aebi, M. (2011) Curr. Opin. Struct. Biol., 21, 576–582.
2) Varki, A. (1993) Glycobiology, 3, 97–130.
3) Helenius, A. & Aebi, M. (2004) Annu. Rev. Biochem., 73, 1019–1049.
4) Aebi, M. (2013) Biochimica et Biophysica Acta Molecular Cell Research, 1833, 2430–2437.
5) Harada, Y., Hirayama, H., & Suzuki, T. (2015) Cell. Mol. Life Sci., 72, 2509–2533.
6) Aronson, N.N. Jr. & Kuranda, M.J. (1989) FASEB J., 3, 2615–2622.
7) Winchester, B. (2005) Glycobiology, 15, 1R–15R.
8) Suzuki, T. (2009) Trends Glycosci. Glyc., 21, 219–227.
9) Michalski, J.C. (1996) in New Comprehensive Biochemistry (Montreuil, J., Vliegenthart, J.F.G., & Schachtereds, H. eds), Vol. 30, pp. 55–97, Elsevier Science.
10) Suzuki, T., Seko, A., Kitajima, K., Inoue, Y., & Inoue, S. (1993) Biochem. Biophys. Res. Commun., 194, 1124–1130.
11) Suzuki, T. (2003) 生化学,75, 1405–1413.
12) Suzuki, T. (2006) 生化学,78, 1123–1130.
13) Suzuki, T. (2015) J. Biochem., 157, 23–34.
14) Suzuki, T. & Harada, Y. (2014) Biochem. Biophys. Res. Commun., 453, 213–219.
15) Suzuki, T. & Funakoshi, Y. (2006) Glycoconj. J., 23, 291–302.
16) Katoh, T., Ashida, H., & Yamamoto, K. (2009) Trends Glycosci. Glyc., 21, 163–177.
17) Nothaft, H., Liu, X., Li, J.J., & Szymanski, C.M. (2010) Virulence, 1, 546–550.
18) Maeda, M. & Kimura, Y. (2014) Front. Plant Sci., 5, 429.
19) Aronson, N.N. Jr., Backes, M., & Kuranda, M.J. (1989) Arch. Biochem. Biophys., 272, 290–300.
20) Shrimal, S., Cherepanova, N.A., & Gilmore, R. (2015) Semin. Cell Dev. Biol., 41, 71–78.
21) Kelleher, D.J., Karaoglu, D., Mandon, E.C., & Gilmore, R. (2003) Mol. Cell, 12, 101–111.
22) Szymanski, C.M., Yao, R., Ewing, C.P., Trust, T.J., & Guerry, P. (1999) Mol. Microbiol., 32, 1022–1030.
23) Abu-Qarn, M., Yurist-Doutsch, S., Giordano, A., Trauner, A., Morris, H.R., Hitchen, P., Medalia, O., Dell, A., & Eichler, J. (2007) J. Mol. Biol., 374, 1224–1236.
24) Nasab, F.P., Schulz, B.L., Gamarro, F., Parodi, A.J., & Aebi, M. (2008) Mol. Biol. Cell, 19, 3758–3768.
25) Izquierdo, L., Schulz, B.L., Rodrigues, J.A., Guther, M.L., Procter, J.B., Barton, G.J., Aebi, M., & Ferguson, M.A. (2009) EMBO J., 28, 2650–2661.
26) Hese, K., Otto, C., Routier, F.H., & Lehle, L. (2009) Glycobiology, 19, 160–171.
27) Cacan, R., Hoflack, B., & Verbert, A. (1980) Eur. J. Biochem., 106, 473–479.
28) Hanover, J.A. & Lennarz, W.J. (1981) Arch. Biochem. Biophys., 211, 1–19.
29) Anumula, K.R. & Spiro, R.G. (1983) J. Biol. Chem., 258, 15274–15282.
30) Gao, N.G., Shang, J., & Lehrman, M.A. (2005) J. Biol. Chem., 280, 17901–17909.
31) Harada, Y., Buser, R., Ngwa, E.M., Hirayama, H., Aebi, M., & Suzuki, T. (2013) J. Biol. Chem., 288, 32673–32684.
32) Hirayama, H., Seino, J., Kitajima, T., Jigami, Y., & Suzuki, T. (2010) J. Biol. Chem., 285, 12390–12404.
33) Harada, Y., Masahara-Negishi, Y., & Suzuki, T. (2015) Glycobiology, 25, 1196–1205.
34) Suzuki, T., Yano, K., Sugimoto, S., Kitajima, K., Lennarz, W.J., Inoue, S., Inoue, Y., & Emori, Y. (2002) Proc. Natl. Acad. Sci. USA, 99, 9691–9696.
35) Nothaft, H., Liu, X., McNally, D.J., Li, J.J., & Szymanski, C.M. (2009) Proc. Natl. Acad. Sci. USA, 106, 15019–15024.
36) Spiro, M.J. & Spiro, R.G. (1991) J. Biol. Chem., 266, 5311–5317.
37) Gao, N., Shang, J., Huynh, D., Manthati, V.L., Arias, C., Harding, H.P., Kaufman, R.J., Mohr, I., Ron, D., Falck, J.R., & Lehrman, M.A. (2011) Mol. Biol. Cell, 22, 2994–3009.
38) Gao, N. & Lehrman, M.A. (2013) Methods Mol. Biol., 1022, 277–282.
39) Hasan, M., Fermaintt, C.S., Gao, N., Sakai, T., Miyazaki, T., Jiang, S., Li, Q.Z., Atkinson, J.P., Morse, H.C. 3rd, Lehrman, M.A., & Yan, N. (2015) Immunity, 43, 463–474.
40) Caramelo, J.J. & Parodi, A.J. (2008) J. Biol. Chem., 283, 10221–10225.
41) Aebi, M., Bernasconi, R., Clerc, S., & Molinari, M. (2010) Trends Biochem. Sci., 35, 74–82.
42) Hosokawa, N., Kamiya, Y., & Kato, K. (2010) Glycobiology, 20, 651–660.
43) Benyair, R., Ogen-Shtern, N., & Lederkremer, G.Z. (2015) Semin. Cell Dev. Biol., 41, 99–109.
44) Moore, S.E.H. & Spiro, R.G. (1994) J. Biol. Chem., 269, 12715–12721.
45) Moore, S.E.H., Bauvy, C., & Codogno, P. (1995) EMBO J., 14, 6034–6042.
46) Moore, S.E.H. (1998) Glycobiology, 8, 373–381.
47) Haga, Y., Totani, K., Ito, Y., & Suzuki, T. (2009) Glycobiology, 19, 987–994.
48) Moore, S.E.H. & Spiro, R.G. (1990) J. Biol. Chem., 265, 13104–13112.
49) Spiro, M.J., Bhoyroo, V.D., & Spiro, R.G. (1997) J. Biol. Chem., 272, 29356–29363.
50) Durrant, C. & Moore, S.E.H. (2002) Biochem. J., 365, 239–247.
51) Hsu, A.F., Baynes, J.W., & Heath, E.C. (1974) Proc. Natl. Acad. Sci. USA, 71, 2391–2395.
52) Kmiecik, D., Herman, V., Stroop, C.J.M., Michalski, J.C., Mir, A.M., Labiau, O., Verbert, A., & Cacan, R. (1995) Glycobiology, 5, 483–494.
53) Peric, D., Durrant-Arico, C., Delenda, C., Dupre, T., De Lonlay, P., de Baulny, H.O., Pelatan, C., Bader-Meunier, B., Danos, O., Chantret, I., & Moore, S.E.H. (2010) PLoS ONE, 5, e11675.
54) Vleugels, W., Duvet, S., Peanne, R., Mir, A.M., Cacan, R., Michalski, J.C., Matthijs, G., & Foulquier, F. (2011) Biochimie, 93, 823–833.
55) Harada, Y., Nakajima, K., Masahara-Negishi, Y., Freeze, H.H., Angata, T., Taniguchi, N., & Suzuki, T. (2013) Proc. Natl. Acad. Sci. USA, 110, 19366–19371.
56) Harada, Y., Huang, C., Yamaki, S., Dohmae, N., & Suzuki, T. (2016) J. Biol. Chem., in press.10.1074/jbc.M115.685313
57) Belard, M., Cacan, R., & Verbert, A. (1988) Biochem. J., 255, 235–242.
58) Stark, N.J. & Heath, E.C. (1979) Arch. Biochem. Biophys., 192, 599–609.
59) Gershman, H. & Robbins, P.W. (1981) J. Biol. Chem., 256, 7774–7780.
60) Rearick, J.I., Chapman, A., & Kornfeld, S. (1981) J. Biol. Chem., 256, 6255–6261.
61) Turco, S.J. & Pickard, J.L. (1982) J. Biol. Chem., 257, 8674–8679.
62) Baumann, H. & Jahreis, G.P. (1983) J. Biol. Chem., 258, 3942–3949.
63) Doerrler, W.T. & Lehrman, M.A. (1999) Proc. Natl. Acad. Sci. USA, 96, 13050–13055.
64) Needham, P.G. & Brodsky, J.L. (2013) Biochimica et Biophysica Acta Molecular Cell Research, 1833, 2447–2457.
65) Merulla, J., Fasana, E., Solda, T., & Molinari, M. (2013) Traffic, 14, 767–777.
66) Suzuki, T., Park, H., & Lennarz, W.J. (2002) FASEB J., 16, 635–641.
67) Suzuki, T. (2007) Semin. Cell Dev. Biol., 18, 762–769.
68) Hirayama, H., Hosomi, A., & Suzuki, T. (2015) Semin. Cell Dev. Biol., 41, 110–120.
69) Suzuki, T., Huang, C., & Fujihira, H. (2016) Gene, 577, 1–7.
70) Hirsch, C., Blom, D., & Ploegh, H.L. (2003) EMBO J., 22, 1036–1046.
71) Misaghi, S., Pacold, M.E., Blom, D., Ploegh, H.L., & Korbel, G.A. (2004) Chem. Biol., 11, 1677–1687.
72) Blom, D., Hirsch, C., Stern, P., Tortorella, D., & Ploegh, H.L. (2004) EMBO J., 23, 650–658.
73) Kario, E., Tirosh, B., Ploegh, H.L., & Navon, A. (2008) J. Biol. Chem., 283, 244–254.
74) Huang, C., Harada, Y., Hosomi, A., Masahara-Negishi, Y., Seino, J., Fujihira, H., Funakoshi, Y., Suzuki, T., Dohmae, N., & Suzuki, T. (2015) Proc. Natl. Acad. Sci. USA, 112, 1398–1403.
75) Chalkley, R.J., Thalhammer, A., Schoepfer, R., & Burlingame, A.L. (2009) Proc. Natl. Acad. Sci. USA, 106, 8894–8899.
76) Trinidad, J.C., Barkan, D.T., Gulledge, B.F., Thalhammer, A., Sali, A., Schoepfer, R., & Burlingame, A.L. (2012) Mol. Cell. Proteomics, 11, 215–229.
77) Trinidad, J.C., Schoepfer, R., Burlingame, A.L., & Medzihradszky, K.F. (2013) Mol. Cell. Proteomics, 12, 3474–3488.
78) Nagae, M., Morita-Matsumoto, K., Arai, S., Wada, I., Matsumoto, Y., Saito, K., Hashimoto, Y., & Yamaguchi, Y. (2014) Glycobiology, 24, 693–702.
79) Kim, Y.C., Jahren, N., Stone, M.D., Udeshi, N.D., Markowski, T.W., Witthuhn, B.A., Shabanowitz, J., Hunt, D.F., & Olszewski, N.E. (2013) Plant Physiol., 161, 455–464.
80) Hart, G.W. (2014) J. Biol. Chem., 289, 34422–34423.
81) Bond, M.R. & Hanover, J.A. (2015) J. Cell Biol., 208, 869–880.
82) Suzuki, T., Park, H., Hollingsworth, N.M., Sternglanz, R., & Lennarz, W.J. (2000) J. Cell Biol., 149, 1039–1051.
83) Kato, T., Kitamura, K., Maeda, M., Kimura, Y., Katayama, T., Ashida, H., & Yamamoto, K. (2007) J. Biol. Chem., 282, 22080–22088.
84) Chantret, I., Fasseu, M., Zaoui, K., Le Bizec, C., Yaye, H.S., Dupre, T., & Moore, S.E.H. (2010) PLoS ONE, 5, e11734.
85) Suzuki, T., Hara, I., Nakano, M., Shigeta, M., Nakagawa, T., Kondo, A., Funakoshi, Y., & Taniguchi, N. (2006) Biochem. J., 400, 33–41.
86) Kato, A., Wang, L., Ishii, K., Seino, J., Asano, N., & Suzuki, T. (2011) J. Biochem., 149, 415–422.
87) Wang, L. & Suzuki, T. (2013) J. Biol. Chem., 288, 11887–11896.
88) Paciotti, S., Persichetti, E., Klein, K., Tasegian, A., Duvet, S., Hartmann, D., Gieselmann, V., & Beccari, T. (2014) J. Biol. Chem., 289, 9611–9622.
89) Grard, T., Herman, V., SaintPol, A., Kmiecik, D., Labiau, O., Mir, A.M., Alonso, C., Verbert, A., Cacan, R., & Michalski, J.C. (1996) Biochem. J., 316, 787–792.
90) Kumano, M., Omichi, K., & Hase, S. (1996) J. Biochem., 119, 991–997.
91) Saint-Pol, A., Bauvy, C., Codogno, P., & Moore, S.E. (1997) J. Cell Biol., 136, 45–59.
92) Saint-Pol, A., Codogno, P., & Moore, S.E.H. (1999) J. Biol. Chem., 274, 13547–13555.
93) Seino, J., Wang, L., Harada, Y., Huang, C.C., Ishii, K., Mizushima, N., & Suzuki, T. (2013) J. Biol. Chem., 288, 26898–26907.
94) Chantret, I., Frenoy, J.P., & Moore, S.E.H. (2003) Biochem. J., 373, 901–908.
95) Hirayama, H. & Suzuki, T. (2011) Glycobiology, 21, 1341–1348.
96) Chantret, I., Kodali, V.P., Lahmouich, C., Harvey, D.J., & Moore, S.E. (2011) J. Biol. Chem., 286, 41786–41800.
97) Hossain, T.J., Hirayama, H., Harada, Y., & Suzuki, T. (2016) Biosci. Biotechnol. Biochem., 80, 152–157.
98) Tarentino, A.L., Gomez, C.M., & Plummer, T.H. Jr. (1985) Biochemistry, 24, 4665–4671.
99) Suzuki, T., Seko, A., Kitajima, K., Inoue, Y., & Inoue, S. (1994) J. Biol. Chem., 269, 17611–17618.
100) Fan, J.Q. & Lee, Y.C. (1997) J. Biol. Chem., 272, 27058–27064.
101) Kaartinen, V., Mononen, T., Laatikainen, R., & Mononen, I. (1992) J. Biol. Chem., 267, 6855–6858.
102) Strecker, G., Peers, M.C., Michalski, J.C., Hondiassah, T., Fournet, B., Spik, G., Montreuil, J., Farriaux, J.P., Maroteaux, P., & Durand, P. (1977) Eur. J. Biochem., 75, 391–403.
103) Dorland, L., Haverkamp, J., Vliegenthart, J.F.G., Strecker, G., Michalski, J.C., Fournet, B., Spik, G., & Montreuil, J. (1978) Eur. J. Biochem., 87, 323–329.
104) Kuriyama, M., Ariga, T., Ando, S., Suzuki, M., Yamada, T., & Miyatake, T. (1981) J. Biol. Chem., 256, 2316–2321.
105) Abraham, D., Blakemore, W.F., Jolly, R.D., Sidebotham, R., & Winchester, B. (1983) Biochem. J., 215, 573–579.
106) van Pelt, J., Kamerling, J.P., Vliegenthart, J.F., Hoogeveen, A.T., & Galjaard, H. (1988) Clin. Chim. Acta, 174, 325–335.
107) Van Pelt, J., Van Kuik, J.A., Kamerling, J.P., Vliegenthart, J.F., Van Diggelen, O.P., & Galjaard, H. (1988) Eur. J. Biochem., 177, 327–338.
108) van Pelt, J., Hard, K., Kamerling, J.P., Vliegenthart, J.F., Reuser, A.J., & Galjaard, H. (1989) Biol. Chem. Hoppe Seyler, 370, 191–203.
109) Bruggink, C., Poorthuis, B.J., Piraud, M., Froissart, R., Deelder, A.M., & Wuhrer, M. (2010) FEBS J., 277, 2970–2986.
110) Bruggink, C., Poorthuis, B.J., Deelder, A.M., & Wuhrer, M. (2012) Anal. Bioanal. Chem., 403, 1671–1683.
111) Boomkamp, S.D., Rountree, J.S.S., Neville, D.C.A., Dwek, R.A., Fleet, G.W.J., & Butters, T.D. (2010) Glycoconj. J., 27, 297–308.
112) Xia, B.Y., Asif, G., Arthur, L., Pervaiz, M.A., Li, X.L., Liu, R.P., Cummings, R.D., & He, M. (2013) Clin. Chem., 59, 1357–1368.
113) Song, Z., Li, S.C., & Li, Y.T. (1987) Biochem. J., 248, 145–149.
114) Fisher, K.J. & Aronson, N.N. Jr. (1992) J. Biol. Chem., 267, 19607–19616.
115) Park, C., Meng, L., Stanton, L.H., Collins, R.E., Mast, S.W., Yi, X.B., Strachan, H., & Moremen, K.W. (2005) J. Biol. Chem., 280, 37204–37216.
116) Persichetti, E., Klein, K., Paciotti, S., Lecointe, K., Balducci, C., Franken, S., Duvet, S., Matzner, U., Roberti, R., Hartmann, D., Gieselmann, V., & Beccari, T. (2012) BBA Mol. Basis Dis., 1822, 1137–1146.
117) Jones, M.Z. & Laine, R.A. (1980) Fed. Proc., 39, 2082–2082.
118) Daniel, P.F., Warren, C.D., & James, L.F. (1984) Biochem. J., 221, 601–607.
119) Tulsiani, D.R.P., Broquist, H.P., James, L.F., & Touster, O. (1984) Arch. Biochem. Biophys., 232, 76–85.
120) Matsuura, F. & Jones, M.Z. (1985) J. Biol. Chem., 260, 5239–5245.
121) Abraham, D., Daniel, P., Dell, A., Oates, J., Sidebotham, R., & Winchester, B. (1986) Biochem. J., 233, 899–904.
122) Hancock, L.W., Jones, M.Z., & Dawson, G. (1986) Biochem. J., 234, 175–183.
123) Hard, K., Mekking, A., Kamerling, J.P., Dacremont, G.A.A., & Vliegenthart, J.F.G. (1991) Glycoconj. J., 8, 17–28.
124) Jones, M.Z., Rathke, E.J.S., Gage, D.A., Costello, C.E., Murakami, K., Ohta, M., & Matsuura, F. (1992) J. Inherit. Metab. Dis., 15, 57–67.
125) Ohashi, S., Iwai, K., Mega, T., & Hase, S. (1999) J. Biochem., 126, 852–858.
126) Ishizuka, A., Hashimto, Y., Naka, R., Kinoshita, M., Kakehi, K., Seino, J., Funakoshi, Y., Suzuki, T., Kameyama, A., & Narimatsu, H. (2008) Biochem. J., 413, 227–237.
127) Misonou, Y., Shida, K., Korekane, H., Seki, Y., Noura, S., Ohue, M., & Miyamoto, Y. (2009) J. Proteome Res., 8, 2990–3005.
128) Yabu, M., Korekane, H., Hatano, K., Kaneda, Y., Nonomura, N., Sato, C., Kitajima, K., & Miyamoto, Y. (2013) Glycobiology, 23, 634–642.
129) Yabu, M., Korekane, H., Takahashi, H., Ohigashi, H., Ishikawa, O., & Miyamoto, Y. (2013) Glycoconj. J., 30, 247–256.
130) Huang, C., Seino, J., Wang, L., Haga, Y., & Suzuki, T. (2015) Biosci. Biotechnol. Biochem., 79, 553–557.
131) Wang, L., Seino, J., Tomotake, H., Funakoshi, Y., Hirayama, H., & Suzuki, T. (2015) Biomolecules, 5, 1499–1514.
132) Iwatsuka, K., Watanabe, S., Kinoshita, M., Kamisue, K., Yamada, K., Hayakawa, T., Suzuki, T., & Kakehi, K. (2013) J. Chromatogr. B Analyt. Technol. Biomed. Life Sci., 928, 16–21.