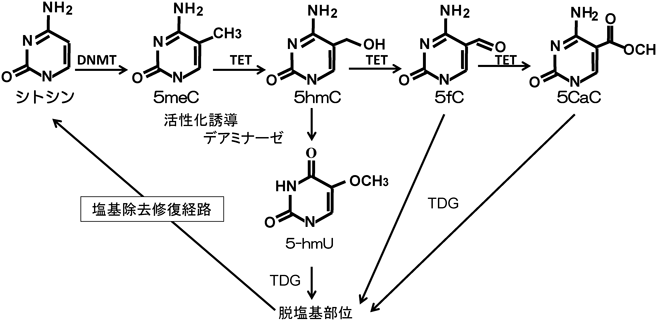
我々の体を構成する細胞は,一つの受精卵が細胞分裂を繰り返すことにより形成されており,同じDNAを持ちながら,異なった形質を発現,維持することができている.明らかにされてきたその仕組みは,DNAのメチル化やヒストンのメチル化,アセチル化,リン酸化やユビキチン化といったDNAの塩基配列によらない,後天的な化学修飾(エピジェネティクス)による遺伝子発現制御機構の存在である.DNAのシトシンの5位のメチル化(5-メチルシトシン:5 mC)は,哺乳類のゲノムDNAに認められる主要な化学修飾である.遺伝子のプロモーター領域やその周辺部に多くみられるCpGアイランドのシトシンがメチル化されると,プロモーターは不活性状態となり転写が抑制される.このシトシンのメチル化は,DNAメチルトランスフェラーゼ(DNA methyltransferase:DNMT)がS-アデノシル-L-メチオニンをメチル基供与体として使用することにより誘導される.一方,5 mCの脱メチル化機構も明らかとなってきた.Ten-eleven-translocation(Tet)と呼ばれる酵素が関わる脱メチル化機構では,まず5meCがTetにより6番目の塩基ともいわれている5-ヒドロキシメチルシトシン(5-hydroxymethylcytosine:5-hmC)に代わり,その後5-ホルミルシトシン(5-formylcytosine:5-fC),5-カルボキシシトシン(5-carboxylcytosine:5-caC)という順に酸化され,最終的に塩基除去修復機構により脱メチル化が完成する(図1).この5 mCはDNAだけではなくRNAにも存在するが,RNAにおいてはDNAと異なり5meC以外に100種類以上の修飾がmRNA, tRNA, rRNA, small nuclear RNA(snRNA),small nucleolar RNA(snoRNA)などの塩基や糖に付加されていることは以前から知られていた.しかしRNAにおけるそれら修飾の生物学的重要性やその制御分子機構に関しては多くが不明なままであった.我々がprostate cancer antigen-1(PCA-1)としてクローニングしていた分子が,このメチル化RNAの脱メチル化を担う酵素としてその後同定された.
2. prostate cancer antigen-1とAlkB
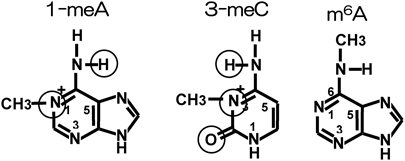
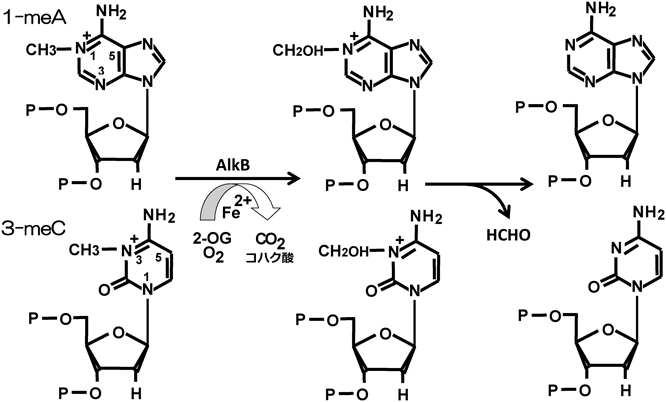
我々は,がんの新たな治療標的分子を探索する目的で,前立腺がん術後検体を用いたディファレンシャル・ディスプレイ解析による網羅的遺伝子の発現解析を行った.その結果,非がん部と比べがん部で発現上昇が認められる遺伝子としてprostate cancer antigen-1(PCA-1)と命名した遺伝子を発見した1).ホモロジー解析により,PCA-1は大腸菌タンパク質AlkBとそのC末端領域においてアミノ酸レベルで23%という高い類似性を示すことが明らかとなった.alkBは他の三つの遺伝子(ada, alkA, aidB)とともに,大腸菌をメチル化剤処理することにより適応応答により発現が誘導される遺伝子である.Adaタンパク質はO6-メチルグアニン-DNAメチルトランスフェラーゼ活性を有し,AlkAは3-メチルアデニン-DNAグリコシラーゼとしてそれぞれDNA損傷修復に関わる.一方,AidBはある種のアルキル化剤を分解する作用が推測されていた.しかしAlkBの機能は明らかにされていなかった.その後,AlkBはメチル化剤で誘導された一本鎖DNAの1-メチルアデニン(1-methyladenine:1-meA),3-メチルシトシン(3-methylcytosine:3-meA)(図2)を脱メチル化するDNA損傷修復酵素であることが示された2).アデニンの1位あるいはシトシンの3位にメチル基が存在すると,Watson-Crick型塩基対形成が障害される.よってこれらのメチル化損傷を修復できなければ細胞死が誘導される.AlkBは3-メチルアデニン-DNAグリコシラーゼが関わる塩基除去修復機構やO6-メチルグアニン-DNAメチルトランスフェラーゼによる直接修復機構とまったく異なる機序によりメチル化塩基を脱メチル化する酵素であった3).この機構は酸化的脱メチル化と呼ばれ,2-オキソグルタル酸(2-oxoglutarate:2-OG)を補助基質,Fe(II)を補因子としてまず1-meAや3-meAをヒドロシメチル化する(図3).この塩基のヒドロキシメチル化体は不安定中間体であり,ホルムアルデヒドを放出することにより安定化状態となる.その結果,脱メチル化が完成されるというものである.一方,1-meAや3-meAはDNAだけではなく,RNAにも存在するが,AlkBはRNAのこれらのメチル化塩基も脱メチル化することも明らかとされた4, 5).これらの知見は,これまでRNAの損傷は修復されずに分解されるという分子生物学のセントラルドグマを塗り変えるもので,RNAにも損傷修復機構が存在するとの考えをもたらすものとなった.
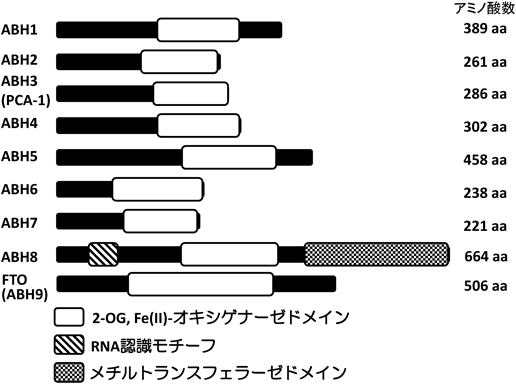
我々がPCA-1を発見したころ,DuncanらもAlkBの2-OG, Fe(II)オキシゲナーゼドメインを有するヒトホモログをクローニングし,すでにAlkBと類似する領域を有するとして遺伝子配列情報のみが報告されていたものをALKBH1,彼らが発見したものをALKBH2と命名し,PCA-1をALKBH3とした6).その後in silicoの解析からヒトにおいてAlkBホモログが少なくても7種類(ALKBH1–ALKBH7)存在する可能性が示された.そこで我々はこれら7種類の遺伝子がヒトにおいて発現しているかを確認するとともに新たなALKBHファミリー分子としてALKBH8をクローニングした7).下記に示すようにALKBH8は他のALKBHファミリー分子と異なる特徴的ドメインを有している.その後,fat mass- and obesity-associated protein(FTO)として研究が進められていた分子も酸化的脱メチル化によりRNA塩基の脱メチル化に関わることが報告され,現在ALKBHファミリー分子はFTOを含め9種が存在する(図4).そこで次にまずALKBH3について述べ,次いでRNAエピジェネティクスに関係するALKBHファミリー分子へと順に特徴を述べる.
1)ALKBH3
ALKBH3は二本鎖DNAよりも一本鎖DNAやRNA中の1-meAや3-meAを効率的に脱メチル化する.核や細胞質に局在することからも細胞内での酵素活性発現と相関している.DangoらはALKBH3による核内での一本鎖DNAの1-meAや3-meAに対する脱メチル化分子機構を解明した.ALKBH3は核内でヘリカーゼであるactivating signal cointegrator complex(ASCC)3と会合して存在している8).メチル化損傷を受けたDNAはASCC3により二本鎖DNAが一本鎖へと巻き戻される.その結果,一本鎖DNAに選択性を有するALKBH3が脱メチル化酵素活性を発現できるようになる.DNAのメチル化損傷が修復されない場合,その損傷が蓄積した細胞は細胞死が誘導される.事実ALKBH3をノックダウンしたがん細胞では細胞死が誘導される.一方,ALKBH3を高発現するがん細胞は,翻訳や複製時に効率的にDNAメチル化損傷が修復されるため生存できると考えられる.これらの知見から,ALKBH3はがんの有望な分子標的となることが推測される.またAlkbh3ノックアウトマウスは顕著な表現型が認められておらず,小児脳腫瘍,前立腺がん,非小細胞肺がん,膵がん9)や泌尿器科がんでALKBH3の発現上昇やALKBH3遺伝子変異が報告されていることからも創薬応用への期待が大きい10).
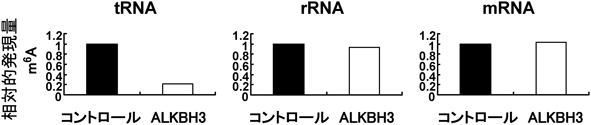
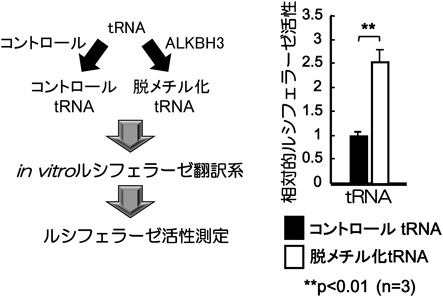
ALKBH3はメチル化DNA塩基を脱メチル化するが,生体内に種々に存在するメチル化RNAに対する作用は不明な点が多く残されている.しかし最近我々は,質量分析装置を用いmRNA, rRNAやtRNAに存在する修飾塩基の検出系を構築し,カイコで作製したリコンビナントALKBH3がそれらRNAに存在する1-meAや3-meAを2-OGとFe(II)依存的に脱メチル化できることを確認した.さらに興味深い点はALKBH3がtRNAのm6Aを効率的に脱メチル化するが,mRNAのm6Aを脱メチル化しないことにある(図5).下記に記載するようにALKBH5やFTO(ALKBH9)はmRNAのm6Aを脱メチル化するとの報告があり,その再現性はカイコリコンビナントALKBH5でも確認した.しかし一方で,カイコリコンビナントALKBH5はtRNAのm6Aを脱メチル化できなかった.これらの結果は,ALKBH3とALKBH5がm6Aの脱メチル化に対して,おそらくRNA高次構造から選択性を発現していると考えられる.またカイコリコンビナントALKBH3で脱メチル化したtRNAはタンパク質の翻訳効率を上昇させることも明らかとしている(図6).これらの結果もALKBH3の発現上昇とがん悪性化との関係をRNA脱メチル化という観点からも示唆させる.
2)ALKBH5
下記のFTO(ALKBH9)に続きmRNAのm6Aを酸化的に脱メチル化する酵素であることが明らかにされたのがALKBH5である11).Alkbh5ノックアウトマウスは減数分裂異常に伴う精子や卵の形成に障害が認められている.また転写因子hypoxia inducible factor 1(HIF-1α)のターゲットとなっていることも明らかとされ,低酸素下での制御機能が推測されている.
3)ALKBH8
他のALKBHファミリータンパク質と同様ALKBH8は,2-OG, Fe(II)オキシゲナーゼドメインを有するとともに,RNA認識モチーフとメチルトランスフェラーゼドメインを有するという特徴を持つ(図4).ALKBH8は細胞質にのみ局在することから,RNAに対する特異的な修飾制御機構が推測されていた.最近,ALKBH8はいくつかのtRNAのアンチコドンループのゆらぎ位置(wobble position)において5-メトキシカルボニルメチルウリジン(5-methoxycarbonylmethyluridine:mcm5U)の生合成の最終段階に関わっていることが報告された12, 13).このtRNAの修飾により,ストップコドンがセレノシステインをコードするコドンとなることから,ALKBH8はセレノプロテインの発現制御に関わっていることが考えられる.事実,Alkbh8ノックアウトマウスでは発生や発育には顕著な表現型は認められていないが,セレノプロテインレベルの低下が明らかとされている.
4)ALKBH9(FTO)
最初にmRNAのm6A脱メチル化酵素活性が報告されたのが,核スペックルに局在性が示されているFTO(ALKBH9)である14).Ftoノックアウトマウスでは成長の遅延や白色脂肪組織の欠損やエネルギー代謝の増加等が認められている.またヒトFTOの多型と肥満や糖尿病との相関が明らかとなっているが,FTOの機能との関係は明確になっていない.
5)ALKBH1
1996年哺乳類で最初に同定されたALKBHである.ALKBH1はDNA/RNAの脱メチル化を担う機能は認められていないが,ヒストンH2Aのメチル化状態を制御して神経発生に機能していることや,酵母のホモログであるOfd2がヒストンH2Aを脱メチル化することなどの報告から,ヒストンタンパク質のメチル化制御との関係が注目されている.Alkbh1ノックマウスマウスは二つのグループから作製報告がなされており15–17),表現型の相違が少しみられるが,初期発生に大きな影響が認められている.
6)ALKBH2
ALKBH2は二本鎖DNAの1-meA, 3-meAの脱メチル化に機能し,Alkbh2ノックアウトマウスはゲノムにおいて1-meAの蓄積が確認されている.しかしそれに伴う明確な表現型は認められていない18).ALKBH2は通常核内に局在し,細胞周期のS期に複製部位でproliferating cell nuclear antigen(PCNA)と結合して存在している.ALKBH2はメチル化DNAの脱メチル化活性を有することから,ALKBH2の発現と抗がん剤として使用されているアルキル化剤に対する抵抗性に相関が考えられる.事実,ヒトグリオーマ細胞におけるALKBH2の高発現は,化学療法剤に対して抵抗性を獲得している.また脳腫瘍や胃がん検体においてALKBH2の変異体の発現や発現上昇が認められている.
7)ALKBH4
Alkbh4ノックアウトマウスは精子形成不全であり,また初期胚致死となる19).またアクチンのモノメチルリシン残基(K84)を脱メチル化する酵素活性を有することも報告された20).細胞質分裂において収縮環はF-アクチンやnon-muscle myosin II(NMII)を含む多くのタンパク質で構成されているが,NMIIは非メチル化アクチンとのみ結合できる.よって,ALKBH4は有糸分裂における収縮環の形成にアクチンの脱メチル化を介して機能していると考えられている.
8)ALKBH6
ALKBHファミリー分子の中で最も解析が遅れているのがALKBH6である.核や細胞質に局在することからDNAやRNAを標的として制御していることも推測されているが,実験的には証明されていない.Alkbh6ノックアウトマウスは作製されているが顕著な表現型の報告はない.ALKBH6は肝がんで顕著な発現が認められており,病理組織学的にはその発現強度と予後不良性の相関もあることから,がん治療標的分子となることも期待できる.
9)ALKBH7
ALKBH7はミトコンドリアシグナルを有し,ミトコンドリアに局在している21).よって代謝やエネルギー産生に関わっていることが推測される.実際,Alkbh7ノックアウトマウスは体重や体脂肪量の増加が認められる22).ALKBH7はプログラムされたネクローシス(ネクロトーシス)にも重要な役割が認められており,アポトーシス抵抗性がんに対する治療標的となる可能性も推測されている.
アデニンのN6位のメチル化であるN6-メチルアデノシン(N6-methyladenosine:m6A)はmRNAにおいて最も高頻度に存在するメチル化修飾塩基である.このm6Aがpoly(A)+ RNAに存在することは,1974年7-メチルグアニンによる5′末端のキャップ構造の研究中に発見された23).しかしRNA塩基配列のどの位置にm6Aが存在するかは不明であった.その後,methylated RNA immunoprecipitation followed by sequence(MeRIP),site-specific cleavage and radioactive labelling followed by ligation-assisted extraction and TLC(ACARLET)やm6A individual-nucleotide-resolution crosslinking and immunoprecipitation(miCLIP)などの技術革新により,ヒト細胞において現在までに約7000のmRNAや非翻訳RNAにm6Aが存在すること,またその修飾のコンセンス配列はRRm6ACH[R=G/A(G>A),H=U/A/C(U>A>G)]であることなどが明らかとなっている.さらにm6AはmRNAの5′非翻訳領域(5′-untranslated region:5′-UTR),3′-UTRやストップコドン周辺24, 25)に集中して存在することから,mRNAの組織特異性発現やマイクロRNA(microRNA:miRNA)との結合制御などUTRの機能に重要な役割を演じていることも考えられている.
RNAの修飾が機能的に重要な役割を担うためには,アデノシンの6位をメチル化するwriter,脱メチル化に機能するeraserとm6Aに結合して修飾mRNAの運命を決定するreaderの存在が必要となる.m6A writerはmethyltransferase like(METTL)3, METTL14とWilm’s tumour-1-associated protein(WTAP)の酵素複合体で構成されていることが示された.この複合体は核スペックルに存在し,METTL3とMETTL14が触媒サブユニットを構成し,WTAPがその酵素活性の制御に関わっている26, 27).RNAのm6Aの生物学的重要性はMettl3変異ES(embryonic stem)細胞により示された.この細胞は分化が抑制されるが,その理由としてm6Aを有さないmRNAは転写と分解のバランスが変わることがあげられている.
RNAのm6Aはその二次構造や三次構造を変えて,readerの結合をもたらす.哺乳類細胞ではこれまでに少なくても五つのm6A結合タンパク質が同定されている.それらはRNA結合YTHドメインを有するYTHDF2とYTHDF3やその後明らかにされたYTHDF1, YTHDC1, YTHDC2である.YTHDF1–YTHDF3は細胞質に存在し,YTHDF1はmRNAの翻訳促進に,YTHDF2は特異的mRNAの不安定化を誘導する.YTHDC1とYTHDC2は核に局在しているが,その核内機能は明らかとされていない.さらに最近,RNAの選択的スプライシングや一次miRNA(pri-miRNA)のプロセシングにおいてRNA結合タンパク質のheterogeneous nuclear ribonucleoproteins(hnRNP)ファミリーメンバーであるHNRNPA2B1がm6A readerとなって制御していることが示された.METTL3によってメチル化されたpri-miRNAのm6AをHNRNPA2B1が認識して結合し,その後DROSHAとともにマイクロプロセッサー複合体を構成するDGCR8が結合することにより,pre-miRNAへとプロセシングされる.
そしてこのm6Aを脱メチル化する酵素が上記のようにFTO(ALKBH9)やALKBH5である.さらにtRNAのm6Aを脱メチル化するALKBH3との関わり合いも注目される.