ヒトをはじめとする哺乳類はレトロウイルスとの共進化の過程でレトロウイルスに対する特異的な感染・増殖抑制因子を獲得してきた.その中の抑制因子の一つとしてAPOBEC3(A3)ファミリーがあげられる.A3遺伝子は,ヒト22番染色体にA3AとA3B, A3C, A3D, A3F, A3G, A3Hの7種がタンデムにコードされ1, 2),ポリヌクレオチドシチジン脱アミノ化酵素に保存された亜鉛配位配列を有する.A3の抗ウイルス作用についてはヒト免疫不全ウイルス(HIV-1)を中心に研究されてきた経緯があり,以下HIV-1を中心に説明する.A3はシチジン脱アミノ化酵素活性依存的あるいは酵素活性非依存的な機序により強力にレトロウイルスの増殖を抑制すると考えられている.酵素活性依存的な作用機序では,A3がHIV-1の逆転写反応中間産物である一本鎖DNA(single-stranded DNA:ssDNA)のデオキシシチジン(deoxycytidine:dC)をデオキシウリジン(deoxyuridine:dU)に変換する.そのためその相補鎖のセンス鎖DNAにデオキシグアノシン(deoxyguanosine:dG)からデオキシアデノシン(deoxyadenosine:dA)への変異が過剰に蓄積し,アミノ酸置換や終止コドンの挿入が引き起こされ,感染性の子孫ウイルスの産生が抑制される.一方,酵素活性非依存的な作用機序では,一本鎖の核酸(DNAあるいはRNA)との結合に高い親和性を持つA3が逆転写酵素の伸長反応を物理的に阻害する3, 4).このような生化学的特性を持つため,通常A3はHIV-1感染細胞から放出されるウイルス粒子に取り込まれ,逆転写反応が起こる新たな感染細胞においてHIV-1の複製を抑制する能力を発揮する.特にA3のうち,A3G,次いでA3Fが最も強い抗HIV-1活性を示す5–10).ヒトA3Hには7種のハプロタイプが人種間で異なって分布し,アフリカ系人種で頻度が高い特殊なハプロタイプIIは,A3Gに匹敵する抗HIV-1活性を示すことがわかっている11–14).
しかしながら,この抗HIV-1作用はHIV-1がコードする遺伝子産物Vif(viral infectivity factor)によって分解不活性化される6, 15).その分解機構は次のとおりである.まずVifがCullin 5(Cul5),Elongin B/C(EloB/C),RING-box protein 2(Rbx2),core-binding factor subunit β(CBF-β)と結合し,ユビキチンリガーゼ(E3)複合体を形成する.Vifはアダプタータンパク質としてA3とも結合して,E3複合体上でA3をリクルートし,ポリユビキチン化,それに続く26Sプロテアソームによる分解へと導く6, 15).これにより感染細胞内ではA3が消失するため放出されるウイルス粒子にはA3が取り込まれず,HIV-1はA3による抑制機構から逃れて新たな感染を繰り返す.
現在,このA3とVifが抗HIV-1薬の新たな創薬ターゲットとして注目を浴びている8, 16).本稿では,A3のVif結合領域(結合インターフェイス)の分子構造およびA3のssDNA結合領域について概説したい.
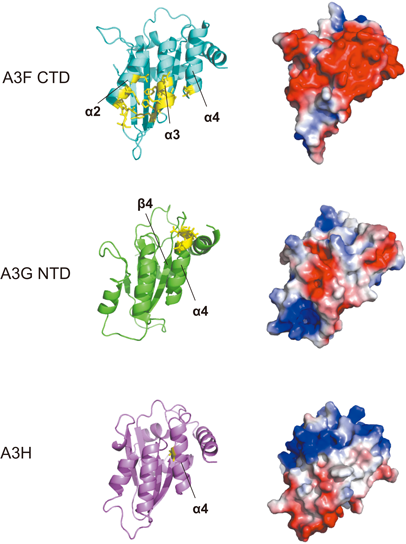
A3は亜鉛イオンを配位する(H/C)XE(X)23–28CXXC配列モチーフからなるZnクラスタードメインを一つ(A3AとA3C, A3H)あるいは二つ(A3BとA3D, A3F, A3G)もつ.遺伝学的に各Znクラスタードメインの配列は,Z1あるいはZ2, Z3の3種に分類される.A3AとA3BのC末端ドメイン(C-terminal domain:CTD),A3G CTDがZ1, A3BのN末端ドメイン(N-terminal domain:NTD)とA3C, A3DとA3Fの両末端ドメイン,A3G NTDがZ2, A3HがZ3にそれぞれ分類される2).各ドメインの立体構造は,保存性が高い五つのβシートと六つのαへリックスからなる基本構造と各ドメインの異なった機能に寄与するループ領域からなる17).二つのZnクラスタードメインからなるA3D, A3F, A3GではCTDが,A3BではNTD, CTD両ドメインが酵素活性中心として機能する18).Znクラスタードメインの立体構造のコアとなる亜鉛配位モチーフ配列は,モノヌクレオチドシチジン脱アミノ化酵素を含むシチジン脱アミノ化酵素スーパーファミリーで高度に保存されている.また,このモチーフをコードするゲノム遺伝子領域は単一エキソン上にあるため,機能的なカセットとしてファミリー遺伝子の増幅が進化学的になされてきたとも考えられる.Znクラスターは二つのシステインとヒスチジンが基本となり,酵素活性中心となる.酵素反応は,配位亜鉛に結合した水分子が,電子供与体となるグルタミン酸側鎖の電子共役によりシチジン残基を求核攻撃することにより起こる19, 20).さらに,A3はポリヌクレオチド型シチジン脱アミノ化酵素活性だけでなく,核酸結合能を有する.結合能は,一本鎖の核酸(DNAあるいはRNA)に特異的かつ協同的である1, 4, 21).
HIV-1がコードする遺伝子産物Vifは,アダプタータンパク質としてCul5-EloB/C-Rbx2 E3ユビキチン複合体にA3をリクルートする.Vifの機能的な立体構造の維持にはCBF-βによる裏打ちが必須である.E3複合体上でポリユビキチン化されたA3は,それに続く26Sプロテアソームによる分解へと導かれる6, 15).この分解はA3とVifの結合の特異性によって規定されており,結合に重要な領域に変異を導入することによってA3の分解が阻害される22–24).これまで,A3タンパク質の立体構造に基づく変異体解析により,Vif-A3の結合には3タイプあり,A3には3種の結合インターフェース(A3C/D/F型,A3G型,A3H型)があると報告されている25, 26)(図1).2012年,我々はVifが結合するドメイン構造としてA3CのX線結晶構造(PDB ID:3VOW)を決定し,A3C/D/F型のVif結合インターフェースはα2およびα3領域に保存された10アミノ酸(A3CにおけるL72, F75, C76, I79, L80, S81, Y86, E106, F107, H111)で構成され,負電荷を帯びた浅い溝を形成していることを同定した27).さらに,A3Dではその溝の近傍にあるE337もVifの結合に重要であることが示唆された27, 28).A3Fはα2およびα3領域にわたるVif結合領域に加えてα4底部に広がる領域の4アミノ酸(L291とA292, R293, E324)も重要で,我々のA3F CTDのX線結晶構造解析(PDB ID:3WUS)からその領域は負に帯電していることが示唆された27–29).一方,A3G型のVif結合インターフェースはA3C/D/F型とはまったく異なる領域が責任領域であることがNMRによる構造解析により報告された30).A3G型のVif結合インターフェースはβ4およびα4間のループ領域にあるDPD(128–130)モチーフが重要であり31),負の表面電荷領域を形成している.またT35, R24がVifとの結合に関与するという報告もある32).一方,Vif分子上のA3結合領域の解析も進んでいる.VifのX線結晶構造33)からVifのA3Fとの結合責任領域はA3Cの責任領域に比べ若干広い正電荷領域が必要であり29),Vif上で重要なA3結合領域も正に帯電している.これらVif中のA3結合領域の静電的特徴は,A3中のVif結合責任領域と符合する.しかし,A3FとA3Gでは結合する領域が異なることが報告されている34, 35).
三つ目のA3H型のVif結合インターフェースは,これまでD121がVifの結合に関与するアミノ酸として唯一同定されている36, 37).このアミノ酸はA3G型のDPD(128–130)モチーフの近傍に位置しているが,A3HのD121付近の配列は他のA3の配列との相同性が低いため,A3C/D/F型やA3G型のVif結合インターフェースとは異なる結合インターフェースが存在することが考えられる.A3G型でVifの結合に重要な負に帯電したDPD(128–130)モチーフはA3Hモデル分子構造上では正に帯電しており,A3C/D/F型でVifの結合に重要な負に帯電した溝はA3Hでも負に帯電しているものの疎水性が低下しており,A3H型Vif結合インターフェースがA3C/D/F型,A3G型とは異なることを示唆する.
以上のことから,A3とVifの結合において3タイプの結合様式が示唆されている.ウイルスの進化の過程で,一つのvif遺伝子産物が複数のA3に対抗し,ウイルスと宿主防御因子A3とのしのぎ合いが非常にダイナミックに行われてきたことをうかがわせる38).おそらく,これらの適応進化はウイルス遺伝子領域の制限と構造学的な制約が深く関与しているであろう.
4. 基質特異性を決定するA3タンパク質の構造学的基盤
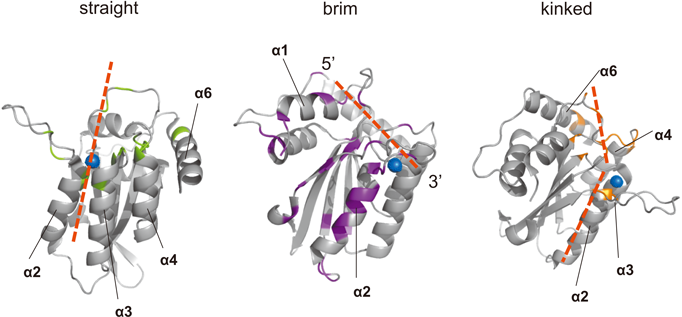
A3による基質特異性は,1)ssDNAが入り込める基質結合領域の溝によるふるい分け効果,および2)脱アミノ化されるdCとその5′側の塩基(5′-TCあるいは5′-CC配列)をA3が認識することによって規定されると考えられている.特に,A3G CTDおよびA3F CTD, A3Aについて,変異体解析やリアルタイムNMR法によって結合領域や結合機序が明らかになってきた.A3G CTDとssDNA基質との相互作用は異なる三つのモデルが提唱されている.brimモデル17)およびstraightモデル39),kinkedモデル40)の三つである.brimモデルでは,loop1, 4, 5領域とα1, α2領域によって形成された溝の縁にある正電荷を帯びた残基(R213およびR215, R256, R320)や芳香環を持つ残基(W285とY315, H367)が基質認識に重要とされ,ssDNAはαへリックスに沿ってN末端側からC末端方向に結合する.straightモデルでは,loop1, 3で形成された溝およびloop5, 7で形成された溝が重要であるとされる.しかしながら,brimモデルとは異なり,ssDNAの結合する方向は不明である.kinkedモデルではloop1, 2, 5, 6領域(R213およびH216, N244, W285, Y315, D316, Q318, R320, R374, R376)が曲がった溝を形成しssDNAと結合する.特にpH 5.5の条件下ではH216がssDNAとの相互作用の活性に重要であるという報告もある41).現在のところ,これら三つのモデルのどれが正しいのか明らかとなっていない.A3F CTDにおけるssDNAの結合領域はloop1, 3, 7で形成された溝であり,六つの芳香族アミノ酸(W277, Y307, Y308, F309, W310, Y314)と電荷を持ったアミノ酸(R305, D313)で形成されている.特に,R305およびW277, Y307はA3Gにおいても保存されており,W277とY307はssDNAとの芳香環どうしのπ–π相互作用に,R305はW277の固定化に重要なアミノ酸である42).一方,A3AのssDNAとの結合モデルはA3G CTDのkinkedモデルに類似している.A3Aのα2, α3, α4領域とloop3, 7領域のアミノ酸(N57, H70, E72, F102, S103, W104, G105, D133, L135, E138)が曲がった溝を形成し,ssDNAと結合することを示す結果が得られている43).また最近の研究により,A3に対する基質の配列特異性を決める責任領域についても明らかとなってきている.A3Gは5′-CC, A3AとA3Fは5′-TC配列を好み44–48),A3G CTDではloop7のD317がその特異性を決定する残基であることが報告されている49).A3Aでは317番目はFであり,D317Y変異型A3Gの基質特異性が5′-CCからA3Aと同じ5′-TCへと変化することが示されている.おそらく,D317がloop7の可動域を制限することによりTよりもCが選択されやすくなっていたためと示唆されている.さらに,A3G CTDのloop7領域のYDDQモチーフをA3F CTDのYYFWモチーフに変換することで,基質特異性がA3F CTDと同じ5′-TCへと変化することも示された42).この場合は,YDDQモチーフのグルタミンやアスパラギン酸が5′-CCのCのアミノ基と水素結合していたため,YYFWモチーフに変換して水素結合をなくしたために基質特異性が変化したと示唆される.
A3とVifの生化学的な研究が進み,これらのX線結晶構造も解かれたことでA3の抗HIV-1活性の分子メカニズムやVifによるA3の分解の分子機序も明らかになりつつある.本稿で取り上げたA3とVifの結合インターフェースは抗HIV-1薬の新しい魅力的な創薬ターゲットである16).創薬を目指し,A3-Vif複合体の構造予測も始まっている29, 38, 50).この相互作用の阻害によりVifによるA3の分解を阻害し,本来ヒトが保持しているA3の抗HIV-1活性を発揮し,HIV-1の増殖を抑制できる低分子化合物の発見が期待されている.今後,Vif-A3タンパク質,あるいはA3と核酸の複合体構造などが解け結合様式が明らかになれば,VifによるA3の分解機序あるいは阻害剤探索,A3による抗ウイルス作用機序の解明が期待できるのではないかと考えられる.