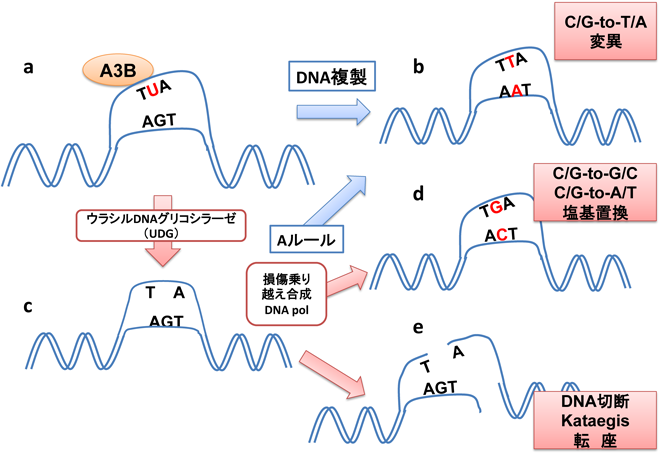
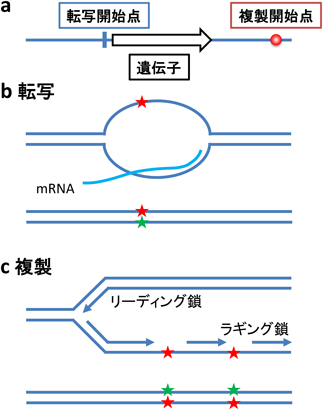
16) Bohn, M.F., Shandilya, S.M., Albin, J.S., Kouno, T., Anderson, B.D., McDougle, R.M., Carpenter, M.A., Rathore, A., Evans, L., Davis, A.N., Zhang, J., Lu, Y., Somasundaran, M., Matsuo, H., Harris, R.S., & Schiffer, C.A. (2013) Structure, 21, 1042–1050.
22) Greenman, C., Stephens, P., Smith, R., Dalgliesh, G.L., Hunter, C., Bignell, G., Davies, H., Teague, J., Butler, A., Stevens, C., Edkins, S., O’Meara, S., Vastrik, I., Schmidt, E.E., Avis, T., Barthorpe, S., Bhamra, G., Buck, G., Choudhury, B., Clements, J., Cole, J., Dicks, E., Forbes, S., Gray, K., Halliday, K., Harrison, R., Hills, K., Hinton, J., Jenkinson, A., Jones, D., Menzies, A., Mironenko, T., Perry, J., Raine, K., Richardson, D., Shepherd, R., Small, A., Tofts, C., Varian, J., Webb, T., West, S., Widaa, S., Yates, A., Cahill, D.P., Louis, D.N., Goldstraw, P., Nicholson, A.G., Brasseur, F., Looijenga, L., Weber, B.L., Chiew, Y.E., DeFazio, A., Greaves, M.F., Green, A.R., Campbell, P., Birney, E., Easton, D.F., Chenevix-Trench, G., Tan, M.H., Khoo, S.K., Teh, B.T., Yuen, S.T., Leung, S.Y., Wooster, R., Futreal, P.A., & Stratton, M.R. (2007) Nature, 446, 153–158.
23) Nik-Zainal, S., Alexandrov, L.B., Wedge, D.C., Van Loo, P., Greenman, C.D., Raine, K., Jones, D., Hinton, J., Marshall, J., Stebbings, L.A., Menzies, A., Martin, S., Leung, K., Chen, L., Leroy, C., Ramakrishna, M., Rance, R., Lau, K.W., Mudie, L.J., Varela, I., McBride, D.J., Bignell, G.R., Cooke, S.L., Shlien, A., Gamble, J., Whitmore, I., Maddison, M., Tarpey, P.S., Davies, H.R., Papaemmanuil, E., Stephens, P.J., McLaren, S., Butler, A.P., Teague, J.W., Jönsson, G., Garber, J.E., Silver, D., Miron, P., Fatima, A., Boyault, S., Langerød, A., Tutt, A., Martens, J.W., Aparicio, S.A., Borg, Å., Salomon, A.V., Thomas, G., Børresen-Dale, A.L., Richardson, A.L., Neuberger, M.S., Futreal, P.A., Campbell, P.J., & Stratton, M.R., Breast Cancer Working Group of the International Cancer Genome Consortium. (2012) Cell, 149, 979–993.
24) Burns, M.B., Lackey, L., Carpenter, M.A., Rathore, A., Land, A.M., Leonard, B., Refsland, E.W., Kotandeniya, D., Tretyakova, N., Nikas, J.B., Yee, D., Temiz, N.A., Donohue, D.E., McDougle, R.M., Brown, W.L., Law, E.K., & Harris, R.S. (2013) Nature, 494, 366–370.
27) Roberts, S.A., Lawrence, M.S., Klimczak, L.J., Grimm, S.A., Fargo, D., Stojanov, P., Kiezun, A., Kryukov, G.V., Carter, S.L., Saksena, G., Harris, S., Shah, R.R., Resnick, M.A., Getz, G., & Gordenin, D.A. (2013) Nat. Genet., 45, 970–976.
28) Alexandrov, L.B., Nik-Zainal, S., Wedge, D.C., Aparicio, S.A., Behjati, S., Biankin, A.V., Bignell, G.R., Bolli, N., Borg, A., Børresen-Dale, A.L., Boyault, S., Burkhardt, B., Butler, A.P., Caldas, C., Davies, H.R., Desmedt, C., Eils, R., Eyfjörd, J.E., Foekens, J.A., Greaves, M., Hosoda, F., Hutter, B., Ilicic, T., Imbeaud, S., Imielinski, M., Jäger, N., Jones, D.T., Jones, D., Knappskog, S., Kool, M., Lakhani, S.R., López-Otín, C., Martin, S., Munshi, N.C., Nakamura, H., Northcott, P.A., Pajic, M., Papaemmanuil, E., Paradiso, A., Pearson, J.V., Puente, X.S., Raine, K., Ramakrishna, M., Richardson, A.L., Richter, J., Rosenstiel, P., Schlesner, M., Schumacher, T.N., Span, P.N., Teague, J.W., Totoki, Y., Tutt, A.N., Valdés-Mas, R., van Buuren, M.M., van’t Veer, L., Vincent-Salomon, A., Waddell, N., Yates, L.R., Zucman-Rossi, J., Futreal, P.A., McDermott, U., Lichter, P., Meyerson, M., Grimmond, S.M., Siebert, R., Campo, E., Shibata, T., Pfister, S.M., Campbell, P.J., Stratton, M.R. Australian Pancreatic Cancer Genome InitiativeICGC Breast Cancer ConsortiumICGC MMML-Seq ConsortiumICGC PedBrain. (2013) Nature, 500, 415–421.
32) Haradhvala, N.J., Polak, P., Stojanov, P., Covington, K.R., Shinbrot, E., Hess, J.M., Rheinbay, E., Kim, J., Maruvka, Y.E., Braunstein, L.Z., Kamburov, A., Hanawalt, P.C., Wheeler, D.A., Koren, A., Lawrence, M.S., & Getz, G. (2016) Cell, 164, 538–549.
39) Maruyama, W., Shirakawa, K., Matsui, H., Matsumoto, T., Yamazaki, H., Sarca, A.D., Kazuma, Y., Kobayashi, M., Shindo, K., Takaori-Kondo, A. (2016) Biochem. Biophy. Res. Commun., in press.
40) Sieuwerts, A.M., Willis, S., Burns, M.B., Look, M.P., Meijer-Van Gelder, M.E., Schlicker, A., Heideman, M.R., Jacobs, H., Wessels, L., Leyland-Jones, B., Gray, K.P., Foekens, J.A., Harris, R.S., & Martens, J.W. (2014) Horm. Cancer, 5, 405–413.
41) Walker, B.A., Wardell, C.P., Murison, A., Boyle, E.M., Begum, D.B., Dahir, N.M., Proszek, P.Z., Melchor, L., Pawlyn, C., Kaiser, M.F., Johnson, D.C., Qiang, Y.W., Jones, J.R., Cairns, D.A., Gregory, W.M., Owen, R.G., Cook, G., Drayson, M.T., Jackson, G.H., Davies, F.E., & Morgan, G.J. (2015) Nat. Commun., 6, 6997.
42) Xu, L., Chang, Y., An, H., Zhu, Y., Yang, Y., & Xu, J. (2015) Urol. Oncol., 33, 340, e341–348.
43) Zhang, J., Wei, W., Jin, H.C., Ying, R.C., Zhu, A.K., & Zhang, F.J. (2015) Int. J. Clin. Exp. Pathol., 8, 5089–5096.
46) Imahashi, M., Izumi, T., Watanabe, D., Imamura, J., Matsuoka, K., Ode, H., Masaoka, T., Sato, K., Kaneko, N., Ichikawa, S., Koyanagi, Y., Takaori-Kondo, A., Utsumi, M., Yokomaku, Y., Shirasaka, T., Sugiura, W., Iwatani, Y., & Naoe, T. (2014) PLoS ONE, 9, e92861.
47) Komatsu, A., Nagasaki, K., Fujimori, M., Amano, J., & Miki, Y. (2008) Int. J. Oncol., 33, 261–270.
48) Nik-Zainal, S., Wedge, D.C., Alexandrov, L.B., Petljak, M., Butler, A.P., Bolli, N., Davies, H.R., Knappskog, S., Martin, S., Papaemmanuil, E., Ramakrishna, M., Shlien, A., Simonic, I., Xue, Y., Tyler-Smith, C., Campbell, P.J., & Stratton, M.R. (2014) Nat. Genet., 46, 487–491.