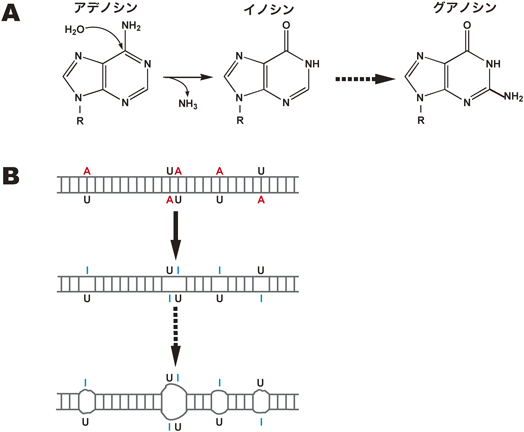
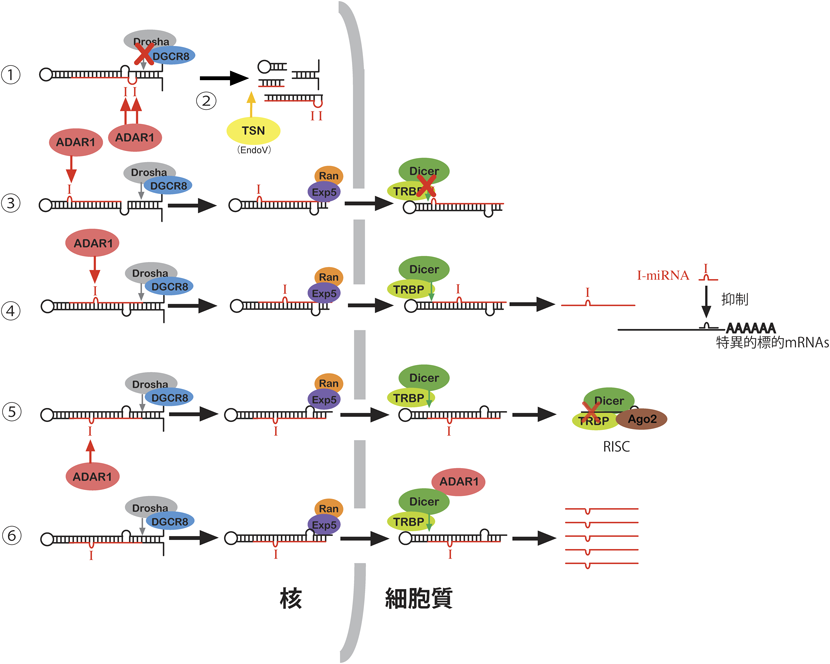
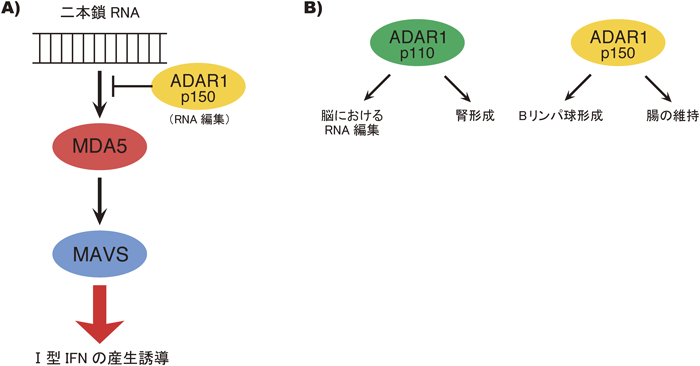
9) Sebastiani, P., Montano, M., Puca, A., Solovieff, N., Kojima, T., Wang, M.C., Melista, E., Meltzer, M., Fischer, S.E., Andersen, S., Hartley, S.H., Sedgewick, A., Arai, Y., Bergman, A., Barzilai, N., Terry, D.F., Riva, A., Anselmi, C.V., Malovini, A., Kitamoto, A., Sawabe, M., Arai, T., Gondo, Y., Steinberg, M.H., Hirose, N., Atzmon, G., Ruvkun, G., Baldwin, C.T., & Perls, T.T. (2009) PLoS ONE, 4, e8210.
16) Iizasa, H., Wulff, B.E., Alla, N.R., Maragkakis, M., Megraw, M., Hatzigeorgiou, A., Iwakiri, D., Takada, K., Wiedmer, A., Showe, L., Lieberman, P., & Nishikura, K. (2010) J. Biol. Chem., 285, 33358–33370.
19) Salameh, A., Lee, A.K., Cardo-Vila, M., Nunes, D.N., Efstathiou, E., Staquicini, F.I., Dobroff, A.S., Marchio, S., Navone, N.M., Hosoya, H., Lauer, R.C., Wen, S., Salmeron, C.C., Hoang, A., Newsham, I., Lima, L.A., Carraro, D.M., Oliviero, S., Kolonin, M.G., Sidman, R.L., Do, K.A., Troncoso, P., Logothetis, C.J., Brentani, R.R., Calin, G.A., Cavenee, W.K., Dias-Neto, E., Pasqualini, R., & Arap, W. (2015) Proc. Natl. Acad. Sci. USA, 112, 8403–8408.
23) Rice, G.I., Kasher, P.R., Forte, G.M., Mannion, N.M., Greenwood, S.M., Szynkiewicz, M., Dickerson, J.E., Bhaskar, S.S., Zampini, M., Briggs, T.A., Jenkinson, E.M., Bacino, C.A., Battini, R., Bertini, E., Brogan, P.A., Brueton, L.A., Carpanelli, M., De Laet, C., de Lonlay, P., del Toro, M., Desguerre, I., Fazzi, E., Garcia-Cazorla, A., Heiberg, A., Kawaguchi, M., Kumar, R., Lin, J.P., Lourenco, C.M., Male, A.M., Marques, W. Jr., Mignot, C., Olivieri, I., Orcesi, S., Prabhakar, P., Rasmussen, M., Robinson, R.A., Rozenberg, F., Schmidt, J.L., Steindl, K., Tan, T.Y., van der Merwe, W.G., Vanderver, A., Vassallo, G., Wakeling, E.L., Wassmer, E., Whittaker, E., Livingston, J.H., Lebon, P., Suzuki, T., McLaughlin, P.J., Keegan, L.P., O’Connell, M.A., Lovell, S.C., & Crow, Y.J. (2012) Nat. Genet., 44, 1243–1248.
24) Kono, M., Matsumoto, F., Suzuki, Y., Suganuma, M., Saitsu, H., Ito, Y., Fujiwara, S., Moriwaki, S., Matsumoto, K., Matsumoto, N., Tomita, Y., Sugiura, K., & Akiyama, M. (2016) J. Invest. Dermatol., 136, 875–878.
25) Mannion, N.M., Greenwood, S.M., Young, R., Cox, S., Brindle, J., Read, D., Nellaker, C., Vesely, C., Ponting, C.P., McLaughlin, P.J., Jantsch, M.F., Dorin, J., Adams, I.R., Scadden, A.D., Ohman, M., Keegan, L.P., & O'Connell, M.A. (2014) Cell Reports, 9, 1482–1494.
30) Chen, L., Li, Y., Lin, C.H., Chan, T.H., Chow, R.K., Song, Y., Liu, M., Yuan, Y.F., Fu, L., Kong, K.L., Qi, L., Li, Y., Zhang, N., Tong, A.H., Kwong, D.L., Man, K., Lo, C.M., Lok, S., Tenen, D.G., & Guan, X.Y. (2013) Nat. Med., 19, 209–216.
31) Jiang, Q., Crews, L.A., Barrett, C.L., Chun, H.J., Court, A.C., Isquith, J.M., Zipeto, M.A., Goff, D.J., Minden, M., Sadarangani, A., Rusert, J.M., Dao, K.H., Morris, S.R., Goldstein, L.S., Marra, M.A., Frazer, K.A., & Jamieson, C.H. (2013) Proc. Natl. Acad. Sci. USA, 110, 1041–1046.
32) Fumagalli, D., Gacquer, D., Rothe, F., Lefort, A., Libert, F., Brown, D., Kheddoumi, N., Shlien, A., Konopka, T., Salgado, R., Larsimont, D., Polyak, K., Willard-Gallo, K., Desmedt, C., Piccart, M., Abramowicz, M., Campbell, P.J., Sotiriou, C., & Detours, V. (2015) Cell Reports, 13, 277–289.
34) Han, L., Diao, L., Yu, S., Xu, X., Li, J., Zhang, R., Yang, Y., Werner, H.M., Eterovic, A.K., Yuan, Y., Li, J., Nair, N., Minelli, R., Tsang, Y.H., Cheung, L.W., Jeong, K.J., Roszik, J., Ju, Z., Woodman, S.E., Lu, Y., Scott, K.L., Li, J.B., Mills, G.B., & Liang, H. (2015) Cancer Cell, 28, 515–528.