古今東西を問わず天才外科医の話が大人気ではあるが,どんな病気でも薬を飲むだけで痛くも苦しくもなく完治すればそれにこしたことはない.いくつかの疾患についてカルパインの阻害剤はそのような夢の治療薬になりうるかもしれない.
カルパインは主に細胞質内で機能するCa2+要求性のシステインプロテアーゼである.以前は,プロテアソームやカスパーゼ,さらにはオートファジーよりも知名度は高かったのだが,現状は逆転している.その理由は,カルパインの生理機能を明示的に(たとえば100字で)記述することができないという,よくいえば懐の広さ,悪くいえば茫漠としたファジー性にあるのだろうか.何事にも短期間での明確な成果が要求される昨今,かなり腰を据えて研究しても「〇〇のときに,△△を切断することにより,□□を引き起こす,それが生理機能だ」と,なかなかいい切れないのがカルパインである.
しかし,見方を変えればまさにダイヤの原石(たとえば,夢の治療薬への手がかり)がごろごろ転がっているわけで,一部には,密かに,原石を全部拾うまで,カルパインのメジャーデビューは控えさせたいという声もある.そのためこのような総説はもしかしたら営業妨害になるかもしれないが,せっかくなので原石の“鑑定過程”を紹介させていただきたい.なお紙面の都合上,阻害剤などの分子構造や治験などの情報については割愛したので,これらについては最近の英文総説1)を参照していただけると幸いである.
カルパインは1964年(論文投稿は1963年7月)に初めて報告された2).ちなみに,C. de Duve博士が初めてオートファジーという言葉を使ったのは1963年だが,大きく注目され始めたのは2016年ノーベル生理学・医学賞に輝かれた大隅良典博士が酵母で分子機構を発見した1990年代以降である.また,プロテアソームとカスパーゼがどちらも1980年代に発見されたことを考えると,カルパインは比較的長い研究歴を持つ細胞内プロテアーゼということができる.ただし,鈴木紘一博士によって「カルパイン」という名称に統一されたのは1991年で,それまではカルシウム依存性中性プロテアーゼ(Ca2+-activated neutral protease:CANP)の他,カルシウム依存性プロテアーゼ(Ca2+-dependent protease:CDP),キナーゼ活性化因子(kinase-activating factor:KAF),カルシウム依存性筋細胞質因子(Ca2+-activated sarcoplasmic factor:CASF)などと各々のカルパイン研究者が呼称していた(よくある話ではあるが).
「カルパイン」という名称は村地孝博士によって1981年に考案されたもの3)である.ポスター発表前夜の丑三つ時にトロントのホテルで“calpastatin”(カルパインの内在性特異的阻害タンパク質:後述)をまず思いつき,そこから派生して“calpase”(当初はcalpainではなかった)を創語し,ポスターにわざわざ大きく貼り出したという興味深いいきさつが村地博士自身により記されている4).しかし,なぜ10年近くも本邦で,そして全世界的に二つの名称,カルパインとCANPとが共存していたかについては,鈴木博士による含蓄に富む総説5)をぜひご参照いただきたい.ポイントは,まず,今堀和友博士らが提唱したCANPという名称で,1977年に鈴木博士が「CANPは阻害剤に対する挙動からシステインプロテアーゼと考えられる」と発表したところ,村地博士から「活性中心にSH基の存在を確かめるまでは阻害剤の結果だけからシステインプロテアーゼと呼ぶべきではない」とすぐにその場で反論され,しかしもっともと納得した.当時,活性中心の同定には約100 mg(鶏胸肉50~60枚からの精製量に匹敵する)の酵素が必要であり,実際に同定されたのは1983年のことであった6).ところが,村地博士の提唱した「カルパイン」はCa2+の「カル」とパパインなどのシステインプロテアーゼを表す“ain”との造語であり4),システインプロテアーゼと呼ぶべきでないといっていた本人が,1981年の時点で「カルパイン」の名を作り出したことに,今堀博士のグループから派生した研究者たちは筋が通らないと考えたわけであった.その後,1990年6月のゴードン会議で名称の統一を議論し,同年10月の第8回細胞内タンパク質分解に関する国際会議(The 8th International Conference on Proteolysis and Protein Turnover:ICOP)で,in vivoでの活性化にそれぞれµM, mMレベルのCa2+を必要とする2種の酵素として,μ-カルパイン,m-カルパインという名称が関係者に正式に周知された.大変残念なことにゴードン会議の直前に村地博士は急逝され,鈴木博士と今堀博士は2010年4月と2016年5月にそれぞれ逝去された.今では天国でカルパインについて再び激論を交わされていることであろう.
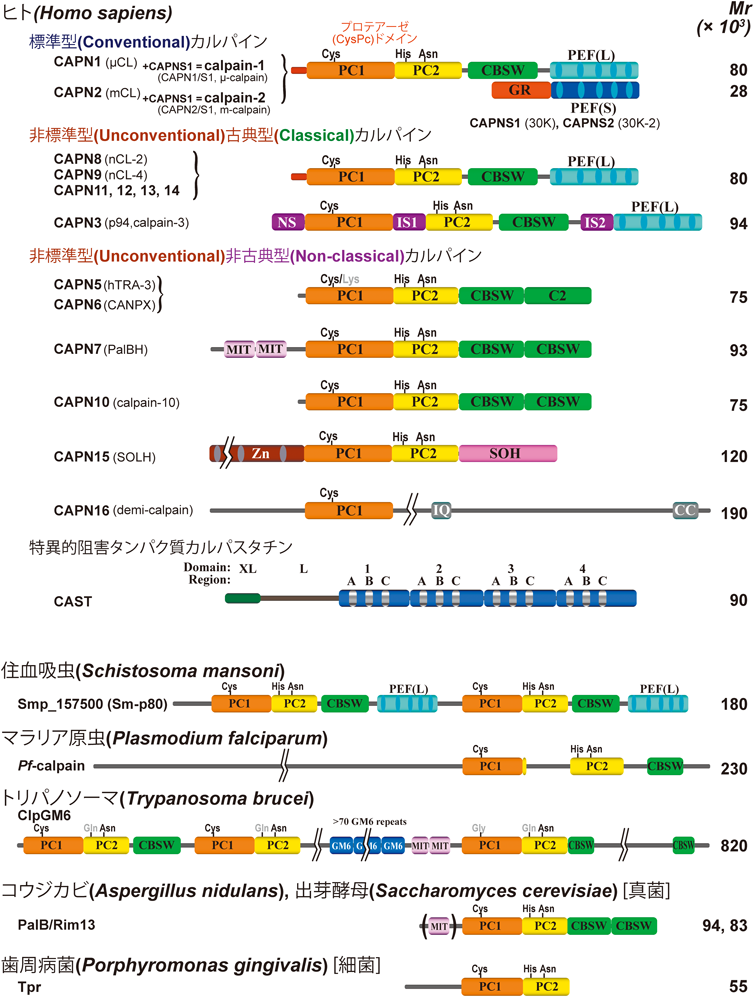
その後もカルパインの分子種名やドメインの名称でしばらく混乱があったが,今はおさまりつつある(と筆者は信じている).誰でも,自分たちが最初に見つけて命名した名称を,後から相談もなく勝手に変えられては,気分がよくない(だろう).特に若いときは(きっと).カナダのPeter L. Davies博士のグループ7)と,鈴木博士らとドイツのWolfram Bode博士との共同グループ8)とが決定したカルパインの立体構造に基づき,2013年のアメリカ実験生物学会研究カンファレンス(Federation of American Societies for Experimental Biology Science Research Conference:FASEB SRC)において出席者全員のコンセンサスを得た命名法を図1に示す.何とかこれを浸透させるべく,本稿もこの命名に従う.本音をいうと,現在のカルパイン関係の呼称(カルパイン-1~16やドメイン名)は,欧米勢に押し切られたような感じで,ちょっと残念ではある.しかし,共通の言葉を使用するという科学の発展の基本精神に則って,一日も早くカルパインを研究する者全員が共通の用語を用いるようになってほしいと切望している.
もう一点大事なこととして,カルパイン(CAPN)という分子を定義しておきたい.これも研究者によって微妙に異なるが,ここでは単純に,ヒトのカルパイン-1(μ-カルパイン,μCANP,μ80K,カルパインI,CDP Iなどと呼ばれていた)の触媒ドメインに有意に相同性のある配列領域を有する遺伝子産物,とする.このドメインは,アメリカNCBI (National Center for Biotechnology Information)のCDD (Conserved Domain Database)に“CysPc”(何の略かはどこにも記載がないが,Cysteine Protease domain, calpain-typeといった感じか)として登録されているモチーフである.
また,酵素としての活性を問題にしていないことに注意してほしい.実は,カルパインの中には活性を持たないと考えられる分子種や,酵素としてはサブユニット構造をとるものも存在する.「カルパイン」という名称は,集合的には上記で定義される遺伝子産物を示し,しかしながら,個別にカルパイン-1,-2のように使う場合は,生体内での本来の構造を持つもの(=活性のあるものならintactな酵素)を意味する.一方,サブユニットや本来の構造が不明なものについては遺伝子名を用いた遺伝子産物としてCAPN1, CAPN2のように呼ぶ.ややこしくて申しわけないが,カルパイン-1=CAPN1+CAPNS1 (CAPN1/S1とも表記),カルパイン-2=CAPN2+CAPNS1 (CAPN2/S1)となる(どちらもヘテロ二量体で,CAPNS1は,両者に共通の制御サブユニット,図1).
哺乳類のカルパイン-1,-2および鳥類のカルパイン-11(当初はカルパイン-1と-2の中間型と考えられていた)は,カルパイン研究の黎明期に最もよく解析されたため,conventional(標準型)カルパインと呼ばれる.それ以外はすべてunconventional(非標準型)カルパインとなる(図1).以下で論ずる構造は標準型を基準として,同様のドメイン構造を有するカルパインをclassical(古典型)カルパインと呼ぶ.これらは歴史的経緯による命名なので,実際には,古典型カルパインは動物にしか見いだされず,最も普遍的に存在しているカルパインはnon-classical(非古典型)カルパインのCAPN7である(後述).以前は,プロテインキナーゼCに倣って,古典型を“typical”,非古典型を“atypical”と呼んでいたが,atypicalの方が普遍的に存在するのはおかしいということで,classical/non-classicalに変更した.
古典型カルパインは,N末端αヘリックス,CysPcドメイン[PC1およびPC2 (protease core domain 1および2)の二つからなる],CBSW (calpain-type β-sandwich)ドメイン,PEF(penta-EF-hand)ドメインという構造を持つ(図1).カルパイン-1および-2はヘテロ二量体であり,CAPNS1という非カルパイン遺伝子産物をサブユニットに持つ.CAPNS1は,N末端にグリシンのクラスタ領域(Gly-rich:GRドメイン)を,後半にはPEFドメインを持ち,活性サブユニットの安定化に寄与する.また,胃に遍在するカルパイン-8/9 (CAPN8/9, G-カルパインとも呼ぶ)はCAPN8とCAPN9のヘテロ二量体である.これ以外のカルパインは四次構造が不明なので,どれも遺伝子産物として表記することが望ましい.たとえば,カルパイン-3やカルパイン-10は,CAPN3やCAPN10を活性サブユニットに持つ仮想的酵素であり,その実体はまだ不明確である.そのため,本来はCAPN3, CAPN10と表記すべきである.カルパイン-3に関しては,in vitroではCAPN3のホモ二量体の可能性が示唆されているが,in vivoでは未確認である.さらに,in vivoではコネクチン(タイチンとも呼ばれる)との結合状態で存在し,少なくともin vitroではPLEIAD(platform element for inhibition of autolytic degradation)9)と結合して安定化するなど,酵素としてのカルパイン-3の実体解明にはさらなる解析が必要である.
ヒトには15のカルパイン遺伝子が存在し,複数の選択的スプライス転写産物を持つ遺伝子もあるため,50以上のカルパイン分子種が存在する.ヒトの非古典型カルパインにはCysPcのうち活性中心のCys残基やPC2を失っているものも存在する(図1).全生物的には,カルパインはほぼすべての真核生物と一部の原核生物に存在する.そして,CysPcドメインに組み合わされるモチーフの種類は40を超え,その中にはMIT(microtubule interacting and transport)モチーフ,Znフィンガーモチーフ,ユビキチン結合(ubiquitin-associated:UBA)ドメインなども存在する10).
一方,カルパインの進化を考えるにあたって,不可解な事例がいくつかある.その最たるものとして,同じ真菌の中でも,出芽酵母(Saccharomyces cerevisiae)やコウジカビ(Aspergillus nidulans)にはCAPN7ホモログであるRim13/PalBが存在するのに,分裂酵母(Schizosaccharomyces属;S. cryophilus, S. octosporus, S. pombeおよびS. ponicus)には,カルパイン遺伝子が存在しないことがあげられる.Rim13/PalBはS. cerevisiaeやA. nidulansが生育環境のアルカリ側への変動に適合して生存するために必要なシグナル伝達系の構成成分として,転写調節因子Rim101/PacCをプロセシング・活性化することが知られている(表1)11).
表1 酵母Rim101経路関連分子と,他生物でのオルソログ| 機能 | S. pombe | S. cerevisiae | A. nidulans | H. sapiens | 特徴 |
|---|
| 細胞外シグナルを検出 | (α-arrestins)*1 | Rim8 | PalF | (α-arrestins)*1 | α-arrestin様タンパク質 |
| SPCC1919.12c*2 | Rim21/Dfg16 | PalH | (7TM)*1 | 7回膜貫通タンパク質 |
| SPCC1739.10 | Rim9 | PalI | —*3 | SUR7/PalIモチーフを持つ3回膜貫通タンパク質 |
| 膜輸送系 | Vps32 | Snf7 | AN4240.2 | CHMP4A | ESCRT-III構成成分 |
| 転写調節因子のプロセシング・活性化 | SPAC2G11.05c | Rim20 | PalA | ALIX | BRO-1ドメインを持つ足場タンパク質 |
| —*3 | Rim13 | PalB | CAPN7 | プロテアーゼ |
| —*3 | Ygr122w | PalC | —*3 | BRO-1ドメインタンパク質 |
| アルカリ環境適応遺伝子群の転写 | (Zn-finger TFs)*1 | Rim101 | PacC | (Zn-finger TFs)*1 | C2H2 Znフィンガー転写調節因子,プロセシングを受けて活性化する |
ESCRT:endosomal sorting complex required for transport, TF:転写調節因子,TM:膜貫通.
* 1類似構造の分子が複数あり,対応する分子が特定されていない.
* 2メタロペプチダーゼドメインを含む.
* 3対応する分子が存在しない. |
これらのシグナル伝達系を構成する分子の,少なくとも一部については,分裂酵母,さらにはヒトにもオルソログが存在している(表1).S. cerevisiaeやA. nidulansと同様のpHを感知するシグナル伝達系をヒトが有するとは考えにくいが,分裂酵母が備えている可能性はあるだろう.また,pH変化以外の要因に対応するための伝達系をこれらオルソログが構成している可能性もある.いずれにしても,①Rim13/PalBの活性化には,Rim20/PalA(分子N末端にBRO-1ドメインという構造を持つ)との複合体形成,およびそれを介したESCRT (endosomal sorting complex required for transport)膜輸送系との相互作用が必要であること,②ESCRT系は分裂酵母からヒトに至るまで保存されていること,③ヒトCAPN7に関してもBRO-1ドメインタンパク質であるALIXとの相互作用が報告されている12)ことから,ヒトでも部分的に変更されながらも保存されたシグナル伝達系として重要な機能を担っている可能性が高い.一方で,出芽酵母と同様の生活環境変化への対応が必要と考えられる分裂酵母が,なぜ,カルパインなしで生きていけるのか,ぜひその仕組みを知りたいものである.
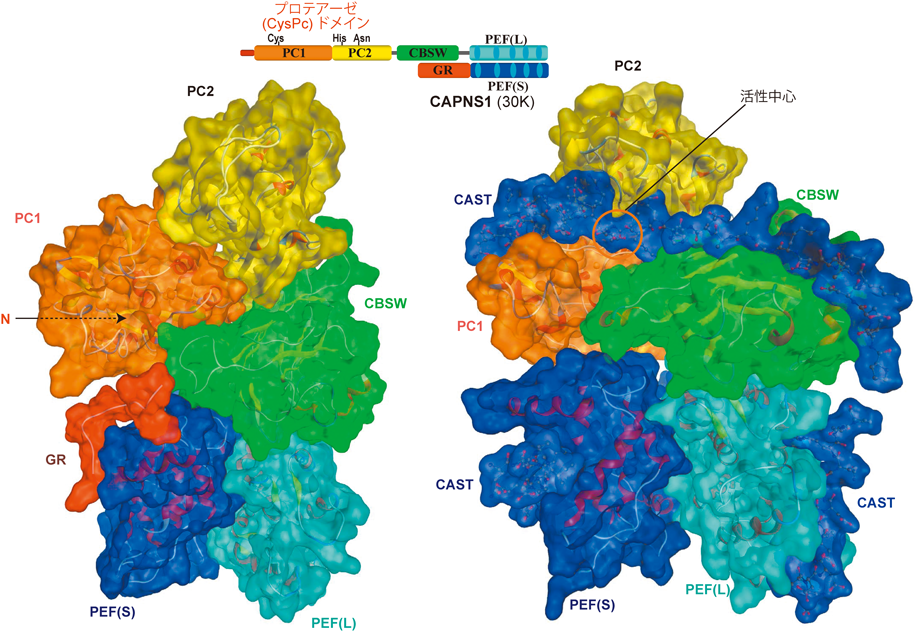
カルパイン-2については非活性型と活性型の両方の全体立体構造が解かれている.各ドメインは時計回りに活性サブユニットのN→C末端方向へ,さらに制御サブユニットのC→N末端方向へと卵形に美しく並んでいる(図2).カルモジュリンのCa2+結合モチーフとして有名なEFハンドモチーフを五つ含有するPEFドメインにカルパインのCa2+依存性は担われていると考えられてきたが,実際には両サブユニットのPEFドメインは触媒(CysPc)ドメインからは最も離れて位置しており,Ca2+の結合の有無によってもほとんど立体構造を変化させないことが明らかとなった13–15).さらに,CysPcドメインのPC1とPC2とに1分子ずつのCa2+が直接結合すること,その結合によってPC1とPC2の配置が変化し,活性中心が初めて形成されることが判明した16–18).
逆にいうと,Ca2+がCysPcに結合していない状態ではPC1/2の配置が開いた状態となり,活性中心が形成されないため不活性な状態が維持されている.カルパインは,細胞質内に存在しており他のタンパク質といつでも相互作用することが可能なため,その活性発現は厳密に制御される必要があるということを反映しているのだろうか.この構造的な危険防止機能に加えて,他にも,100%の活性を出すためには非生理的に高いCa2+濃度が必要である,活性中心が奥まったところに位置しており,構造を持つタンパク質がアクセスしにくい,特異的阻害タンパク質カルパスタチンが過剰量存在している,など二重,三重の安全装置が備わっている.
カルパスタチン(CAST)との複合体の活性型カルパイン-2の立体構造(図2右),すなわち阻害された構造,をみると,カルパインがどのように基質を認識しているかがよくわかる.CASTは鳥類,哺乳類で見いだされる内在性のカルパインに対する特異的阻害タンパク質であり,他のプロテアーゼを一切阻害しない.いくつかのスプライスバリアントが存在するが,基本構造は四つの繰り返し領域であり,各々が1分子のカルパイン-1と-2を(多少の違いはあるが)数nMのKiで阻害する(図1).その他に古典型カルパインのCAPN8/919),CAPN9/S120),CAPN(8)n21)も阻害するが,CAPN3に対しては非常によい基質となってしまいまったく阻害できない22).また,CysPcドメインだけにしたCAPN1(ミニCAPN1と呼ぶ)はCa2+依存的Cysプロテアーゼ活性を保持するが,CASTによる阻害への感受性をほぼ失っている16, 23).
CASTは溶液中では特定の構造をとらない天然変性タンパク質(intrinsically unstructured/disordered protein:IUPまたはIDP)で,Ca2+結合により活性状態になったカルパインと,繰り返し領域内に定義される三つの配列A, B, C(図1)でそれぞれ活性サブユニットPEF (PEF(L))ドメイン,CysPcドメイン,制御サブユニットPEF (PEF(S))ドメインと相互作用している.この際にCysPcの活性中心付近にくる部分は誘導適合(induced-fit)が起こり,一部分がαヘリックスを構成して親和性が高まるのと同時に,そのままでは活性中心と接触してしまうペプチド結合をちょうど避難させるように,カルパイン分子の外側に向かってβターンによる小ループの形成が起こる.その結果,カルパインは猿ぐつわをされたようになり,活性を阻害される.3配列のうちA, Cを欠失させても阻害活性を保持するため,PEFへの結合は,阻害活性の安定性を高めるものの必須ではない.
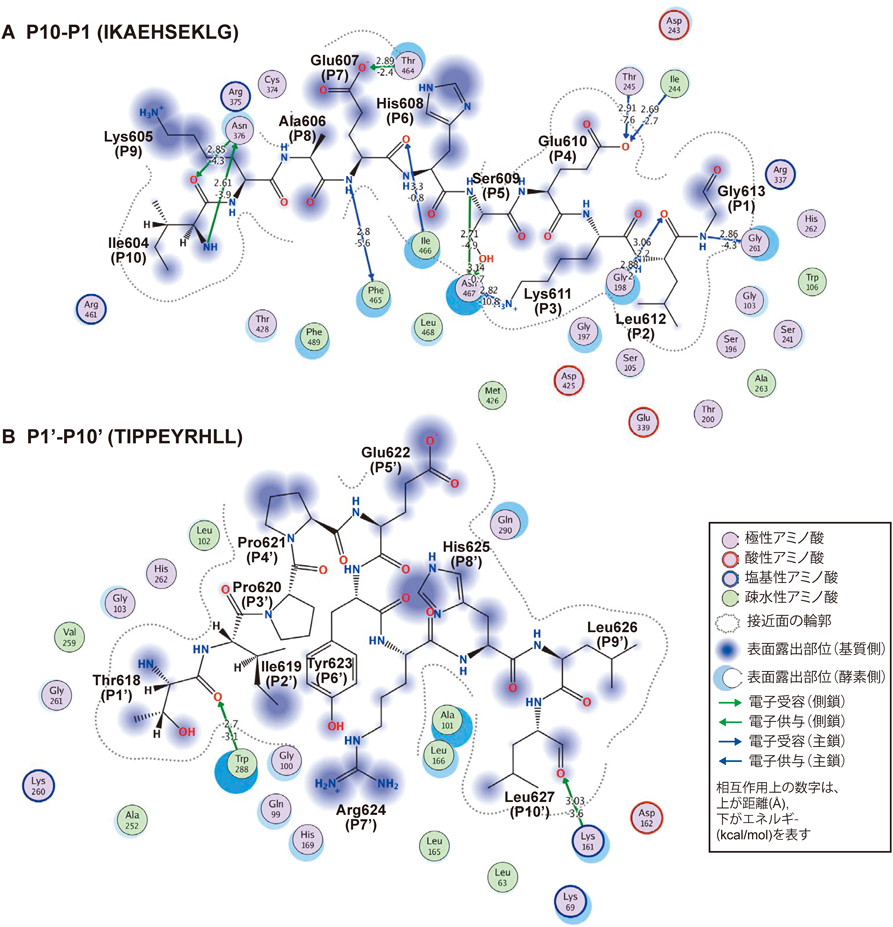
一方,CAPN3とミニCAPN1がCASTに阻害されないことから,CASTとCBSWドメインとの相互作用の重要性が示唆される.CASTの構造から,P3およびP5以前の部位(切断部位のすぐN末端側のアミノ酸をP1,その前をP2…と,逆にC末端側のアミノ酸をP1′,その後ろをP2′…と呼ぶ)はCysPcからはみ出してCBSWドメインと相互作用しているため,この部分が安定で特異的な阻害に必要と推定される(図2).ミニCAPN1にはCBSWドメインが存在せず,CAPN3ではIS1という挿入配列のために,CysPc–CBSWドメインの位置関係が他の古典型カルパインとは異なっている可能性がある.そのためCASTはこれらを阻害できないのだろう.
CASTとの複合体の立体構造から明らかとなったもう一つの特徴は,カルパイン側の原子が基質のアミノ酸側鎖原子とほとんど相互作用していない,という点である(図3).CysPc, CBSWドメインと相互作用するCASTのアミノ酸側鎖は,ほぼすべてがカルパインからみて外側に位置しており,カルパインと直接相互作用しているのはペプチド結合の酸素,窒素原子であった17, 18).これは,カルパインには基質特異性が存在するにも関わらず(たとえば,ある基質タンパク質においては,一定の条件では特定の分解部位が存在する),基質のアミノ酸配列に明示的なルールが見いだせないという現象(後述)の構造的な理由となる.その中で,わずかにP2部位(CASTではLeu)の側鎖は,弱いながらも広範囲でカルパイン分子表面の原子と疎水的相互作用をしており,過去に開発され有効性が認められた多くのカルパイン阻害剤がP2部位にLeuまたは疎水性のアミノ酸を持つこととも矛盾しない.
しかしながら,P2側鎖との疎水性相互作用は多くのClan CAプロテアーゼ[パパインやCysカテプシン(カテプシンの中でCysプロテアーゼである分子種)などの総称,MEROPS(http://merops.sanger.ac.uk/)におけるペプチダーゼ分類法による]に共通するため,これらも阻害してしまう.すなわち現状では,「カルパイン阻害剤」と呼ばれるものはCASTを除いてすべて特異性に問題があるといわざるをえない.この,カルパイン阻害剤の特異性の向上という課題は,いつの時代も多くの化学者を巻き込んで追及されており,最近では,いくつかの新展開がみられている(後述).難しい問題ではあるが,CASTという模範解答が存在するのだから,解のない絶望的な問題ではなく,道はあるはずだ.
カルパインによる基質分解は限定分解と称され,基質の機能・構造を変換・調節する効果をもたらす.いい換えると,カルパインはモジュレータプロテアーゼであるため,その基質特異性,すなわち,カルパインが“何の”タンパク質を“どの部位”で切断するか,という情報はカルパインの生理機能の本質である.問題は,それがなかなか明示できない(既述のように,そもそもカルパインが側鎖をほとんどみていない)ことであるが,最近,新しい方向性がみえてきた.実際,カルパインの基質特異性の研究の歴史は古く,どのようなタンパク質を切断するか,というアプローチに始まり24–26),基質の切断部位配列解析27, 28),さらにアラインメントによる詳細な解析(たとえば部位特異的スコア行列などを用いた方法)が行われてきた29).しかし,これらは既述のP2に疎水性のアミノ酸が比較的多いということ以上に目立った成果をもたらしていない.一方,近年のコンピュータと生物情報学の発達に伴い,プロテアーゼ研究においても機械学習という手法が注目されてきている.ペプチドライブラリーなどを併用して,カルパインの基質特異性に関してもいくつかの解析がなされ,切断部位予測器の作出に至っている30–37).たとえば,我々は,基質配列由来のペプチドアレイを用いた定量的活性構造相関(quantitative activity-structure relationship:QSAR)解析により,陽性的中率(切断すると予測した部位の何%が切断されるか)87.5%という高い値を持つ切断部位予測器を実現した.特に,立体構造が明らかにされている場合には,新規基質における切断部位(の少なくとも一部)の予測が可能であり,P2に加えてP3′およびP4′が切断効率に重要であるということも明らかとなった33).
ただし,in vivoにおいてカルパインが基質タンパク質を切断する状況は,ほとんど構造をとらないペプチドを切断する状態とは著しく異なっているうえ,生化学的にin vitroでカルパインに切断されやすいタンパク質や部位が,必ずしもin vivoでも同様に切断されているとは限らない.将来的には,カルパインと基質タンパク質がどのように細胞内局在するか,足場タンパク質としてどのようなものと相互作用しているか,局所的なCa2+濃度がどのように変化するか,などの条件も(きわめて困難と考えられるが)切断予測器に加味することが必要である.同時に,カルパインという酵素の基本的なスペックを評価するために,上記in vitro基質特異性の解析を有効活用すべきであろう.実際のin vivoとin vitroでの切られ方の比較から,なぜカルパインがわざわざ切れにくい(やすい)部位を切る(切らない)ようになったのか,ということに対する進化的,化学量論的なヒントが得られるかもしれない.たとえばG-タンパク質は酵素としての性能(回転数)を極端に落とすことで多彩な生理機能を果たすようになったとされる38).同様の歴史(未来)が,カルパインのプロテアーゼとしての性質にも起きた(起きる)のかもしれない.
カルパインが関与する疾患は次の3通りに大別される.
(1)ヒトの標準型カルパインの活性が疾患の増悪に関与するもの.神経変性疾患,心・筋疾患,虚血性疾患,がん,眼疾患など.
(2)カルパイン遺伝子の病原性変異によって起こる遺伝性疾患(「カルパイノパチー」と呼ばれる,表2).肢帯型筋ジストロフィー(limb-girdle muscular dystrophy:LGMD)2A型(LGMD2A),常染色体優性新生血管炎症性硝子体網膜症(autosomal dominant neovascular inflammatory vitreoretinopathy:ADNIV),魚鱗症(ichtyosis),好塩基球性食道炎(eosinophilic esophagitis:EoE),遺伝性痙性対麻痺(hereditary spastic paraplegia:HSP)など.
表2 カルパイノパチー| 遺伝子 | 発現 | 遺伝子変異と関連する疾患 | 文献 |
|---|
| CAPN1, Capn1 | 全細胞 | 痙性対麻痺,血小板不全(in vitro) | 103, 164) |
| Capn2 | ほぼ全細胞 | 胎生致死 | 165, 166) |
| Capns1 | 全細胞 | 胎生致死 | 167, 168) |
| CAPN3, Capn3 | 主に骨格筋 | 筋ジストロフィーetc. | 85, 87, 169, 170) |
| CAPN5, Capn5 | ほとんどの細胞 | 硝子体網膜症 | 90, 95) |
| Capn6 | 主に胎生筋,胎盤 | 発育過度 | 139) |
| CAPN7 | ほとんどの細胞 | np* | — |
| Capn8 | 主に胃腸 | 胃出血 | 19) |
| Capn9 | 主に胃腸 | 胃出血 | 19) |
| CAPN10 | ほとんどの細胞 | 2型糖尿病 | 161) |
| CAPN11 | 主に精巣 | np | — |
| CAPN12 | 主に毛包,皮膚 | 魚鱗症 | 196) |
| CAPN13 | ほとんどの細胞 | np | — |
| CAPN14 | 主に食道 | 好塩基球性食道炎 | 96) |
| CAPN15/SOLH | ほとんどの細胞 | np | — |
| CAPN16/ADGB/C6orf103 | 主に精巣 | np | — |
CAPNxおよびCapnxは各々ヒト,マウスの遺伝子を示す.
* np:ヒト,マウス両方ともまだ報告がない. |
(3)寄生虫・病原性微生物が原因となる疾患,つまり感染症で,ヒトのカルパインあるいは自身のカルパインが病原体の生存や感染などに必須なもの.マラリア,トリパノソーマ症,ジストソーマ(住血吸虫)症,カンジダ症,歯周病など.
これらの疾患に対して,どのような治療戦略が可能だろうか.まず,(1)のほとんどの疾患に対して,ヒトの標準型カルパインの阻害剤が治療薬となりうる.また,(2)では変異型カルパインの機能不全もしくは過剰活性化が発症原因と考えられる.そのため,遺伝子治療などでその活性を正常化することが究極の治療であるが,過剰活性化に対処するにはカルパイン阻害剤が有効と考えられる.また,特定の活性実体に着目した,活性賦活化の可能性も示唆されている(後述).(3)においては,いわばハイジャックされた状態ともいえるヒト(=宿主)のカルパインに対して(1)と同様の戦略で対処するとともに,寄生虫・微生物自身のカルパインを標的とした治療法が検討されている.
以下でも引用しているが,阻害剤ではなくカルパインあるいはCASTの遺伝子改変[ノックアウトあるいは過剰発現トランスジェニック(Tg)]マウスを用いた疾患研究は,因果関係を明確に示せる点で非常に優れている.ただし,これらの系は阻害剤に比べると極端な状況となるため,そのデータの解釈には注意を要する.以下,各カテゴリーを代表する疾患例を概説するが,紙面の都合上,疾患モデルにおいて検討された阻害剤と合わせて表3とした.
表3 カルパイン阻害剤の効果に関する報告(抜粋,2010年以降)| 阻害剤,ワクチン | 改善がみられた疾患モデル(悪影響がみられた現象) | 発症/感染後の処置* | 標的カルパイン | 文献 |
|---|
| ALLNal | コクサッキーウイルスB3感染 | x | n/a** | 171) |
| (子宮着床) | | CAPN2 | 172) |
| 滑脳症 | x | n/a | 173) |
| ALLNal, PD150606 | 網膜色素変性症 | x | n/a | 174) |
| ALLNal, calpeptin | 乳がん | x | CAPN1 | 175) |
| Calpeptin | パーキンソン病 | | n/a | 176) |
| 多発性硬化症 | x | n/a | 177, 178) |
| 特発性炎症性筋疾患 | x | n/a | 179) |
| 肺線維症 | x | n/a | 180) |
| MDL28170, calpeptin | 黒色腫 | x | n/a | 181) |
| MDL28170 | 心虚血–再灌流 | | CAPN1 | 54, 182) |
| 慢性心疾患 | x | n/a | 183) |
| リーシュマニア症 | x | L. amazonencisカルパイン | 147) |
| アフリカ睡眠病 | x | T. cruziカルパイン | 184) |
| 子癇前症 | | n/a | 185) |
| 脊髄損傷 | x | n/a | 186) |
| Calpain inhibitor XI (AK295), calpastatin peptide (CAST) | 網膜色素変性症 | x | n/a | 187) |
| SNJ1945 | 自己免疫性脳炎・多発性硬化症 | x | n/a | 188) |
| 網膜虚血 | x | n/a | 189) |
| 滑脳症 | | n/a | 47) |
| 心虚血–再灌流 | | n/a | 190) |
| 外傷性脳障害 | x | n/a | 191) |
| BDA-410 | マカドジョセフ病 | | n/a | 192) |
| 加齢症候群 | x | CAPN1 | 80) |
| 鎌形赤血球症 | x | CAPN1 | 193) |
| 腹部大動脈瘤・アテローム性動脈硬化症 | x | CAPN1 | 58) |
| Macrocyclic aldehyde (CAT811) | 白内障 | x | n/a | 72) |
| NH2-GRKKRRQRRRPP QPDALKSRTLR-COOH (Tat-μCL), PD150606 | 網膜色素変性症 | x | CAPN1 | 194) |
| Dipeptidyl α,β-unsaturated ester | マラリア | x | Pf-カルパイン(?) | 195) |
| Hypervalent organotellurium compound (RF19) | マラリア | x | Pf-カルパイン(?) | 145) |
| Sm-p80(組換えタンパク質) | ジストソーマ症 | x | Smp_157500 (S. mansoniカルパイン) | 150, 151) |
*「x」は,報告した論文において,発症後・感染後に阻害剤を投与して効果をみていることを表す.
** n/a:特定のカルパイン分子種に限定されない. |
1)カルパイン活性による症状増悪(神経変性疾患,心・筋疾患,がん,眼疾患)
カルパインは,多くの神経変性疾患(アルツハイマー病,パーキンソン病,滑脳症など,表3参照)において,増悪因子として位置づけられる39–43).ただし,アルツハイマー病の発症機序におけるカルパインの関与は限られたものと,現在では考えられているようである44–46).また,遺伝性疾患の滑脳症では,責任遺伝子産物の一つであるLIS1がモータータンパク質ダイニンの制御に関与しており,カルパインにより分解される非常に半減期の短いタンパク質であることが明らかとなった.一方のアリルにLIS1の機能損失型変異を持つヘテロ接合体では,正常アリルからのLIS1だけではタンパク質量が不足するため,細胞内輸送が破綻をきたし発症すると考えられている.カルパイン阻害剤の投与によってLIS1の減少を抑制すると症状が緩和されることから,投与時期を適切に設定することにより,発症を予防する効果が注目されている47, 48).
多くの心疾患(急性および慢性)において,カルパインの活性は心筋タンパク質の過剰分解などにより症状を悪化させることが示されてきた49–54).また,動脈硬化モデルにおいてもカルパインの活性化が見いだされている.そのため,これらの疾患に対して,発症前後からカルパインを阻害することが症状緩和や治療効果をもたらすと期待されている55–58).ただし,遺伝子改変マウスの表現型,CAST過剰発現Tgマウスが拡張性心筋症と瘢痕治癒障害を示し,心筋特異的なCapns1ノックアウトマウスでは血流ストレスにより脆弱となること,には要注意である59, 60).また,カルパインのタイムリーな活性化が心筋症状や心筋毒性の克服に,たとえば免疫系の調節を通じて必要であることも示されている61, 62).
同様に,カルパイン-CAST系ががんの病態機序に関与することは古くから知られており,多くの場合,がんの進行に伴ってCAPN1, CAPN2,あるいはCAPNS1の高進が報告されているが,逆にCASTの高進も観察されている63, 64).また,がん組織などでよくみられる病的血管新生がCASTの抑制によるカルパイン-2の高進によって増強されるという報告65)や,非標準型カルパインであるCAPN3の黒色腫細胞株における発現変動に関する報告66–68)など,その役割(活性ががんを促進するか抑制するか)や細胞種依存性について,混沌としつつも新たな知見が蓄積されつつある.
カルパイン阻害剤を眼疾患の治療に用いる試みも盛んである.これは,主にカルパインにより部分分解されたクリスタリンの凝集が白内障の原因となるためである.第一世代阻害剤であるE-6469),SJA6017(第二世代)70),を経て,最近では大環状分子構造を持つ阻害剤(第三世代)について検証モデルでの好成績が報告された71, 72).カルパイン阻害剤の治療薬としての実用化が最も近い分野の一つといえる.他に,カルパイン阻害が有効と考えられる眼疾患として,ロドプシンやホスホジエステラーゼなどの遺伝子変異により発症する網膜色素変性症がある.この疾患では,カルパインの下流でカスパーゼやカテプシンも活性上昇し73, 74),熱ショックタンパク質HSP70の発現量が低下していること75)も増悪因子である.これらに対する処置をカルパイン阻害剤に組み合わせるという,治療戦略が想定される.
一方,デュシェンヌ型筋ジストロフィー(Duchenne muscular dystrophy:DMD)は長らくカルパインの阻害により症状が緩和すると信じられてきたが,最近のマウスモデル76)やイヌ(ゴールデンリトリーバー)モデル77)での結果によると,カルパインはDMDにおいて活性上昇していても,治療標的とするのは難しいことが示されている78).ただし,DMDのマウスモデル(mdxマウス)やDMD患者の骨格筋では,早老症の責任遺伝子産物であるα-klothoの発現が減少しており,mdxマウスにα-klothoを発現させると筋ジストロフィーの症状が緩和されることが最近見いだされた79).α-klothoはCa2+恒常性をつかさどっており,その欠損によるカルパイン-1の過剰活性化が早老症状の発症に大きく寄与し,カルパイン阻害剤によってその症状が緩和されることも明らかとなっている80).そのため,筋ジストロフィーにおいてα-klothoを補償する薬剤とともにカルパイン阻害剤を使用することは,大きなメリットがあるかもしれない.
2)カルパイノパチー(LGMD2A, ADNIV, EoE,その他)
LGMDは四肢の近位筋に症状が集中する筋ジストロフィーであり,LGMD2Aは常染色体劣性のLGMDの1つである81, 82).世界的には最も頻度の高いLGMDであり(約20人/100万人),バスク国,レユニオン島など特定の地域では,かなり高い罹患率(50~10,000人/100万人)を示す83, 84).1995年,骨格筋に偏って発現するCAPN3遺伝子の変異がLGMD2Aを引き起こすことが発見85)され,カルパインの機能損失がヒト疾患を引き起こす,いわゆる「カルパイノパチー」の最初の例となった85–87).LGMD2Aでは,ほとんどの場合CAPN3が機能不全となっており86),このことも他の筋ジストロフィーでは標準型カルパインの活性が症状の悪化につながっている事実78)とまさに対照的である.その後,LGMD2Aマウスモデル(Capn3−/−,およびCapn3C129S/C129S)を用いた実験により,CAPN3はプロテアーゼ機能とは独立に筋細胞のCa2+動態制御にも関与することが示された88, 89).よって,LGMD2Aの場合はCAPN3を阻害するのではなく,CAPN3の機能を遺伝子治療などで回復させることが必要である.
ADNIVは網膜症から失明に至る重篤な優性遺伝性疾患であり,CAPN5が責任遺伝子産物となっている90, 91).CAPN5はほぼすべての細胞に発現するが,中枢神経系での発現が高い92, 93).ADNIVではこれまでにCAPN5の病原性変異としてR243L, L244P, K250Nが同定されているが91),このうちR243Lは機能獲得型の過剰活性化体であることが示唆されており,構造機能相関の視点からも興味深い.実際に,CAPN5:R243Lを発現するTgマウスは,組織学・生理学的にADNIV様症状を呈する一方94),Capn5−/−では顕著な表現型が観察されていない95).このTgマウスでは症状が顕著になる以前から炎症関連遺伝子の発現変動が認められており,ADNIV発症の初期機構や共通の症状を示すブドウ膜炎の病態解析に有効なモデルマウスとしても注目されている.以上よりADNIVの治療には,過剰なCAPN5活性を阻害することが有効と考えられる.他の眼疾患への取り組みが参考となる一方で,CAPN5の活性を標準型カルパインの活性と見分けることが課題とされる.
EoEは,近年,世界的に発症増加が報告されているアレルギー性の消化管疾患である.ゲノムワイド関連解析によりCAPN14遺伝子の一塩基置換(single nucleotide polymorphism:SNP)がリスク因子の一つとして同定された96, 97).CAPN14は,in vitroではIL-4によって発現誘導される.しかし,マウスでは偽遺伝子となっていることもあり,あまり注目されてこなかった98–100).しかし,食道に偏って発現すること,EoEで発現が高進しており,in vitroではIL-13によっても発現が誘導されることが明らかとなった96).現時点では発症機序はまったく不明であるが,高リスク型のSNPでは,発症時の発現高進が抑制されているという興味深い現象が起きている.CAPN14がプロテアーゼとして機能している可能性について,現時点では間接的にのみ示されている.そのため,今後の機能解析により,EoE治療の標的として,CAPN14の活性(機能)を阻害または賦活する方法論がみえてくると期待される.
ヒト遺伝性疾患との関連はまだ報告されていないが,消化器官機能との関係において胃粘膜に局在するCAPN8, CAPN9も注目に値する19, 101).遺伝子改変マウスの実験より,Capn8−/−, Capn9−/−,あるいはCapn8C105S/C105Sは,野生型マウスよりアルコールストレスによる胃出血傾向が顕著に高い19).また,CAPN8と9は複合体G-カルパイン(CPAN8/9)を形成しており,一方の欠損でG-カルパインの機能は損なわれてしまう19).すでに,ヒトCAPN8, CAPN9とも多くのSNPが報告されていて,その一部はCAPN8, 9の活性を失わせる19).つまり,これらのSNPを持つ人でストレス性胃出血傾向の増加がある可能性,その治療としてはLGMD2Aと同様にカルパインの活性回復・賦活が有効であることが示唆されている102).
ごく最近,CAPN1が常染色体劣性遺伝性疾患であるHSPの責任遺伝子(SPG76に相当)として報告された103).また,CAPN1のSNPがヒトおよびイヌの脊髄小脳変性症にリンクしていることも明らかとなってきた104, 105).遺伝形式およびモデル生物での解析結果より,これらの疾患においてもCAPN1を補完することが適切な治療方針であることが示されている.さらに,CAPN12の活性損失型変異が魚鱗症に関与するという報告もなされた196).現時点ではCAPN12の病態生理機能は不明で,他の魚鱗症責任遺伝子(脂質トランスポーターのABCA12など)の修飾遺伝子(modifier gene)の可能性もあり,今後の解析が待たれる.
3)カルパインを介した感染症(マラリアから歯周病まで)
寄生虫や病原性微生物による疾患はいまだに世界的な脅威である.特筆すべきことに,これらの生物には多様なカルパイン分子種が存在し,その病原性や感染・生存に重要な役割を果たしており,前途有望な治療標的となっている106).以下に述べる疾患のうち,トリパノソーマ症,リーシュマニア症,およびジストソーマ症は,世界保健機関(World Health Organization:WHO)によって「顧みられない熱帯病(neglected tropical diseases:NTD)」と定められており,世界で10億人以上を苦しめている.関連する研究成果を予防・治療へと応用することが,世界レベルでの包括的取り組みへの一助として期待されている.
マラリアは,アピコンプレックス門の胞子虫Plasmodium falciparumに感染したハマダラカによって媒介され,発熱,貧血,脾腫などを引き起こす最も重篤な寄生虫病の一つである.P. falciparumにただ一種あるカルパイン(Pf-カルパイン)は細胞周期に関与しており,生存に必須である107).他にも宿主(ヒト)のヘモグロビンを消化して生き延びるために重要なAspおよびCysプロテアーゼが複数同定されており108).これらのプロテアーゼに対する阻害剤開発と併用が主要な治療戦略とされる.一方,感染された宿主のカルパイン-1の役割については不明確で,P. falciparumの増殖に必要であるという報告109),および,それを反証する報告110)がある.
トリパノソーマ症およびリーシュマニア症は,ユーグレノゾア門Trypanosoma属やLeishmania属によるもので,それぞれ複数の病型がある.いずれも昆虫によって媒介され,たとえば,アフリカ睡眠病は,ツェツェバエにより媒介されたTripanosoma bruceiにより発症,睡眠周期撹乱から昏睡・死に至るもので,代表的なNTDである111).驚くべきことに,これらの寄生虫はカルパイン遺伝子を18~27種も有するが,そのほとんどで活性中心を形成すべきCys, His,またはAsn残基が他のアミノ酸に置換している112).本稿の定義では,これらもカルパインであり(2節参照),実際に,寄生虫の生活環において重要な機能を担っているようである[例:T. bruceiのClpGM6は,CysPc(His→GlnあるいはCys→Gly)およびCBSWドメインを各三つずつと二つのMITドメインの他に,68アミノ酸からなる繰り返しユニットが70回以上繰り返した構造を持つ820 kDaの巨大タンパク質で,形態変化に必須である113).図1参照].今後,これらのやや変則的なカルパイン分子種についても,機能に関する知見に基づいた治療・予防手段の開発が期待されている.
ジストソーマ症は,扁形動物門の住血吸虫Schistosoma mansoni, S. japonicum, S. haematobiumがヒラマキガイによって媒介されて発症し,尿路線維症,腎炎,下痢・血便,肝硬変などの慢性症状を呈する114).S. mansoniは七つのカルパイン遺伝子を持ち,そのうち四つは古典型カルパインを,一つはCAPN7ホモログをコードする115).すでに,古典型カルパインの一つ,Smp_157500 (図1)は有望なワクチン候補として注目されているが,その生理機能自体はまったく不明である.
カビ・酵母では,その多くに(本稿執筆時点で全ゲノム解析済みの真菌類449属のうち431属)カルパイン遺伝子が1コピー以上見いだされている(しかし,カルパインは,SchizosaccharomycesやPneumocystisなどには存在しない.3節参照).真菌類には,白癬(水虫・たむしなど)の原因菌(Trichophyton tonsuransなど)から,重篤な感染症,特に日和見感染によって致死的な全身症状を引き起こすもの(Candida albicans, Cryptococcus neoformans, Aspergillus fumigatus, Coccidioides immitisなど)が含まれる.
真菌の多くは低pH(酸性)環境で成育していることが多いため,ヒト組織に寄生するためには高pH(中性)側に環境適応をする必要がある.何とその経路で機能するのが,前述のように,真菌のカルパインRim13/PalBであり116–118),これらはヒトCAPN7のホモログである(表1)119).高pH適応シグナル経路の要はRim13/PalBがリクルートされた転写調節因子のRim101/PacCを限定切断・活性化して,適応に関わる遺伝子群の発現を切り換えることである119–121).このカスケードは,真菌感染の防止・関連疾患の治療にきわめて効果的な標的を提示するものであるが,細胞ストレスに反応してカルパインが活性化する機構を遺伝学的に明示した初めて(現在でもほぼ唯一の)の例としても重要である.
以前より指摘されていることだが,細菌のカルパインホモログは少なく,現時点で解読されている4000以上の細菌の全ゲノム上にわずか67遺伝子(55の細菌)しか存在しない122).その中で,ヒトの健康・疾患に関わるものとして,歯周病を引き起こすPorphyromonas gingivalisが有するTpr (thiol proteaseの略)というカルパインホモログ遺伝子があげられる.Tprは菌の生存や感染に必須であり,実際にCa2+依存的なCysプロテアーゼ活性を持つ.また,ヒトの補体タンパク質などを基質とするため,口腔内の免疫系から逃れるために機能することが示唆されている123).
カルパイン関連疾患のほとんどについて,カルパイン阻害剤が治療の第一戦略となる.阻害剤の特異性,作用機序の理解や扱いやすさは,現在,どのレベルに達しているのか.また,カルパイン遺伝子変異による疾患,カルパイノパチーの中でLGMD2Aは機能欠失が発症原因だと確認されている.つまり,CAPN3の活性を補完することが必要となる.現在までのCAPN3に関する知見は,治療戦略にどのような可能性を与えるだろうか.
1)カルパインをやっつけろ!(できれば特異的に)
第一世代阻害剤(ロイペプチン,E-64やその誘導体1))は,カルパインに特異的ではなく,他のパパイン様プロテアーゼ124–126)のみならず,たとえばマトリックスメタロプロテアーゼ(matrix metalloproteinase:MMP) 2127)などに対しても広い阻害活性を示す.続いて,SJA601770)やPD150606128)などの第二世代阻害剤が開発されているが,残念ながら特異性はまだ十分ではない.しかし,その後の構造生物学の発展により,非常に有望な第三世代阻害剤が開発されている.たとえば,既述のようにCASTによるカルパインの阻害は,部分的なαヘリックス構造形成が特異性を担保する.これに倣い,ペプチドの側鎖の一部に分子内架橋を施してαヘリックス構造を固定し,カルパインへの特異性向上に成功したことが報告されている129).また,大環状の分子を用いた阻害剤は目的外のプロテアーゼへの誘導適合が起こりにくく,カルパイン以外にもさまざまなプロテアーゼに特異的な阻害剤の開発に応用されている71, 130, 131).カルパイン阻害剤では,白内障治療薬に向けてきわめて有望な結果が得られている72).
特異性の点で希望の光を投げかけるような,ひと味違うアイディアとして,カルパイン自身の一部の配列を阻害剤に用いるという方法論は興味深い132).安定化因子(一種のシャペロン)との相互作用が競合的に阻害されると,そのカルパイン分子が不安定化・不活性化するというものである.また,既存の阻害剤は,ほぼすべて活性中心を標的としているため,逆に特異性を下げる一因となっている(カルパインは基質のアミノ酸側鎖をほとんど認識しないし,P2の疎水性残基嗜好性は他のパパイン様プロテアーゼでも保存されているため).しかし,狙い方を工夫することで,特異性を持たせることはできないだろうか.たとえば,カルパインの活性化とは,すなわちPC1とPC2が近づいてCys-His-Asnからなる活性中心トライアッドを形成することなので,不活性化状態のカルパインのPC1とPC2の間隙に入り込んでこの配置変換を阻害する物質は,カルパインにきわめて特異的なものとなりうるのではないか.
一方,阻害剤を用いて得られる結果については,「本当にカルパインが標的なのか」という点に検討の余地があるだろう.よく使われるALLNalやMDL28170(各々カルパイン阻害剤1および3とも呼ばれる)を含め,カルパイン阻害剤のほとんどはCysカテプシンの多くをよく阻害する.そのため,阻害剤による生物学的な効果ではカルパインとカテプシンの寄与が見分けられず,あいまいになっている報告が山のようにある.たとえば,カテプシンにある程度特異的な阻害剤(カテプシンB, K, S, Lに対して,各々Ca-074, odanacatib, CLIK-060, CLIK-148などが知られている133–135))を用いて検証すべきではないか.その結果,ALLNalと同様の効果があれば,実はカテプシンが増悪因子である可能性が高い.
繰り返しになるが,現在のところCASTが唯一,完全にカルパインに特異的な分子である.CASTの存在は,きちんとカルパインだけを阻害する薬剤は開発可能であることを示している.もちろん,実用に耐えるために解決せねばならない課題は(物性・価格など)ある.
もう一つ興味深い視点は,アロステリック(活性中心とは別の部位に作用する)阻害剤である.これまでに,α-メルカプトアクリル酸誘導体(PD150606など)がPEFドメインに結合し,非競合的にカルパインを阻害することが明らかとなっている128).ところが最近,PD150606はCysPcドメインからのみなるミニカルパインの活性を阻害することから,やはり活性領域に作用していることが判明した136).残念ながら,その詳細な阻害機構は不明であるが,ジスルフィド結合により二量体化したα-メルカプトアクリル酸誘導体は,単量体に比べて数百倍強いカルパイン阻害活性を示すことが報告されている137).これらの阻害剤に関する知見を合理的に応用し,新たなアロステリック阻害剤を開発する可能性が期待される.実際,CycPcドメインと立体構造の類似するカテプシンKでは,生物情報学的手法でアロステリック阻害剤の開発に成功しており138),カルパインにおいても十分実現可能と考えられる.
さらにまったく別のコンテキストから,CAPN6の抑制というストラテジーも興味深い.CAPN6は,ヒトカルパインの中で唯一活性中心のCysがLysに変異しており,プロテアーゼ活性を有しないユニークな分子で,胎生筋・骨や成体の胎盤に発現している.Capn6ノックアウトマウスでは骨格筋の有意な肥大に伴う体重の増加が観察されており,CAPN6は骨格筋の発達抑制に関与すると考えられる139).そのため,ミオスタチン(筋発達を抑制するサイトカイン)の抑制ストラテジー140, 141)と同様に,DMDやLGMD2AにおいてCAPN6の抑制による筋量増加が治療効果を与える可能性が考えられている.興味深いことに,CAPN6はマクロファージでmRNAスプライシングに関与しており,その抑制によりアテローム性動脈硬化を有意に改善できるため142),新しい標的カルパインとして大きな注目を浴びている.
2)寄生虫・病原性微生物のカルパインもやっつけろ!(活性はともあれ)
既述のとおり,ヒトに深刻な疾患をもたらす寄生虫・病原性微生物の多くにおいてもカルパインは,生存,増殖,感染などに必須の機能を担っている.これらは,厳密にはヒトカルパインとは異なる性質を持つことが予想されるので,疾患治療における特異的な標的として非常に有望である.中には,ヒトカルパインに対する阻害剤が効くものもある.また,宿主が発現するヒトカルパインが寄生虫・病原性微生物の感染,増殖のためにハイジャックされている場合には,ヒトカルパインに対する阻害剤が一石二鳥の効果をあげることも予想される.たとえば,マラリアの原因となるP. falciparumのPf-カルパインも生存に必須であるが,ALLNal, ALLMal, BDA-410など従来のカルパイン阻害剤がP. falciparumの赤血球への侵入や増殖を抑制したという報告がある143, 144).最近ではTeを含む超原子価有機テルル化合物がPf-カルパインを阻害するという報告もある145).肝心のPf-カルパインの活性や機能については不明な点が多く,最近確立された活性を持つ組換えPf-カルパインの発現系を用いた解析に興味が持たれる146).同様に,カルパイン阻害剤MDL28170は,宿主細胞への毒性はほとんどなく,TrypanosomaやLeishmaniaの増殖を抑制する効果が報告されている147, 148).しかしながら,これらの寄生虫のカルパインは活性中心残基のいずれかを失っているものがほとんどのため112),MDL28170が何に作用しているのかは不明であり,宿主のカルパイン阻害が上記の効果を引き起こしている可能性も大きい.
S. mansoniのカルパインSm-p80(細胞膜上に提示されているカルパインSmp_157500のC末端80 kDa部分)は,ジストソーマ症の有望なワクチン候補の一つとなっている149, 150).残念ながらこのカルパインの生理機能については明らかではないが,ヒヒを用いた感染実験やさまざまな属の住血吸虫を用いた実験から,ワクチンとしての優秀性と属を超えた広汎性が示されている150, 151).寄生虫のカルパインを標的とした,特異的戦略の例である.
真菌類,細菌(歯周病菌)のカルパインは上記寄生虫のカルパインよりもさらにヒトカルパインとの構造の差異が著しいため,特異的阻害剤のデザインにはもってこいであると考えられる.特に,抗生物質の使いにくい真菌類の引き起こす日和見感染などに対して貢献できる可能性がある.現時点では,これらのカルパインに対する阻害剤の解析はほぼ皆無で,立体構造解析も行われていない.“宝の山”となる可能性を秘めた分野に思える.
3)カルパインの活性を取り戻せ!(でも,どうやって?)
現在のところ,LGMD2Aは唯一,ヒトカルパイン遺伝子の機能損失型変異で生じることが確認されている遺伝病「カルパイノパチー」である.この疾患の治療にはCAPN3の活性を補完することが必要となる.ヒトでの疾患はいまだ確認されていないが,CAPN8, 9, 11, 12, 13, 15, 16に起因するカルパイノパチー(もしあれば,であるが)も,同様にカルパインの活性を取り戻すことが必要と考えられる.カルパインの分子量が現存するウイルスベクターに入りうるサイズ(~100 kDa)であることと,カルパインにはよい活性化剤が存在しないことから,現状では遺伝子治療が第一選択となる140, 152, 153).
LGMD2Aの遺伝子治療にあたって,CAPN3のプロテアーゼ活性の制御機構がいまだに不明であることは,大きなデメリットである.たとえば,CAPN3発現AAVベクターを用いた遺伝子治療開発の過程で,骨格筋はCAPN3の外部からの発現を許容する(制御できる)が,心筋その他の組織ではそうではなく,CAPN3の発現が組織を損傷してしまうことが明らかとなった59, 153).解決法として,発現ベクターのプロモーターの骨格筋特異性を(たとえば,CAPN3の遺伝子領域を用いるなどして)高めることなどが模索されている153).これに関連して,活性のある完全長のCAPN3を外来遺伝子として発現させることに危険が伴うとすれば,点変異のLGMD2Aの場合(全LGMD2Aの1/3以上に当たる)には,遺伝子編集技術とiPS細胞の技術を用いて,変異を修正するということも今後視野に入ってくるだろう154).
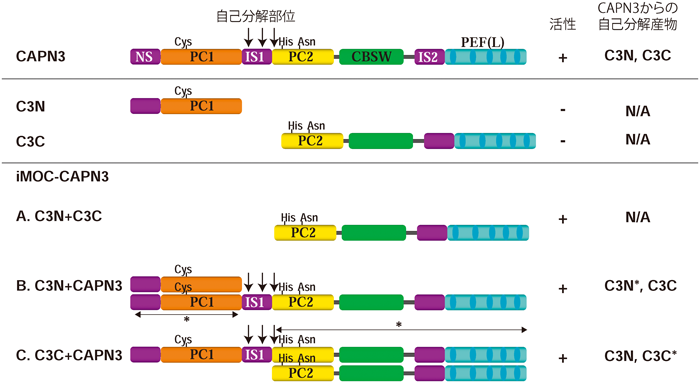
一方,CAPN3には分子間相補(intermolecular complementation:iMOC)という特筆すべき性質が存在する155)(図4).CAPN3は強力な自己分解活性を持っており,in vitroでは半減期10分未満で消失してしまう.この分解によって,約35 kDaのN末端断片(C3N)と約58 kDaのC末端断片(C3C)が生じる.前者には活性中心のCys残基が,後者にはHis, Asn残基が含まれるため,それぞれだけではプロテアーゼ活性を持ちえない.
しかしながら,CAPN3の両断片は再び会合して活性を回復できるのである.さらに,C3NとC3Cの間でのiMOCに加えて,全長とC3N(あるいはC3C)との間でもiMOCが生じる.すなわち,点変異により不活性となった全長CAPN3は,その点変異の位置を含む野生型断片(C3NまたはC3C)が存在するとiMOCにより活性を取り戻すことができることとなり,LGMD2Aの遺伝子治療を野生型断片(C3N, C3C)の発現で行えるかもしれない.これらの野生型断片は単独では活性がないため,CAPN3を発現していない骨格筋以外の組織では活性を持ちえないと期待される.これは安全性の面で大きな利点であろう.
iMOC自体は,ちょうど大腸菌のβ-ガラクトシダーゼのα相補と同様の機能であるが,自己分解を経ての相補という現象は,原核・真核細胞のプロテアーゼでは,現在のところ唯一の例であり,他にサイトメガロウイルス(CMV)と単純ヘルペスウイルス(HHV, HSV)のプロテアーゼ(アセンブリンと呼ばれる)で報告156, 157)があるのみである.カスパーゼ,やクラミジアのプロテアーゼ(CPAF)なども前駆体が2断片に切断されて分子内二量体を形成して活性化するが,CAPN3のように両断片を溶液中で混合するだけで活性を取り戻すことはない.このように,特異な性質であるため,自己分解とiMOCという性質を利用した治療法の可能性を現実化するためには,いつ,どこで,どのような形でCAPN3が活性を発現するのかについてもっと研究を進める必要がある.
一方,LGMD2A型変異CAPN3の中には活性が野生型よりも高進しており,過剰な自己分解によりCAPN3タンパク質が消失してしまっている86, 158)というケースも存在している.これらの多くはPEFドメインに点変異体がみられるため,前述のPD150606などの,PEFドメインに相互作用する分子を利用して過剰活性化型変異体CAPN3の活性を部分的に阻害すると,CAPN3が安定化されて症状が軽減するという可能性も考えられる.
プロテアーゼがさまざまな疾患治療の標的となりうることはすでに周知のこととなっている159).その中で,カルパインに関しては,酵素としての実態・生理機能理解のところで少し立ち遅れていることは否めない.今回,あえてCAPN10と2型糖尿病(T2DM)に関しては言及しなかったが,それは,CAPN10がどのようにT2DMに関与しているのか,治療に際して補完するべきか阻害するべきか,がまったく不明なままであるからだ160–162).今後の研究の進展が要注目である.
本稿では,阻害剤の特異性についてしばしば言及したが,「使える」阻害剤という概念はその目的(疾患治療vs純粋科学的実験)によっても変わる131, 163).疾患治療を考えれば,阻害剤に特異性がなくても,副作用なく症状を緩和できるなら,まったく問題ないともいえる.たとえばカルパインとカテプシンの両方が症状悪化をもたらしているケースも少なくないが,このような場合はむしろ両者に広く効く阻害剤の方が望ましいとさえいえる.
しかしながら,対症療法ならではの後手感や未知の因子に対する脆弱性は除かれるべきだし,長期的な視点からも,思わぬ副作用を避けるため,阻害剤の特異性は十分に磨かれるべきものであると考える.また,疾患モデルにおけるカルパイン阻害剤の効果は,使用目的に応じて,発症前からの投与と発症後の投与による治療効果をそれぞれ評価することが,「使える」阻害剤を見分けるために必要であろう.理想的なカルパイン阻害剤を目指して,特異性を高めるための可能性はいくつかあげたとおりである.筆者らはカルパインがどのように基質特異性を発揮するのかという観点から,今後の開発を下支えしたいと思う.
カルパイン阻害剤の開発が望まれる一方,カルパイン阻害による副作用も忘れてはならない.特に心筋,免疫系,胎盤などにおけるカルパイン活性の重要性は,比較的最近に認識されてきたことであり,新たなカルパイノパチーメカニズムを内包している可能性がある.さらには,カルパインの阻害・活性化と,他の分子の阻害・活性化とを合理的に組み合わせることを,治療効率という観点から検討することもありうるのではないだろうか.
現段階では,カルパインの生理機能に関する基礎的な理解がまだまだ不足していることを痛感する.しかし,原石にハズレはなく,正しい順番で拾うと綺麗に光るようである.カルパイン自体の解析はもちろん,それが関与する現象・ネットワークを広く理解することが今後のカルパイン研究の発展につながると考えられる.
謝辞Acknowledgments
筆者らがカルパイン研究を始めることができ,今日まで続けることができたのは偏に鈴木紘一先生の思いやり溢れたご指導の賜物で,本総説は天国の先生に捧げます.また,研究へのご指導・ご協力をいただいた旧東京都臨床医学総合研究所遺伝情報研究部門の榎森康文博士,本間好博士,川崎博史博士,秋田朗子博士,大海忍博士,大野茂男博士,西道隆臣博士,阿部啓子博士,旧東京大学分子細胞生物学研究所生体超高分子研究分野の石浦章一博士,川原裕之博士,前田達哉博士,東京都医学総合研究所の田中啓二博士,米川博通博士,多屋長治博士,正井久雄博士,共同研究で大変お世話になりました饗場篤博士,荒畑喜一博士,家村俊一郎博士,大島泰郎博士,木村澄子博士,小松雅明博士,崎村建司博士,反町典子博士,千葉智樹博士,夏目徹博士,埜中征哉博士,林由紀子博士,畠山鎮次博士,原孝彦博士,牧正敏博士,馬見塚拓博士,丸山工作博士,水島昇博士,村上誠博士,村田茂穂博士,柳田光昭博士,Siegfried Labeit博士,Jacques Beckmann博士,Henk Granzier博士,Carol Gregorio博士,Isabelle Richard博士に深謝申し上げます(紙面の都合で現所属を割愛させていただきました).そして,カルパイン研究に邁進し,共にサイエンスしてくれる(た)同志,すなわち現旧全ラボメンバーに心より感謝申し上げます.
引用文献References
1) Ono, Y., Saido, T. C., & Sorimachi, H. (2016) Nat. Rev. Drug Discov., in press.
2) Guroff, G. (1964) J. Biol. Chem., 239, 149–155.
3) Murachi, T., Tanaka, K., Hatanaka, M., & Murakami, T. (1981) Adv. Enzyme Regul., 19, 407–424.
4) Murachi, T. (1989) Biochem. Int., 18, 263–294.
5) 鈴木紘一(1991)細胞工学,10, 545–551.
6) Suzuki, K., Hayashi, H., Hayashi, T., & Iwai, K. (1983) FEBS Lett., 152, 67–70.
7) Hosfield, C.M., Elce, J.S., Davies, P.L., & Jia, Z. (1999) EMBO J., 18, 6880–6889.
8) Strobl, S., Fernandez-Catalan, C., Braun, M., Huber, R., Masumoto, H., Nakagawa, K., Irie, A., Sorimachi, H., Bourenkow, G., Bartunik, H., Suzuki, K., & Bode, W. (2000) Proc. Natl. Acad. Sci. USA, 97, 588–592.
9) Ono, Y., Iemura, S., Novak, S.M., Doi, N., Kitamura, F., Natsume, T., Gregorio, C.C., & Sorimachi, H. (2013) J. Mol. Biol., 425, 2955–2972.
10) Zhao, S., Liang, Z., Demko, V., Wilson, R., Johansen, W., Olsen, O.A., & Shalchian-Tabrizi, K. (2012) BMC Evol. Biol., 12, 193.
11) Maeda, T. (2012) FEBS J., 279, 1407–1413.
12) Maemoto, Y., Kiso, S., Shibata, H., & Maki, M. (2013) FEBS J., 280, 2594–2607.
13) Blanchard, H., Grochulski, P., Li, Y., Arthur, J.S., Davies, P.L., Elce, J.S., & Cygler, M. (1997) Nat. Struct. Biol., 4, 532–538.
14) Lin, G.D., Chattopadhyay, D., Maki, M., Wang, K.K., Carson, M., Jin, L., Yuen, P.W., Takano, E., Hatanaka, M., DeLucas, L.J., & Narayana, S.V. (1997) Nat. Struct. Biol., 4, 539–547.
15) Todd, B., Moore, D., Deivanayagam, C.C., Lin, G.D., Chattopadhyay, D., Maki, M., Wang, K.K., & Narayana, S.V. (2003) J. Mol. Biol., 328, 131–146.
16) Hata, S., Sorimachi, H., Nakagawa, K., Maeda, T., Abe, K., & Suzuki, K. (2001) FEBS Lett., 501, 111–114.
17) Hanna, R.A., Campbell, R.L., & Davies, P.L. (2008) Nature, 456, 409–412.
18) Moldoveanu, T., Gehring, K., & Green, D.R. (2008) Nature, 456, 404–408.
19) Hata, S., Abe, M., Suzuki, H., Kitamura, F., Toyama-Sorimachi, N., Abe, K., Sakimura, K., & Sorimachi, H. (2010) PLoS Genet., 6, e1001040.
20) Lee, H.J., Tomioka, S., Kinbara, K., Masumoto, H., Jeong, S.Y., Sorimachi, H., Ishiura, S., & Suzuki, K. (1999) Arch. Biochem. Biophys., 362, 22–31.
21) Hata, S., Doi, N., Kitamura, F., & Sorimachi, H. (2007) J. Biol. Chem., 282, 27847–27856.
22) Ono, Y., Kakinuma, K., Torii, F., Irie, A., Nakagawa, K., Labeit, S., Abe, K., Suzuki, K., & Sorimachi, H. (2004) J. Biol. Chem., 279, 2761–2771.
23) Moldoveanu, T., Hosfield, C.M., Lim, D., Elce, J.S., Jia, Z., & Davies, P.L. (2002) Cell, 108, 649–660.
24) Ishiura, S., Sugita, H., Suzuki, K., & Imahori, K. (1979) J. Biochem., 86, 579–581.
25) Sasaki, T., Kikuchi, T., Yumoto, N., Yoshimura, N., & Murachi, T. (1984) J. Biol. Chem., 259, 12489–12494.
26) Wang, K.K., Roufogalis, B.D., & Villalobo, A. (1989) J. Membr. Biol., 112, 233–245.
27) Takahashi, K.(1990) in Intracellular Calcium Dependent Proteolysis (Mellgren, R.L., Murachi, T., eds), pp. 571–598, CRC Press, Boca Raton, FL, USA.
28) Carafoli, E. & Molinari, M. (1998) Biochem. Biophys. Res. Commun., 247, 193–203.
29) Tompa, P., Buzder-Lantos, P., Tantos, A., Farkas, A., Szilagyi, A., Banoczi, Z., Hudecz, F., & Friedrich, P. (2004) J. Biol. Chem., 279, 20775–20785.
30) Cuerrier, D., Moldoveanu, T., & Davies, P.L. (2005) J. Biol. Chem., 280, 40632–40641.
31) Thomas, D.A., Francis, P., Smith, C., Ratcliffe, S., Ede, N.J., Kay, C., Wayne, G., Martin, S.L., Moore, K., Amour, A., & Hooper, N.M. (2006) Proteomics, 6, 2112–2120.
32) Cuerrier, D., Moldoveanu, T., Campbell, R.L., Kelly, J., Yoruk, B., Verhelst, S.H., Greenbaum, D., Bogyo, M., & Davies, P.L. (2007) J. Biol. Chem., 282, 9600–9611.
33) Shinkai-Ouchi, F., Koyama, S., Ono, Y., Hata, S., Ojima, K., Shindo, M., duVerle, D., Ueno, M., Kitamura, F., Doi, N., Takigawa, I., Mamitsuka, H., & Sorimachi, H. (2016) Mol. Cell. Proteomics, 15, 1262–1280.
34) Verspurten, J., Gevaert, K., Declercq, W., & Vandenabeele, P. (2009) Trends Biochem. Sci., 34, 319–323.
35) Liu, Z., Cao, J., Gao, X., Ma, Q., Ren, J., & Xue, Y. (2011) PLoS ONE, 6, e19001.
36) Fan, Y.X., Zhang, Y., & Shen, H.B. (2013) Proteins, 81, 622–634.
37) duVerle, D.A., Ono, Y., Sorimachi, H., & Mamitsuka, H. (2011) PLoS ONE, 6, e19035.
38) Pils, B. & Schultz, J. (2004) J. Mol. Biol., 340, 399–404.
39) Lipton, P. (1999) Physiol. Rev., 79, 1431–1568.
40) Lai, T.W., Zhang, S., & Wang, Y.T. (2014) Prog. Neurobiol., 115, 157–188.
41) Ma, M. (2013) Neurobiol. Dis., 60, 61–79.
42) Cagmat, E.B., Guingab-Cagmat, J.D., Vakulenko, A.V., Hayes, R.L., & Anagli, J. (2015) in Brain Neurotrauma: Molecular, Neuropsychological, and Rehabilitation Aspects (Kobeissy, F.H., ed), Boca Raton (FL).
43) Yokota, M., Tani, E., Tsubuki, S., Yamaura, I., Nakagaki, I., Hori, S., & Saido, T.C. (1999) Brain Res., 819, 8–14.
44) Saito, T., Matsuba, Y., Mihira, N., Takano, J., Nilsson, P., Itohara, S., Iwata, N., & Saido, T.C. (2014) Nat. Neurosci., 17, 661–663.
45) Nilsson, P., Saito, T., & Saido, T.C. (2014) ACS Chem. Neurosci., 5, 499–502.
46) Saito, T., Matsuba, Y., Yamazaki, N., Hashimoto, S., & Saido, T.C. (2016) J. Neurosci., 36, 9933–9936.
47) Toba, S., Tamura, Y., Kumamoto, K., Yamada, M., Takao, K., Hattori, S., Miyakawa, T., Kataoka, Y., Azuma, M., Hayasaka, K., Amamoto, M., Tominaga, K., Wynshaw-Boris, A., Wanibuchi, H., Oka, Y., Sato, M., Kato, M., & Hirotsune, S. (2013) Sci. Rep., 3, 1224.
48) Yamada, M., Yoshida, Y., Mori, D., Takitoh, T., Kengaku, M., Umeshima, H., Takao, K., Miyakawa, T., Sato, M., Sorimachi, H., Wynshaw-Boris, A., & Hirotsune, S. (2009) Nat. Med., 15, 1202–1207.
49) Letavernier, E., Zafrani, L., Perez, J., Letavernier, B., Haymann, J.P., & Baud, L. (2012) Cardiovasc. Res., 96, 38–45.
50) Sorimachi, H. & Ono, Y. (2012) Cardiovasc. Res., 96, 11–22.
51) Chen, M., Won, D.J., Krajewski, S., & Gottlieb, R.A. (2002) J. Biol. Chem., 277, 29181–29186.
52) Khalil, P.N., Neuhof, C., Huss, R., Pollhammer, M., Khalil, M.N., Neuhof, H., Fritz, H., & Siebeck, M. (2005) Eur. J. Pharmacol., 528, 124–131.
53) Kang, M.Y., Zhang, Y., Matkovich, S.J., Diwan, A., Chishti, A.H., & Dorn, G.W. 2nd. (2010) Circ. Res., 107, 903–912.
54) Thompson, J., Hu, Y., Lesnefsky, E.J., & Chen, Q. (2016) Am. J. Physiol. Heart Circ. Physiol., 310, H376–H384.
55) Ni, R., Zheng, D., Xiong, S., Hill, D.J., Sun, T., Gardiner, R.B., Fan, G.C., Lu, Y., Abel, E.D., Greer, P.A., & Peng, T. (2016) Diabetes, 65, 255–268.
56) Letavernier, E., Perez, J., Bellocq, A., Mesnard, L., de Castro Keller, A., Haymann, J.P., & Baud, L. (2008) Circ. Res., 102, 720–728.
57) Miyazaki, T., Taketomi, Y., Takimoto, M., Lei, X.F., Arita, S., Kim-Kaneyama, J.R., Arata, S., Ohata, H., Ota, H., Murakami, M., & Miyazaki, A. (2011) Circulation, 124, 2522–2532.
58) Subramanian, V., Uchida, H.A., Ijaz, T., Moorleghen, J.J., Howatt, D.A., & Balakrishnan, A. (2012) J. Cardiovasc. Pharmacol., 59, 66–76.
59) Galvez, A.S., Diwan, A., Odley, A.M., Hahn, H.S., Osinska, H., Melendez, J.G., Robbins, J., Lynch, R.A., Marreez, Y., & Dorn, G.W. 2nd. (2007) Circ. Res., 100, 1071–1078.
60) Wan, F., Letavernier, E., Le Saux, C.J., Houssaini, A., Abid, S., Czibik, G., Sawaki, D., Marcos, E., Dubois-Rande, J.L., Baud, L., Adnot, S., Derumeaux, G., & Gellen, B. (2015) Am. J. Physiol. Heart Circ. Physiol., 309, H1883–H1893.
61) Perez, J., Dansou, B., Herve, R., Levi, C., Tamouza, H., Vandermeersch, S., Demey-Thomas, E., Haymann, J.P., Zafrani, L., Klatzmann, D., Boissier, M.C., Letavernier, E., & Baud, L. (2016) J. Immunol., 196, 168–181.
62) Zhang, S., Meng, T., Liu, J., Zhang, X., & Zhang, J. (2015) Medicine, 94, e445.
63) Moretti, D., Del Bello, B., Allavena, G., & Maellaro, E. (2014) Arch. Biochem. Biophys., 564, 26–36.
64) Raimbourg, Q., Perez, J., Vandermeersch, S., Prignon, A., Hanouna, G., Haymann, J.P., Baud, L., & Letavernier, E. (2013) PLoS ONE, 8, e60469.
65) Miyazaki, T., Taketomi, Y., Saito, Y., Hosono, T., Lei, X.F., Kim-Kaneyama, J.R., Arata, S., Takahashi, H., Murakami, M., & Miyazaki, A. (2015) Circ. Res., 116, 1170–1181.
66) Weeraratna, A.T., Becker, D., Carr, K.M., Duray, P.H., Rosenblatt, K.P., Yang, S., Chen, Y., Bittner, M., Strausberg, R.L., Riggins, G.J., Wagner, U., Kallioniemi, O.P., Trent, J.M., Morin, P.J., & Meltzer, P.S. (2004) Oncogene, 23, 2264–2274.
67) Gollob, J.A., Sciambi, C.J., Huang, Z., & Dressman, H.K. (2005) Cancer Res., 65, 8869–8877.
68) Moretti, D., Del Bello, B., Allavena, G., Corti, A., Signorini, C., & Maellaro, E. (2015) PLoS ONE, 10, e0117258.
69) Shearer, T.R., Azuma, M., David, L.L., & Murachi, T. (1991) Invest. Ophthalmol. Vis. Sci., 32, 533–540.
70) Fukiage, C., Azuma, M., Nakamura, Y., Tamada, Y., Nakamura, M., & Shearer, T.R. (1997) Biochim. Biophys. Acta, 1361, 304–312.
71) Abell, A.D., Jones, M.A., Coxon, J.M., Morton, J.D., Aitken, S.G., McNabb, S.B., Lee, H.Y., Mehrtens, J.M., Alexander, N.A., Stuart, B.G., Neffe, A.T., & Bickerstaffe, R. (2009) Angew. Chem., 48, 1455–1458.
72) Morton, J.D., Lee, H.Y., McDermott, J.D., Robertson, L.J., Bickerstaffe, R., Jones, M.A., Coxon, J.M., & Abell, A.D. (2013) Invest. Ophthalmol. Vis. Sci., 54, 389–395.
73) Rodriguez-Muela, N., Hernandez-Pinto, A.M., Serrano-Puebla, A., Garcia-Ledo, L., Latorre, S.H., de la Rosa, E.J., & Boya, P. (2015) Cell Death Differ., 22, 476–487.
74) Shinde, V., Kotla, P., Strang, C., & Gorbatyuk, M. (2016) Cell Death Dis., 7, e2085.
75) Koriyama, Y., Ogai, K., Sugitani, K., Hisano, S., & Kato, S. (2016) Adv. Exp. Med. Biol., 854, 237–243.
76) Selsby, J., Pendrak, K., Zadel, M., Tian, Z., Pham, J., Carver, T., Acosta, P., Barton, E., & Sweeney, H.L. (2010) Am. J. Physiol. Regul. Integr. Comp. Physiol., 299, R1192–R1201.
77) Childers, M.K., Bogan, J.R., Bogan, D.J., Greiner, H., Holder, M., Grange, R.W., & Kornegay, J.N. (2011) Front. Pharmacol., 2, 89.
78) Hollinger, K. & Selsby, J.T. (2013) Acta Physiol. (Oxf.), 208, 234–244.
79) Wehling-Henricks, M., Li, Z., Lindsey, C., Wang, Y., Welc, S.S., Ramos, J.N., Khanlou, N., Kuro, O.M., & Tidball, J.G. (2016) Hum. Mol. Genet., in press.
80) Nabeshima, Y., Washida, M., Tamura, M., Maeno, A., Ohnishi, M., Shiroishi, T., Imura, A., Razzaque, M.S., & Nabeshima, Y. (2014) Sci. Rep., 4, 5847.
81) Bushby, K.M. (1999) Brain, 122, 1403–1420.
82) Fanin, M. & Angelini, C. (2015) Muscle Nerve, 52, 163–173.
83) Fanin, M., Nascimbeni, A.C., Fulizio, L., & Angelini, C. (2005) Neuromuscul. Disord., 15, 218–224.
84) Stehlikova, K., Skalova, D., Zidkova, J., Mrazova, L., Vondracek, P., Mazanec, R., Vohanka, S., Haberlova, J., Hermanova, M., Zamecnik, J., Soucek, O., Oslejskova, H., Dvorackova, N., Solarova, P., & Fajkusova, L. (2014) BMC Neurol., 14, 154.
85) Richard, I., Broux, O., Allamand, V., Fougerousse, F., Chiannilkulchai, N., Bourg, N., Brenguier, L., Devaud, C., Pasturaud, P., Roudaut, C., Hillaire, D., Passos-Bueno, M.-R., Zats, M., Tischfield, J.A., Fardeau, M., Jackson, C.E., Cohen, D., & Beckmann, J.S. (1995) Cell, 81, 27–40.
86) Ono, Y., Shimada, H., Sorimachi, H., Richard, I., Saido, T.C., Beckmann, J.S., Ishiura, S., & Suzuki, K. (1998) J. Biol. Chem., 273, 17073–17078.
87) Richard, I., Roudaut, C., Marchand, S., Baghdiguian, S., Herasse, M., Stockholm, D., Ono, Y., Suel, L., Bourg, N., Sorimachi, H., Lefranc, G., Fardeau, M., Sebille, A., & Beckmann, J.S. (2000) J. Cell Biol., 151, 1583–1590.
88) Kramerova, I., Kudryashova, E., Wu, B., Ottenheijm, C., Granzier, H., & Spencer, M.J. (2008) Hum. Mol. Genet., 17, 3271–3280.
89) Ojima, K., Ono, Y., Ottenheijm, C., Hata, S., Suzuki, H., Granzier, H., & Sorimachi, H. (2011) J. Mol. Biol., 407, 439–449.
90) Mahajan, V.B., Skeie, J.M., Bassuk, A.G., Fingert, J.H., Braun, T.A., Daggett, H.T., Folk, J.C., Sheffield, V.C., & Stone, E.M. (2012) PLoS Genet., 8, e1003001.
91) Bassuk, A.G., Yeh, S., Wu, S., Martin, D.F., Tsang, S.H., Gakhar, L., & Mahajan, V.B. (2015) PLoS ONE, 10, e0122352.
92) Dear, N., Matena, K., Vingron, M., & Boehm, T. (1997) Genomics, 45, 175–184.
93) Singh, R., Brewer, M.K., Mashburn, C.B., Lou, D., Bondada, V., Graham, B., & Geddes, J.W. (2014) J. Biol. Chem., 289, 19383–19394.
94) Wert, K.J., Bassuk, A.G., Wu, W.H., Gakhar, L., Coglan, D., Mahajan, M., Wu, S., Yang, J., Lin, C.S., Tsang, S.H., & Mahajan, V.B. (2015) Hum. Mol. Genet., 24, 4584–4598.
95) Franz, T., Winckler, L., Boehm, T., & Dear, T.N. (2004) Mol. Cell. Biol., 24, 1649–1654.
96) Kottyan, L.C., Davis, B.P., Sherrill, J.D., Liu, K., Rochman, M., Kaufman, K., Weirauch, M.T., Vaughn, S., Lazaro, S., Rupert, A.M., Kohram, M., Stucke, E.M., Kemme, K.A., Magnusen, A., He, H., Dexheimer, P., Chehade, M., Wood, R.A., Pesek, R.D., Vickery, B.P., Fleischer, D.M., Lindbad, R., Sampson, H.A., Mukkada, V.A., Putnam, P.E., Abonia, J.P., Martin, L.J., Harley, J.B., & Rothenberg, M.E. (2014) Nat. Genet., 46, 895–900.
97) Sleiman, P.M., Wang, M.L., Cianferoni, A., Aceves, S., Gonsalves, N., Nadeau, K., Bredenoord, A.J., Furuta, G.T., Spergel, J.M., & Hakonarson, H. (2014) Nat. Commun., 5, 5593.
98) Dear, T.N. & Boehm, T. (2001) Gene, 274, 245–252.
99) Ueta, M., Mizushima, K., Yokoi, N., Naito, Y., & Kinoshita, S. (2010) Br. J. Ophthalmol., 94, 1239–1243.
100) Ueta, M., Sotozono, C., & Kinoshita, S. (2011) Jpn. J. Ophthalmol., 55, 405–410.
101) Sorimachi, H., Ishiura, S., & Suzuki, K. (1993) J. Biol. Chem., 268, 19476–19482.
102) Liu, K., Li, L., & Cohen, S.N. (2000) J. Biol. Chem., 275, 31093–31098.
103) Gan-Or, Z., Bouslam, N., Birouk, N., Lissouba, A., Chambers, D.B., Veriepe, J., Androschuck, A., Laurent, S.B., Rochefort, D., Spiegelman, D., Dionne-Laporte, A., Szuto, A., Liao, M., Figlewicz, D.A., Bouhouche, A., Benomar, A., Yahyaoui, M., Ouazzani, R., Yoon, G., Dupre, N., Suchowersky, O., Bolduc, F.V., Parker, J.A., Dion, P.A., Drapeau, P., Rouleau, G.A., & Bencheikh, B.O. (2016) Am. J. Hum. Genet., 98, 1038–1046.
104) Forman, O.P., De Risio, L., & Mellersh, C.S. (2013) PLoS ONE, 8, e64627.
105) Wang, Y., Hersheson, J., Lopez, D., Hammer, M., Liu, Y., Lee, K.H., Pinto, V., Seinfeld, J., Wiethoff, S., Sun, J., Amouri, R., Hentati, F., Baudry, N., Tran, J., Singleton, A.B., Coutelier, M., Brice, A., Stevanin, G., Durr, A., Bi, X., Houlden, H., & Baudry, M. (2016) Cell Rep., 16, 79–91.
106) Hotez, P.J., Pecoul, B., Rijal, S., Boehme, C., Aksoy, S., Malecela, M., Tapia-Conyer, R., & Reeder, J.C. (2016) PLoS Negl. Trop. Dis., 10, e0003895.
107) Russo, I., Oksman, A., Vaupel, B., & Goldberg, D.E. (2009) Proc. Natl. Acad. Sci. USA, 106, 1554–1559.
108) Rosenthal, P.J. (2011) Adv. Exp. Med. Biol., 712, 30–48.
109) Chandramohanadas, R., Davis, P.H., Beiting, D.P., Harbut, M.B., Darling, C., Velmourougane, G., Lee, M.Y., Greer, P.A., Roos, D.S., & Greenbaum, D.C. (2009) Science, 324, 794–797.
110) Hanspal, M., Goel, V.K., Oh, S.S., & Chishti, A.H. (2002) Mol. Biochem. Parasitol., 122, 227–229.
111) Berthier, D., Breniere, S.F., Bras-Goncalves, R., Lemesre, J.L., Jamonneau, V., Solano, P., Lejon, V., Thevenon, S., & Bucheton, B. (2016) Trends Parasitol., 32, 157–168.
112) Ersfeld, K., Barraclough, H., & Gull, K. (2005) J. Mol. Evol., 61, 742–757.
113) Hayes, P., Varga, V., Olego-Fernandez, S., Sunter, J., Ginger, M.L., & Gull, K. (2014) J. Cell Biol., 206, 377–384.
114) Cai, P., Gobert, G.N., You, H., & McManus, D.P. (2016) Int. J. Parasitol., 46, 453–463.
115) Sorimachi, H., Hata, S., & Ono, Y. (2011) Proc. Jpn. Acad., Ser. B, Phys. Biol. Sci., 87, 287–327.
116) Davis, D., Wilson, R.B., & Mitchell, A.P. (2000) Mol. Cell. Biol., 20, 971–978.
117) Li, M., Martin, S.J., Bruno, V.M., Mitchell, A.P., & Davis, D.A. (2004) Eukaryot. Cell, 3, 741–751.
118) Ost, K.S., O’Meara, T.R., Huda, N., Esher, S.K., & Alspaugh, J.A. (2015) PLoS Genet., 11, e1005159.
119) Futai, E., Maeda, T., Sorimachi, H., Kitamoto, K., Ishiura, S., & Suzuki, K. (1999) Mol. Gen. Genet., 260, 559–568.
120) Diez, E., Alvaro, J., Espeso, E.A., Rainbow, L., Suarez, T., Tilburn, J., Arst, H.N. Jr., & Penalva, M.A. (2002) EMBO J., 21, 1350–1359.
121) Hayashi, M., Fukuzawa, T., Sorimachi, H., & Maeda, T. (2005) Mol. Cell. Biol., 25, 9478–9490.
122) Rawlings, N.D. (2015) Biol. Direct, 10, 66.
123) Staniec, D., Ksiazek, M., Thogersen, I.B., Enghild, J.J., Sroka, A., Bryzek, D., Bogyo, M., Abrahamson, M., & Potempa, J. (2015) J. Biol. Chem., 290, 27248–27260.
124) Aoyagi, T., Takeuchi, T., Matsuzaki, A., Kawamura, K., Kondo, S., Hamada, M., Maeda, K., & Umezawa, H. (1969) J. Antibiot., 22, 283–286.
125) Hanada, K., Tamai, M., Ohmura, S., Sawada, J., Seki, T., & Tanaka, I. (1978) Agric. Biol. Chem., 42, 523–528.
126) Hashida, S., Towatari, T., Kominami, E., & Katunuma, N. (1980) J. Biochem., 88, 1805–1811.
127) Ali, M.A., Stepanko, A., Fan, X., Holt, A., & Schulz, R. (2012) Biochem. Biophys. Res. Commun., 423, 1–5.
128) Wang, K.K., Nath, R., Posner, A., Raser, K.J., Buroker-Kilgore, M., Hajimohammadreza, I., Probert, A.W. Jr., Marcoux, F.W., Ye, Q., Takano, E., Hatanaka, M., Maki, M., Caner, H., Collins, J.L., Fergus, A., Lee, K.S., Lunney, E.A., Hays, S.J., & Yuen, P. (1996) Proc. Natl. Acad. Sci. USA, 93, 6687–6692.
129) Jo, H., Meinhardt, N., Wu, Y., Kulkarni, S., Hu, X., Low, K.E., Davies, P.L., DeGrado, W.F., & Greenbaum, D.C. (2012) J. Am. Chem. Soc., 134, 17704–17713.
130) Chen, H., Jiao, W., Jones, M.A., Coxon, J.M., Morton, J.D., Bickerstaffe, R., Pehere, A.D., Zvarec, O., & Abell, A.D. (2012) Chem. Biodivers., 9, 2473–2484.
131) Jones, S.A., Neilsen, P.M., Siew, L., Callen, D.F., Goldfarb, N.E., Dunn, B.M., & Abell, A.D. (2013) ChemMedChem, 8, 1918–1921.
132) Ozaki, T., Nakazawa, M., Yamashita, T., & Ishiguro, S. (2015) PLoS ONE, 10, e0130986.
133) Murata, M., Miyashita, S., Yokoo, C., Tamai, M., Hanada, K., Hatayama, K., Towatari, T., Nikawa, T., & Katunuma, N. (1991) FEBS Lett., 280, 307–310.
134) Duong, L.T., Leung, A.T., & Langdahl, B. (2016) Calcif. Tissue Int., 98, 381–397.
135) Katunuma, N. (2011) Proc. Jpn. Acad., Ser. B, Phys. Biol. Sci., 87, 29–39.
136) Low, K.E., Karunan Partha, S., Davies, P.L., & Campbell, R.L. (2014) Biochim. Biophys. Acta, 1840, 3367–3373.
137) Adams, S.E., Robinson, E.J., Miller, D.J., Rizkallah, P.J., Hallett, M.B., & Allemann, R.K. (2015) Chem. Sci. (Camb.), 6, 6865–6871.
138) Novinec, M., Korenc, M., Caflisch, A., Ranganathan, R., Lenarcic, B., & Baici, A. (2014) Nat. Commun., 5, 3287.
139) Tonami, K., Hata, S., Ojima, K., Ono, Y., Kurihara, Y., Amano, T., Sato, T., Kawamura, Y., Kurihara, H., & Sorimachi, H. (2013) PLoS Genet., 9, e1003668.
140) Bartoli, M., Poupiot, J., Vulin, A., Fougerousse, F., Arandel, L., Daniele, N., Roudaut, C., Noulet, F., Garcia, L., Danos, O., & Richard, I. (2007) Gene Ther., 14, 733–740.
141) Mendell, J.R., Sahenk, Z., Malik, V., Gomez, A.M., Flanigan, K.M., Lowes, L.P., Alfano, L.N., Berry, K., Meadows, E., Lewis, S., Braun, L., Shontz, K., Rouhana, M., Clark, K.R., Rosales, X.Q., Al-Zaidy, S., Govoni, A., Rodino-Klapac, L.R., Hogan, M.J., & Kaspar, B.K. (2015) Mol. Ther., 23, 192–201.
142) Miyazaki, T., Tonami, K., Hata, S., Aiuchi, T., Ohnishi, K., Lei, X.F., Kim-Kaneyama, J.R., Takeya, M., Itabe, H., Sorimachi, H., Kurihara, H., & Miyazaki, A. (2016) J. Clin. Invest., 126, 3417–3432.
143) Olaya, P. & Wasserman, M. (1991) Biochim. Biophys. Acta, 1096, 217–221.
144) Li, X., Chen, H., Jeong, J.J., & Chishti, A.H. (2007) Mol. Biochem. Parasitol., 155, 26–32.
145) El Chamy Maluf, S., Melo, P.M., Varotti, F.P., Gazarini, M.L., Cunha, R.L., & Carmona, A.K. (2016) Parasitol. Int., 65, 20–22.
146) Soh, B.Y., Song, H.O., Lee, Y., Lee, J., Kaewintajuk, K., Lee, B., Choi, Y.Y., Cho, J.H., Choi, S., & Park, H. (2013) Malar. J., 12, 47.
147) Marinho, F.A., Goncalves, K.C., Oliveira, S.S., Goncalves, D.S., Matteoli, F.P., Seabra, S.H., Oliveira, A.C., Bellio, M., Oliveira, S.S., Souto-Padron, T., d’Avila-Levy, C.M., Santos, A.L., & Branquinha, M.H. (2014) PLoS ONE, 9, e87659.
148) Sangenito, L.S., Menna-Barreto, R.F., CM, D.A.-L., Santos, A.L., & Branquinha, M.H. (2014) PLoS ONE, 9, e113957.
149) Andresen, K., Tom, T.D., & Strand, M. (1991) J. Biol. Chem., 266, 15085–15090.
150) Karmakar, S., Zhang, W., Ahmad, G., Torben, W., Alam, M.U., Le, L., Damian, R.T., Wolf, R.F., White, G.L., Carey, D.W., Carter, D., Reed, S.G., & Siddiqui, A.A. (2014) J. Infect. Dis., 209, 1929–1940.
151) Karmakar, S., Zhang, W., Ahmad, G., Torben, W., Alam, M.U., Le, L., Damian, R.T., Wolf, R.F., White, G.L., Carey, D.W., Carter, D., Reed, S.G., & Siddiqui, A.A. (2014) Vaccine, 32, 1296–1303.
152) Bartoli, M., Roudaut, C., Martin, S., Fougerousse, F., Suel, L., Poupiot, J., Gicquel, E., Noulet, F., Danos, O., & Richard, I. (2006) Mol. Ther., 13, 250–259.
153) Roudaut, C., Le Roy, F., Suel, L., Poupiot, J., Charton, K., Bartoli, M., & Richard, I. (2013) Circulation, 128, 1094–1104.
154) Li, H.L., Fujimoto, N., Sasakawa, N., Shirai, S., Ohkame, T., Sakuma, T., Tanaka, M., Amano, N., Watanabe, A., Sakurai, H., Yamamoto, T., Yamanaka, S., & Hotta, A. (2015) Stem Cell Rep., 4, 143–154.
155) Ono, Y., Shindo, M., Doi, N., Kitamura, F., Gregorio, C.C., & Sorimachi, H. (2014) Proc. Natl. Acad. Sci. USA, 111, E5527–E5536.
156) Hall, M.R. & Gibson, W. (1996) J. Virol., 70, 5395–5404.
157) Hall, M.R. & Gibson, W. (1997) Virology, 227, 160–167.
158) Ermolova, N., Kudryashova, E., DiFranco, M., Vergara, J., Kramerova, I., & Spencer, M.J. (2011) Hum. Mol. Genet., 20, 3331–3345.
159) Drag, M. & Salvesen, G.S. (2010) Nat. Rev. Drug Discov., 9, 690–701.
160) Baier, L.J., Permana, P.A., Yang, X., Pratley, R.E., Hanson, R.L., Shen, G.Q., Mott, D., Knowler, W.C., Cox, N.J., Horikawa, Y., Oda, N., Bell, G.I., & Bogardus, C. (2000) J. Clin. Invest., 106, R69–R73.
161) Horikawa, Y., Oda, N., Cox, N.J., Li, X., Orho-Melander, M., Hara, M., Hinokio, Y., Lindner, T.H., Mashima, H., Schwarz, P.E., del Bosque-Plata, L., Horikawa, Y., Oda, Y., Yoshiuchi, I., Colilla, S., Polonsky, K.S., Wei, S., Concannon, P., Iwasaki, N., Schulze, J., Baier, L.J., Bogardus, C., Groop, L., Boerwinkle, E., Hanis, C.L., & Bell, G.I. (2000) Nat. Genet., 26, 163–175.
162) Urbanek, M. (2007) Nat. Clin. Pract. Endocrinol. Metab., 3, 103–111.
163) Siklos, M., BenAissa, M., & Thatcher, G.R. (2015) Acta Pharm. Sin. B, 5, 506–519.
164) Azam, M., Andrabi, S.S., Sahr, K.E., Kamath, L., Kuliopulos, A., & Chishti, A.H. (2001) Mol. Cell. Biol., 21, 2213–2220.
165) Dutt, P., Croall, D.E., Arthur, S.C., De Veyra, T., Williams, K., Elce, J.S., & Greer, P.A. (2006) BMC Dev. Biol., 6, 3.
166) Takano, J., Mihira, N., Fujioka, R., Hosoki, E., Chishti, A.H., & Saido, T.C. (2011) Mol. Cell. Biol., 31, 4097–4106.
167) Arthur, J.S., Elce, J.S., Hegadorn, C., Williams, K., & Greer, P.A. (2000) Mol. Cell. Biol., 20, 4474–4481.
168) Zimmerman, U.J., Boring, L., Pak, J.H., Mukerjee, N., & Wang, K.K. (2000) IUBMB Life, 50, 63–68.
169) Kramerova, I., Kudryashova, E., Tidball, J.G., & Spencer, M.J. (2004) Hum. Mol. Genet., 13, 1373–1388.
170) Ojima, K., Kawabata, Y., Nakao, H., Nakao, K., Doi, N., Kitamura, F., Ono, Y., Hata, S., Suzuki, H., Kawahara, H., Bogomolovas, J., Witt, C., Ottenheijm, C., Labeit, S., Granzier, H., Toyama-Sorimachi, N., Sorimachi, M., Suzuki, K., Maeda, T., Abe, K., Aiba, A., & Sorimachi, H. (2010) J. Clin. Invest., 120, 2672–2683.
171) Li, M., Wang, X., Yu, Y., Yu, Y., Xie, Y., Zou, Y., Ge, J., Peng, T., & Chen, R. (2014) Virus Res., 179, 177–186.
172) Kaneko, Y., Murphy, C.R., & Day, M.L. (2014) Histochem. Cell Biol., 141, 423–430.
173) Sebe, J.Y., Bershteyn, M., Hirotsune, S., Wynshaw-Boris, A., & Baraban, S.C. (2013) J. Neurophysiol., 109, 429–436.
174) Mizukoshi, S., Nakazawa, M., Sato, K., Ozaki, T., Metoki, T., & Ishiguro, S. (2010) Exp. Eye Res., 91, 353–361.
175) Chen, Y., Li, Z., He, Y., Shang, D., Pan, J., Wang, H., Chen, H., Zhu, Z., Wan, L., & Wang, X. (2014) Toxicol. Appl. Pharmacol., 275, 176–181.
176) Samantaray, S., Knaryan, V.H., Shields, D.C., Cox, A.A., Haque, A., & Banik, N.L. (2015) Mol. Neurobiol., 52, 1054–1066.
177) Guyton, M.K., Das, A., Samantaray, S., Wallace, G.C. 4th, Butler, J.T., Ray, S.K., & Banik, N.L. (2010) J. Neurosci. Res., 88, 2398–2408.
178) Das, A., Guyton, M.K., Smith, A., Wallace, G. 4th, McDowell, M.L., Matzelle, D.D., Ray, S.K., & Banik, N.L. (2013) J. Neurochem., 124, 133–146.
179) Nozaki, K., Das, A., Ray, S.K., & Banik, N.L. (2011) J. Neurosci. Res., 89, 536–543.
180) Tabata, C., Tabata, R., & Nakano, T. (2010) Clin. Exp. Immunol., 162, 560–567.
181) Del Bello, B., Toscano, M., Moretti, D., & Maellaro, E. (2013) PLoS ONE, 8, e57236.
182) Guo, A., Hall, D., Zhang, C., Peng, T., Miller, J.D., Kutschke, W., Grueter, C.E., Johnson, F.L., Lin, R.Z., & Song, L.S. (2015) J. Biol. Chem., 290, 17946–17955.
183) Undrovinas, A., Maltsev, V.A., & Sabbah, H.N. (2013) PLOS ONE, 8, e54436.
184) Ennes-Vidal, V., Menna-Barreto, R.F., Santos, A.L., Branquinha, M.H., & d’Avila-Levy, C.M. (2011) PLoS ONE, 6, e18371.
185) Huang, Q., Chen, H., Wang, F., Brost, B.C., Li, J., Gao, Y., Li, Z., Gao, Y., & Jiang, S.W. (2014) Cell. Mol. Life Sci., 71, 3151–3164.
186) Brocard, C., Plantier, V., Boulenguez, P., Liabeuf, S., Bouhadfane, M., Viallat-Lieutaud, A., Vinay, L., & Brocard, F. (2016) Nat. Med., 22, 404–411.
187) Paquet-Durand, F., Sanges, D., McCall, J., Silva, J., van Veen, T., Marigo, V., & Ekstrom, P. (2010) J. Neurochem., 115, 930–940.
188) Trager, N., Smith, A., Wallace Iv, G., Azuma, M., Inoue, J., Beeson, C., Haque, A., & Banik, N.L. (2014) J. Neurochem., 130, 268–279.
189) Suzuki, R., Oka, T., Tamada, Y., Shearer, T.R., & Azuma, M. (2014) J. Ocul. Pharmacol. Ther., 30, 419–428.
190) Takeshita, D., Tanaka, M., Mitsuyama, S., Yoshikawa, Y., Zhang, G.X., Obata, K., Ito, H., Taniguchi, S., & Takaki, M. (2013) J. Physiol. Sci., 63, 113–123.
191) Bains, M., Cebak, J.E., Gilmer, L.K., Barnes, C.C., Thompson, S.N., Geddes, J.W., & Hall, E.D. (2012) J. Neurochem., 125, 125–132.
192) Simoes, A.T., Goncalves, N., Nobre, R.J., Duarte, C.B., & Pereira de Almeida, L. (2014) Hum. Mol. Genet., 23, 4932–4944.
193) De Franceschi, L., Franco, R.S., Bertoldi, M., Brugnara, C., Matte, A., Siciliano, A., Wieschhaus, A.J., Chishti, A.H., & Joiner, C.H. (2013) FASEB J., 27, 750–759.
194) Ozaki, T., Ishiguro, S., Hirano, S., Baba, A., Yamashita, T., Tomita, H., & Nakazawa, M. (2013) PLoS ONE, 8, e71650.
195) Mallik, S.K., Li, Y., Cui, M., Song, H.O., Park, H., & Kim, H.S. (2012) Arch. Pharm. Res., 35, 469–479.
196) Bochner, R., Samuelov, L., Sarig, O., Li, Q., Adase, C. A., Isakov, O., Malchin, N., Vodo, D., Shayevitch, R., Peled, A., Yu, B. D., Fainberg, G., Warshauer, E., Adir, N., Erez, N., Gat, A., Gottlieb, Y., Rogers, T., Pavlovsky, M., Goldberg, I., Shomron, N., Sandilands, A., Campbell, L. E., MacCallum, S., McLean, W. H., Ast, G., Gallo, R. L., Uitto, J., & Sprecher, E. (2016) J. Invest. Dermatol., in press.
著者紹介Author Profile
 反町 洋之(そりまち ひろゆき)
反町 洋之(そりまち ひろゆき)公益財団法人東京都医学総合研究所カルパインプロジェクトプロジェクトリーダー,参事研究員.博士(農学).
略歴1963年神奈川県に生る.86年東大理学部卒.88年同大学院理学系研究科修士修了.88~92年都臨床研研究員.92~97年東大分生研助手.97~2004年東大農学部助教授.04~11年都臨床研副参事(09年~参事)研究員.11年~現職.
研究テーマと抱負研究テーマはカルパイン.抱負は,忙中閑あり,苦中楽あり.また,中学のバスケ部で流行った「死んでも命」というフレーズは,意味不明ながら,時々思い出しては気合いを入れています.
ウェブサイトhttp://www.igakuken.or.jp/calpain/
趣味ワイン,ビール,カメラ,車,野球,水泳,黒子のバスケ,攻殻機動隊,ガリレオ,1Q84,奥様は魔女,カルパイン.
 小野 弥子(おの やすこ)
小野 弥子(おの やすこ)公益財団法人東京都医学総合研究所カルパインプロジェクト副参事研究員.博士(理学).
略歴大分県出身.99年東大大学院理学系研究科博士修了.99~2003年日本学術振興会研究員(東大分生研・アリゾナ大).04~11年都臨床研主任(08年~主席)研究員.11~都医学研主席研究員.16年~現職.
研究テーマカルパイン.
研究テーマと抱負Be curious and critical, but also respect the accomplishments of others(どこかで読んだ).「自分に厳しく」「自分大好き」のバランスを取って,シャキッとする.
ウェブサイトhttp://www.igakuken.or.jp/calpain/
趣味バレエ,語学番組,ラジオ,Percy Jackson & the Olympians,ハリーポッター,カルパイン.