スフィンゴ糖脂質(glycosphingolipid:GSL)は,疎水性のセラミド部分と親水性の糖鎖部分からなり1),これまでに糖鎖構造の違いから400種類以上が同定されている2).また,セラミド部分についても脂肪酸鎖長に豊富なバリエーションがある3).GSLの種類とその代謝系は,細胞の種類のみならず,細胞の増殖や分化段階に応じて,特異的であることから2, 4),GSLは細胞機能に関与していると考えられている.このようなGSLの細胞機能を特徴づける最も重要な性質は,それらが細胞膜の外側の層で互いに凝集(クラスター化)することでドメインを形成し,そのようなドメインに存在する膜タンパク質や細胞内に局在するシグナル伝達分子と会合することで,脂質ラフトと呼ばれる機能的な膜マイクロドメインを形成することである5–8).
脂質ラフトにおけるGSLの役割として,脂質ラフトに会合する受容体タンパク質にリガンドが結合する情報を細胞内に伝えるのをサポートする機能と,GSLが直接リガンドとトランスに結合することにより脂質ラフトのドメインを介して細胞内に情報を伝達する機能がある.前者の例としては,ガングリオシドGM3が脂肪細胞におけるインスリン受容体の機能9)や,上皮成長因子受容体(EGFR)の機能10, 11)を制御することが知られている.また,神経細胞に最も豊富に存在するGM1はニューロトロフィン受容体と会合し12),ラミニン-1により誘導されるTrkAとβ1インテグリンを介した神経突起伸長を制御している13).これらの知見は,脂質ラフトのGSLと受容体とのシス相互作用が,受容体を介した細胞機能を調節していることを示すものである.一方後者の例として,中性のGSLであるラクトシルセラミド(LacCer, CDw17)のヒト好中球における機能がある.LacCerは大腸菌,百日咳菌,赤痢菌,ピロリ菌などのさまざまな細菌やカンジダなどの真菌,ウイルスに結合することが報告されており14–20),LacCerが微生物と宿主との相互作用に関与すると考えられている.LacCerは好中球のGSLの約70%を占め,その1割程度は細胞膜表面に発現している21–23).好中球は細胞膜でSrcファミリーキナーゼのLynなどの膜結合型の細胞内情報伝達分子と会合したLacCerの脂質ラフトを形成し,抗LacCer抗体によりLynに依存した活性酸素を産生する24).これらは,好中球にLacCerの脂質ラフトを介して微生物を殺す仕組みがあることを示唆している.
本稿では,GSLの脂質ラフトの構造と細胞機能発現におけるGSLの糖鎖と脂肪酸鎖の役割について,LacCerの脂質ラフトに関する我々の研究成果を中心に概説する.
脂質二重層からなる生体膜は,リン脂質とコレステロールの海にスフィンゴ脂質や膜タンパク質が漂っているようにたとえられる.各構成分子の物理化学的な性質に従って,これらは膜上に不均一に分布するとともに,特定の分子群が側方向の分子間相互作用によって安定化された超分子複合体のドメインを形成する.GSLは細胞膜の外側の層に特異的に発現しており25),その物理化学的性状,セラミド部分の水酸基とアセトアミド構造から生じる水素結合や炭化水素鎖によるファンデルワールス力に基づく側方向相互作用によって細胞膜上でクラスターを形成することができる4).そのため,前に述べたようにGSLのクラスターは細胞膜でさまざまな分子と会合して脂質ラフトと呼ばれるドメインを形成している26, 27).脂質ラフトとは「GSLとコレステロールに富み,GPIアンカー型タンパク質や脂質を介して細胞膜にアンカリングされた細胞内情報伝達分子を含む膜ドメインである」と定義されている28, 29).しかしながら,脂質ラフトの実際の存在状態についてはいまだに不明な点が多い.その理由は,脂質分子を主成分とするドメインであることから,脂質ラフトをタンパク質のように生化学的に分離精製し,ありのままの状態で可視化することが大変難しいためである.そこで,電子顕微鏡や1分子蛍光標識法などのさまざまな手法で脂質ラフトの構造を可視化する試みが行われている.しかしながら,糖鎖や脂質部分を蛍光標識した分子がもともと存在する非標識分子と同じ細胞内挙動をすることを証明している例はいまだにわずかしかない30).GSLに着目して観察した場合の脂質ラフトが直径50~100 nmのドメインである31, 32)ことは,1分子蛍光標識したGPIアンカー型糖タンパク質を用いて観察した場合の脂質ラフトが10 nm程度の動的なドメインとして観察されることと対照的である33, 34).この違いは観察手法の違いにもよるが,観察に用いる分子の物性によっても大きく左右される.GPIアンカー型タンパク質は,ホスファチジルイノシトールにタンパク質が結合した分子であり,脂質部分は側方向相互作用をする力が弱く,脂質部分の分子サイズに比べてタンパク質のサイズは非常に大きい.一方,GSLは糖鎖部分とセラミド部分のサイズがほとんど同じであり,中性のGSLの融点が65°C以上であるように35),側方向相互作用の力が大変強く細胞膜でも互いにパックされやすい.中性GSLよりも融点がずっと低い酸性GSLであるガングリオシドを用いて再構成した脂質ドメインにおいても,相転移温度は40°C程度であり,体温で液体として存在するリン脂質の膜上では独立したドメインとして存在しうることを示唆している36).また,GM1とGM3あるいはGM3とLacCerといった糖鎖構造のわずかな違いでも,同じ細胞において異なる脂質ラフトを構成し,異なる機能に関与しうることが示されている24, 32).
抗糖脂質抗体が病因に関わるといわれるギラン・バレー症候群の患者血清中の抗GM1抗体の特異性が病態によって異なることが知られている37).同じGSLに対するモノクローナル抗体でも免疫沈降やTLCブロットができる抗体とできない抗体がある.糖脂質がPVDF膜上でさまざまな立体構造をとることから,TLCブロットができない抗GSL抗体は特定の立体構造を認識すると考えられる.これらのことは,同じGSLを認識する単一のエピトープを持つモノクローナル抗体であっても,GSLが細胞膜上で作り出す脂質ラフトの立体構造の違いによって抗体の抗原特異性が異なることを示唆している.
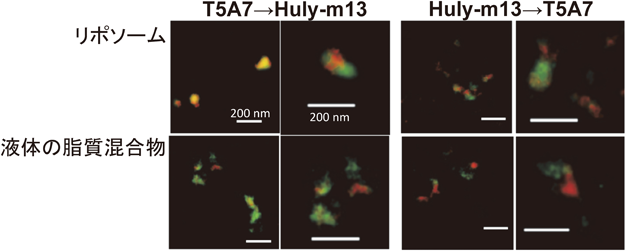
同じマウス抗LacCerモノクローナルIgMであるT5A7とHuly-m13は,TLCブロットや免疫沈降活性が異なっている.LacCerはヒト好中球の細胞膜上で脂質ラフトを形成し,遊走・貪食・活性酸素産生に関与している18, 24, 38).T5A7は遊走や活性酸素産生を誘導するが,Triton X-100で可溶化した膜から得られるLacCerの脂質ラフト画分を免疫沈降することはできない24).一方で,Huly-m13は生物活性こそT5A7に比べて弱いが,LacCerの脂質ラフト画分を免疫沈降し,TLCブロットでLacCerと選択的に結合できる.マウス好中球はヒトに比べて細胞膜表面でのLacCerの発現量が5%程度であり,フローサイトメトリーでT5A7陽性であるが,Huly-m13は陰性である39).また,T5A7はマウス好中球の遊走を惹起し,マウス好中球の貪食を抑制する.一方,Huly-m13はマウス好中球のそれらの機能に影響しない.これらの結果は,マウス好中球にはT5A7では認識できるがHuly-m13では認識できないLacCerの脂質ラフトが存在することを示唆している.細胞膜の脂質ラフトを再構成したリポソームをコートしたプレートと,液体の脂質混合物をコートしたプレートを用いたELISAを行うと,T5A7がLacCerと結合するためにはリン脂質の存在が必要であるが,Huly-m13はLacCerのみを認識することが示される39).また,これら抗体の反応性はLacCerクラスターの密度や糖鎖の並び方,あるいはCaイオンには影響されない.さらに,超解像顕微鏡で抗体の結合するLacCerのドメインを観察すると,T5A7はLacCerのドメインの辺縁部を強く認識し,Huly-m13はLacCerのドメインの中心により結合しやすいことがわかる(図1).これら一連の結果は,細胞膜上のGSLの脂質ラフトにおいて,GSLが作り出す立体構造は均一ではなく,同じGSLに対して異なる抗原特異性を示すモノクローナル抗体ができることを示している.LacCerに対する自己抗体によって全身の神経が相次いで障害される脳脊髄根末梢神経炎(encephalo-myelo-radiculo-neuropathy)の病態にこのような抗LacCer抗体の結合特性が反映されているのかもしれない40).
3. GSLの脂質ラフトの情報伝達における脂肪酸鎖の役割
GSLとスフィンゴミエリン(SM)は,同じセラミド構造を持つスフィンゴ脂質であるが,同じ細胞であってもセラミドに含まれる脂肪酸鎖の種類は両者で大きく異なっている31).哺乳類のセラミドは,6種類のセラミド合成酵素によって異なる長さの脂肪酸鎖がスフィンゴシンに結合することで生合成される41).SMは炭素数16の飽和脂肪酸であるパルミチン酸と,炭素数24の飽和脂肪酸であるリグノセリン酸および不飽和脂肪酸であるネルボン酸が主な脂肪酸である.一方,GSLのセラミドに含まれる脂肪酸は多様であり,細胞のみならず組織や臓器によっても大きく異なる41).これは,脂肪酸の長さによってセラミド合成酵素が異なっており,それら酵素の発現レベルが細胞種によって異なることに由来する.このことは,GSLとSMが同じスフィンゴ脂質であってもその役割が異なっていること,特に脂肪酸鎖の構造が生物学的に重要な意味を持っていることを示唆しているが,今のところGSLとSMのセラミド生合成経路がどのように異なっているかの詳細はよくわかっていない.
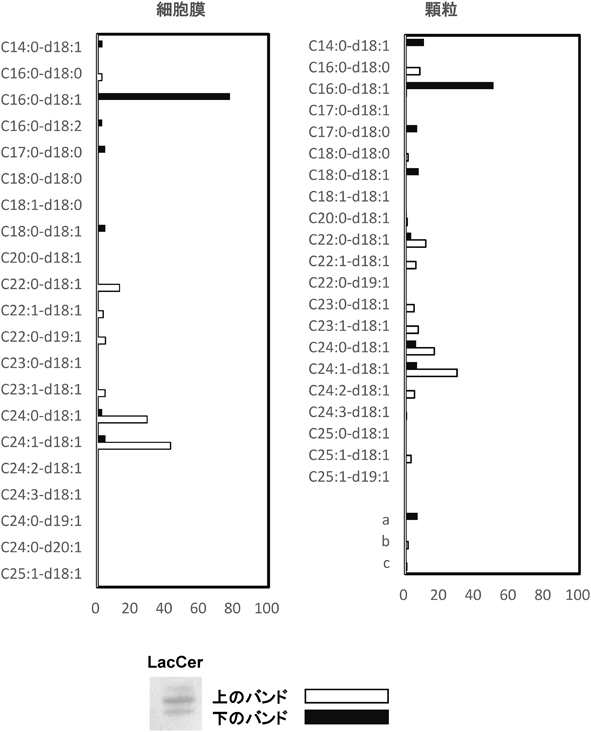
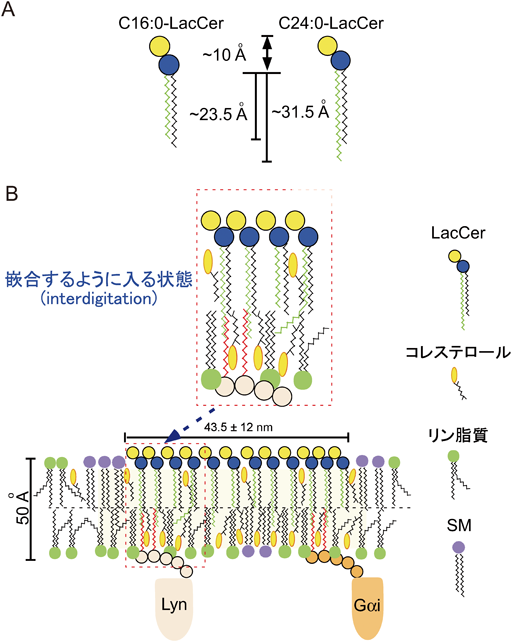
GSLの脂質ラフトがさまざまな膜機能に関与することが示されているが2, 7, 24),どのようにしてGSLが細胞内の情報伝達分子を活性化できるかは長らく不明であった.我々はGSLが細胞内へと情報を伝える仕組みの一端を解き明かす現象を好中球系に分化させたヒト急性骨髄性白血病細胞HL-60で見いだした24).LacCerの脂質ラフトを介した好中球の活性酸素産生はLacCerの脂質ラフトに会合したLynに依存している.しかしながら,DMSOで好中球系細胞に分化したHL-60(D-HL-60)は,LacCerを細胞膜に発現し,活性酸素産生能を獲得しているにも関わらず,抗LacCer抗体で活性酸素を産生できない.興味深いことに,D-HL-60細胞の細胞膜上にはLynと会合したLacCerの脂質ラフトはほとんど存在しないことから,D-HL-60細胞ではLacCerの脂質ラフトの構造に異常があるために,LacCerからLynへと情報が伝えられず,LacCerを介した活性酸素の産生が起こらないと考えられた.そこで,好中球とD-HL-60細胞のLacCerの脂質ラフトに含まれる脂質分子種を解析すると,D-HL-60の細胞膜ではリグノセリン酸とネルボン酸を含むLacCer(C24 : 0-, C24 : 1-LacCer)がヒト好中球に比べ極端に少なかった31).好中球のLacCerは細胞膜に全体の10%,顆粒膜に90%が存在し,分子種組成は細胞膜と顆粒膜で異なっている(図2).細胞膜での主要な分子種であるC16 : 0, C22 : 0, C24 : 0およびC24 : 1の脂肪酸鎖を含むLacCerを合成し,D-HL-60に加えると,C24 : 0-LacCerとC24 : 1-LacCerを取り込ませた場合のみ細胞膜上でLynと会合したLacCerの脂質ラフトが再構成され,抗LacCer抗体でLynがリン酸化され,D-HL-60はLacCerの脂質ラフトを介した活性酸素産生や遊走を引き起こすようになる31).さらに,オプソニン化された菌のみを貪食するD-HL-60細胞が,C24-LacCerを取り込ませることで非オプソニン条件下での貪食能を回復する38).このことは,LacCerを介した細胞機能にはC24の脂肪酸鎖を含むセラミド構造が重要な働きをしていること,特にC24-LacCerの脂質ラフトが好中球の非オプソニン化条件での貪食に必須であることを示している.脂質二重層の人工膜を用いた研究から,上層にあるC24-LacCerの脂肪酸鎖が下層のリン脂質脂肪酸鎖に嵌合(interdigitation)することで,下層の膜流動性を変化させることが示されている42, 43).さらに,示差走査熱量測定とLangmuir薄膜バランスの実験からC24 : 0-LacCerだけではなく,C24 : 1-LacCerによってもパックされたクラスターを形成することが示されている44).脂質ラフトに会合する情報伝達分子の多くはパルミトイル化等で脂肪酸鎖が付加されることで,細胞膜親和性を持つようになる.そこで,C16 : 0-LacCerとC24 : 0-LacCerそれぞれの脂肪酸鎖と同じ長さになるように脂肪酸鎖に光架橋試薬を結合させた3H標識LacCer誘導体を作製した.それらをD-HL-60細胞に取り込ませて,光架橋された分子を分離すると,Lynと三量体Gタンパク質であるGαiがC24 : 0-LacCer誘導体を取り込ませた場合でのみ回収された45).Srcファミリーキナーゼは互いに凝集することで自己リン酸化され細胞内へと情報を伝える46).また,LacCerを介した好中球の遊走は三量体Gタンパク質に依存している18).したがって,LacCerの脂質ラフトは,リガンドと結合するとC24-LacCerと脂肪酸鎖を介して直接会合しているLynやGαiが凝集したり,構造変化したりすることで細胞内へと情報を伝達する情報伝達ユニットとなっていると考えられる(図3).
グリセロ糖脂質であるホスファチジルグルコシド(PtdGlc)はヒト好中球の細胞膜上でLacCerとは異なる脂質ラフトを形成し,抗PtdGlc抗体はヒト好中球にアポトーシスを誘導する47, 48).PtdGlcはC18とC20の飽和の脂肪酸鎖を持つことから49),C24-LacCerのように細胞内の情報伝達分子と直接会合することはできない.抗PtdGlc抗体により誘導される好中球のアポトーシスは,Fasを介したアポトーシス誘導を阻害する抗Fas抗体で阻害されることから,膜貫通タンパク質であるFasを介して誘導されると考えられる.GPIアンカー型タンパク質もパルミチン酸やステアリン酸などの飽和脂肪鎖で構成されていることから50),GPIアンカー型タンパク質を介した細胞の活性化にはシグナルを伝えるFas分子のような膜貫通型のタンパク質分子や51),LacCerの脂質ラフトのような情報伝達ユニットが必要である.一方,脳から得られる酸性のGSLであるガングリオシドの脂肪酸鎖の多くは炭素数が20以下であるが,C24の脂肪酸鎖も存在する52).PC12細胞のGM1はC24の脂肪酸鎖を持っており,コレラ毒素BサブユニットがGM1に結合すると,GM1の脂質ラフトに会合したLynの活性化と神経突起伸長が起こる13).このように,糖脂質の脂質ラフトの機能において脂肪酸鎖の構造は大変重要であり,糖脂質の細胞機能を解明する上で細胞ごとに糖脂質の脂質構造を明らかにすることが重要である.
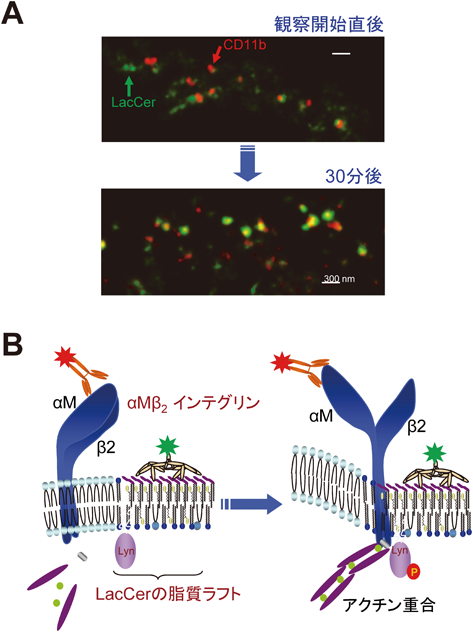
細胞接着分子であるαMβ2インテグリンは接着,遊走,貪食,活性酸素産生や脱顆粒など,好中球やマクロファージのさまざまな機能を仲介する.特に,αMβ2インテグリンはC3bi受容体としてオプソニンを介した貪食を仲介するだけではなく,αMサブユニットが直接リポ多糖(LPS)やβ-グルカンなどのPAMPs(pathogen-associated molecular patterns,病原体関連分子パターン)とレクチン様ドメインを介して結合することで,オプソニン非依存性の貪食を仲介するなど,貪食反応において中心的な役割を果たしている53).しかしながら,αMβ2インテグリンは細胞内にアクチンと結合するためのドメインを持つが,細胞内の情報伝達分子を活性化するような細胞内モチーフは持たない54).通常,αMβ2インテグリンがPAMPsに直接結合するか,ケモカイン・サイトカインあるいは菌体成分が細胞に結合することで引き起こされるインサイド—アウトのシグナルにより,αMβ2インテグリンの立体構造が変化してアクチン重合が起こる55).そして,菌と接合した部位に形成される細胞膜上のファゴサイティックカップ(phagocytic cup)にαMβ2インテグリンが集積するとともに,細胞内へと貪食シグナルを伝達する.このαMβ2インテグリンを介した細胞の活性化にはLynのリン酸化が必須であり,Lynの遺伝子発現を抑制するとαMβ2インテグリンを介した細胞機能は失われる38).D-HL-60はαMβ2インテグリンを発現しているが,オプソニン非依存的な貪食能がない.しかしながら,C24-LacCerを細胞膜に取り込ませてLynと会合したLacCerの脂質ラフトを再構成することで貪食能を獲得させることができる.無刺激状態では抗LacCer抗体で回収したLacCerの脂質ラフト画分にはαMβ2インテグリンは含まれない.一方,αMサブユニットに結合するβ-グルカンを含むザイモサンを好中球やD-HL-60に貪食させ,その食胞から抗LacCer抗体でLacCerの脂質ラフトを回収すると,β2サブユニットのみが回収される.さらに抗αMサブユニット活性化抗体VIM12でLacCerの脂質ラフトに会合しているLynがリン酸化され,このリン酸化はβ2サブユニットの膜境界領域に結合する抗β2サブユニット抗体で抑制される.また,好中球をVIM12で刺激すると,経時的にαMサブユニットがLacCerの脂質ラフトに会合するようすを超解像顕微鏡で観察することができる(図4A).このように,Lynと会合したLacCerの脂質ラフトはαMβ2インテグリンの活性化情報を細胞内へと伝える情報伝達ユニットとして機能している(図4B).GSLがC24の脂肪酸鎖を含む場合は,GSLの脂質ラフトは,常にパルミトイル化された細胞内情報伝達分子と会合した情報伝達ユニットを細胞膜上に形成する.そして,GSLの糖鎖部分を特異的に認識するリガンドがGSLに結合すると,GSLの脂質ラフトに親和性のあるインテグリンなどの分子の情報伝達ユニットとして機能することで細胞機能を発現すると考えられる.
4. 自然免疫におけるGSLの役割とGSLの糖鎖を介した病原体認識機構
貪食細胞である好中球やマクロファージは,生体内に侵入した病原体を除去する自然免疫担当細胞である.そのために,好中球やマクロファージはパターン認識受容体(pattern-recognition receptor:PRR)を発現しており,それらPRRがPAMPsと結合することで,自然免疫応答が開始される.PAMPsは,病原体特有,かつ,病原体の生存に必須の分子群であり,それらは哺乳動物には発現していない56).哺乳動物などが持つPRRは,細菌や真菌,ウイルスなどの病原体に発現する多様なPAMPを認識することができ,PRRの構造,機能,局在によって,さまざまな種類に分類されている57).PRRとしてこれまでに,Toll様受容体58, 59),マンノース受容体60),CD1461),スカベンジャー受容体62, 63),C型レクチン受容体であるDectin-164),αMβ2インテグリン65)等が同定されている.さらにPAMPsの一部はLacCer18, 24, 31, 38)やGM166, 67)のような宿主細胞に発現するGSLの糖鎖構造と結合できることが示されている68).たとえば,ポリオーマウイルス(polyomavirus)はヒト赤血球に発現しているガングリオシドのGD1aやGT1bと結合することができる67).SV40(simian virus 40)とBrucella suisはGM1と選択的に結合する66, 67).また,病原体自体がGSLに結合するだけでなく,それら由来の毒素もGSLに結合する.GM1はコレラ毒素のBサブユニットと特異的に結合する69).ヒト上皮細胞や血管内皮細胞に発現するグロボトリアオシルセラミド(Gb3)は赤痢菌の志賀毒素の受容体として機能している70–72).このようにGSLは病原微生物の感染ターゲットやPRRとして働いており,感染免疫において重要な鍵を握る分子であるといえる.
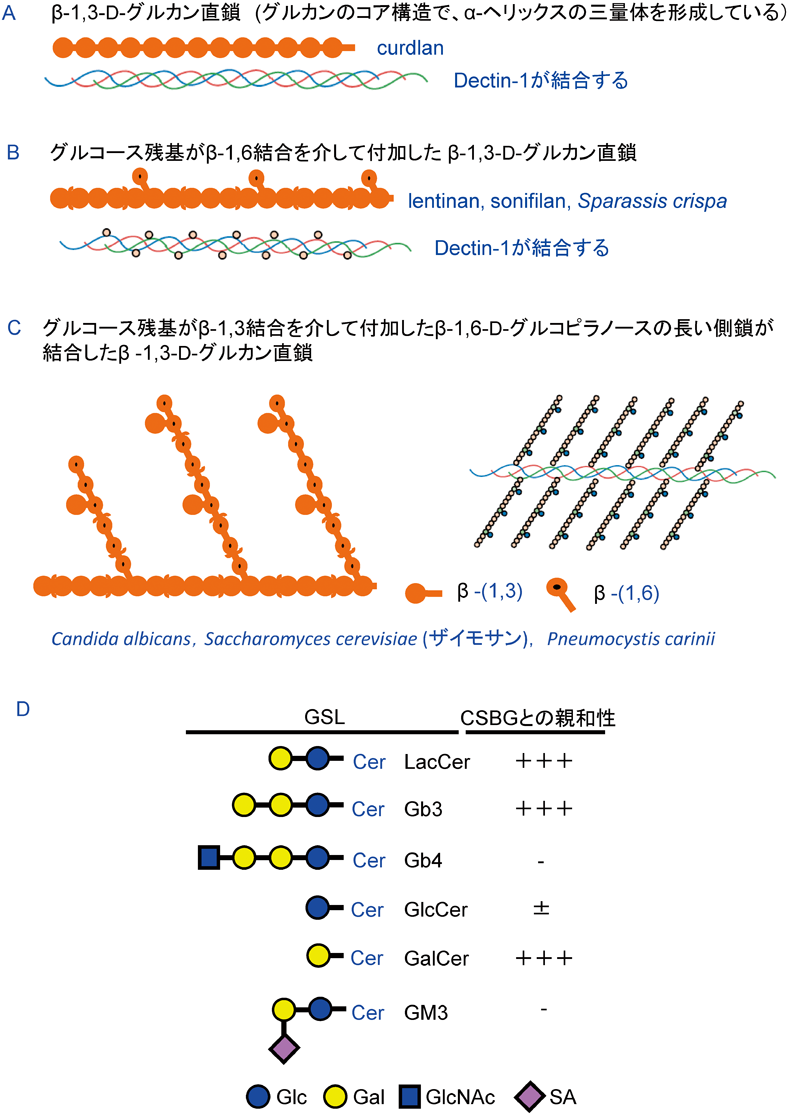
GSLの中でも,LacCerはさまざまな病原微生物と特異的に結合する14–20).それでは,どうしてLacCerはこれほどまでにさまざまな病原微生物と選択的に結合できるのであろうか.その分子機構を解き明かすために,我々は,病原性真菌であるカンジダ(Candida albicans)由来の可溶性β-グルカン(CSBG)がLynと会合したLacCerの脂質ラフトを介して好中球の遊走と活性酸素の産生を誘導することに着目した18, 31).β-グルカンとは,β-D-グルコピラノースがβ-1,3結合により直鎖を形成し,さらに,この直鎖にさまざまな長さの側鎖がβ-1,6結合により付加した不均一なポリマーであり,真菌細胞壁の主要な構成成分である(図5A~C)73, 74).キノコ由来のβ-グルカンは,β-1,3結合で形成されるβ-D-グルコピラノース直鎖が三量体のヘリックス構造をとり,これにグルコース残基がβ-1,6結合を介し付加しており(図5B)75),この三量体構造に,PRRの一つであるデクチン(Dectin-1)が結合する76).CSBGは,β-1,3結合で形成されるβ-D-グルコピラノース直鎖にβ-1,6結合したβ-D-グルコピラノースの長い側鎖が結合し,さらにこの側鎖のところどころにβ-1,3結合でグルコース残基が結合している74).CSBGは,GM1やGM2, GM3のようなガングリオシド,またはGlcCerやGb4には結合せず,ガラクトース末端を有するLacCerやGb3,ガラクトシルセラミド(GalCer)のようなGSLに結合する18)(図5D).反対に,LacCerはカードランのような,β-1,6結合したD-グルコピラノースにより形成される長い側鎖を持たないβ-グルカンには結合しない.したがって,CSBGによるLacCerを介した好中球の遊走に,LacCerのガラクトース構造とβ-1,6結合したD-グルコピラノースの長鎖にβ-1,3結合でβ-D-グルコピラノースが単分子結合している糖鎖の間で生じる糖–糖相互作用が必須であると考えられる.実際,CSBGと同様の構造である酵母由来のpoly-(1,6)-β-D-glucopyranosyl-(1,3)-β-D-glucopyranose glucan(PGG glucan)もガラクトース末端を有するGSLに結合する20).さらに,真菌であるPneumocystis carinii由来のβ-グルカンはCSBGと同じ構造であり,ラットの肺上皮細胞上のLacCerを介して,ケモカインであるMIP-2の産生を誘導する16).これらの結果は,LacCerの脂質ラフトが生物機能を仲介するPRRや病原微生物の感染ターゲットとして機能していることを示唆している.
5. リポアラビノマンナンとLacCerとの糖–糖相互作用による好中球の抗酸菌貪食機構
貪食細胞は病原体を貪食するとファゴソームを形成し,さらにファゴソームにリソソームが融合することで,病原体が分解・除去される.特にマクロファージや樹状細胞のような抗原提示細胞においては,病原体の分解後,主要組織適合抗原(MHC)クラスII分子を介して病原体由来ペプチドや脂質がT細胞に提示され,抗原特異的な獲得免疫系が発動する.リステリア,サルモネラ,病原性抗酸菌等の細胞内寄生細菌は,貪食細胞に取り込まれた後も,宿主の免疫システムを操ることで,細胞内で生存することができる.このような細菌に関しては,非オプソニン条件下において,宿主脂質ラフトを介して細胞内に取り込まれた後,効率よく殺菌を回避するいくつもの例が示されている77).これら細菌がファゴソームの中に取り込まれると,MHCクラスII分子を介した抗原提示機構の活性化を避けるために,菌はファゴソームから細胞質へ逃れるか,病原性抗酸菌のようにリソソームのファゴソームへの融合(ファゴソームの成熟)を妨害する78–86).好中球は結核菌の初期感染に対してきわめて防御的な役割を担っている87–89).しかしながら,好中球による結核菌の貪食機構は,結核菌の病原性を追究する上で重要なトピックであるにも関わらず,マクロファージなどと比較してこれまであまり注意を向けられてこなかった.そこで最後に我々のLacCerを介した抗酸菌の貪食と病原性抗酸菌による最新の知見を紹介する90).
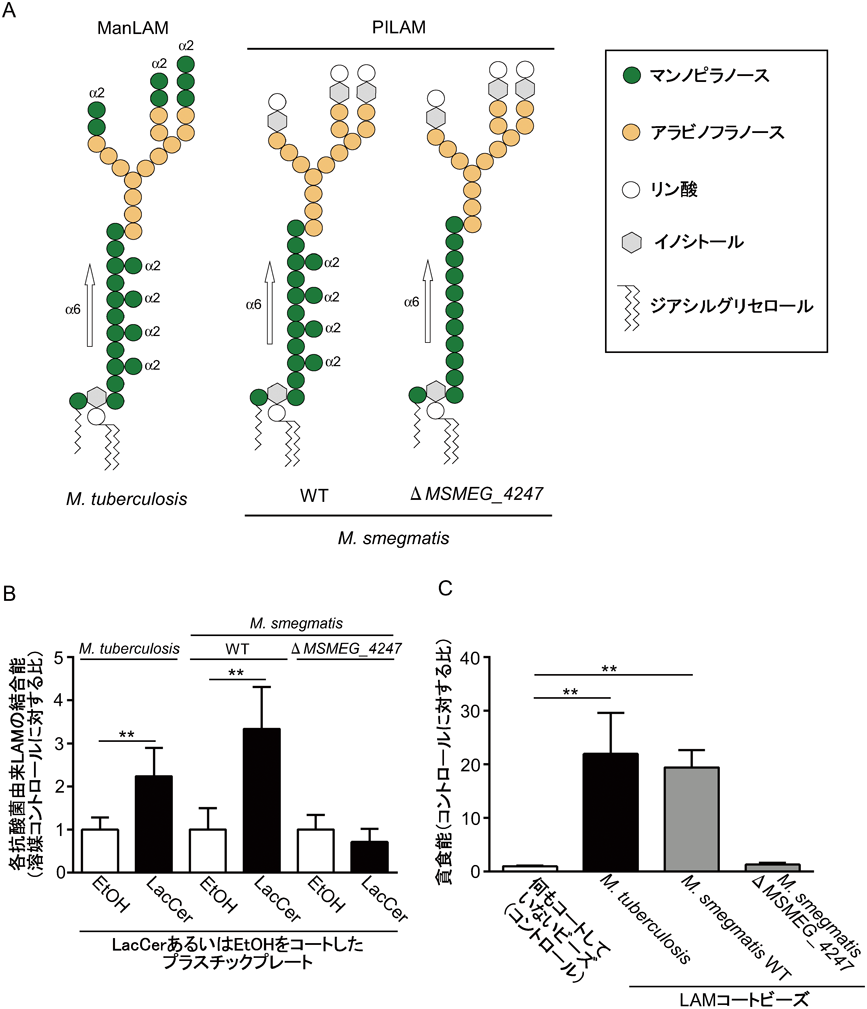
抗酸菌はミコール酸,リポマンナン(LM)やリポアラビノマンナン(LAM)など,抗酸菌に特有の脂質を菌体表面に発現している.結核菌(Mycobacterium tuberculosis)やM. avium-intracellulare Complex(MAC)のような病原性抗酸菌は,マンノースキャップ型のLAM(ManLAM)を発現している.その一方で,M. smegmatisのような非病原性抗酸菌は,ホスファチジルイノシトールキャップ型のLAM(PILAM)や,キャップ構造のないLAMを発現している91).ヒトマクロファージのマンノース受容体とManLAMとの結合が,結核菌によるファゴソーム成熟阻害の一因であることが報告されている92).また,ManLAMコートビーズがマウスマクロファージ様細胞株であるJ774細胞内において,PI3-キナーゼ(phosphatidylinositol 3-kinase:PI3-K)依存的なファゴソームの成熟を抑制する93).さらにマウス骨髄由来マクロファージにおいて,菌体表面のManLAM量を低下させた結核菌H37Rv株の突然変異株(ΔlprG)では,ファゴソームへのリソソームの融合阻害が低減される94, 95).これらの知見は,LAM構造の違いが抗酸菌による病原性を規定している可能性を示唆している.しかしながら,ゼブラフィッシュとマウスに,M. marinumとM. bovis BCGのマンノースキャップ構造を欠失させた変異株をそれぞれ感染させても,肺などの組織における菌の生存率が低下しないことが報告されている96).このように,抗酸菌の病原性におけるManLAMの機能については相反する報告がなされている.特にヒト貪食細胞内における病原性抗酸菌の食胞成熟回避におけるManLAMと宿主側の結合分子との相互作用の分子メカニズムはほとんど解明されてこなかった.
ヒト好中球は,抗酸菌を貪食する際に,病原性に関わらずLacCerを抗酸菌の周囲に集積させ,LacCerの脂質ラフトとαMβ2インテグリンに依存して抗酸菌を貪食する90).LAMは抗酸菌種間に共通して発現しており,その構造は,マンナンコアにアラビノフラノースが付加することで形成されている(図6A)97).マンナンコアは,マンノースがPIに結合したホスファチジルイノシトールマンノシド(PIM)に対して21~34個のα-1,6結合マンナンおよび側鎖として1分子のマンノピラノースがα-1,2結合を介して付加された構造である.抗酸菌から精製したLAMは,病原性に関わらずLacCerのリポソームに選択的に結合する.非病原性抗酸菌であるM. smegmatisは,MSMEG_4247と呼ばれる遺伝子を持っており,この遺伝子がコードするα-1,2-マンノシルトランスフェラーゼは,α-1,6-マンナン直鎖のところどころにα-1,2結合でマンノピラノースを付加する98).この酵素の欠損株であるΔMSMEG_4247のLAMはα-1,2-マンノース側鎖のないマンナンコアでできているが,ΔMSMEG_4247は好中球に貪食されない90).結核菌由来およびM. smegmatisの野生型(WT)株由来のLAMはLacCerと強く結合するが,ΔMSMEG_4247由来LAMは結合しない(図6B).また,好中球は結核菌やM. smegmatis WT由来のLAMをコートしたビーズを貪食するが,ΔMSMEG_4247由来のLAMをコートしたビーズは貪食しない(図6C).これらの結果は,ヒト好中球のLacCerがすべての抗酸菌種の持つLAMのα-1,2-モノマンノース側鎖を含むマンナンコア部分と結合することで抗酸菌を貪食することを示している.
ヒト好中球は,病原性の違いによらず抗酸菌を同じように貪食するが,細胞内へ取り込まれた後の抗酸菌の運命は異なる.非病原性抗酸菌であるM. gordonaeは,好中球に貪食されると,アズール顆粒と呼ばれるリソソーム99)がファゴソームと融合し,消化・殺菌される(図7A).しかしながら,結核菌やMACのような病原性抗酸菌はリソソームの融合を阻止し,細胞内に生き残ることができる(図7A).SrcファミリーキナーゼであるHckは,ヒト好中球の顆粒画分に存在し100),ファゴソームへのリソソーム融合に必須の分子である101).ヒト好中球のLacCerの90%は顆粒膜において脂質ラフトを形成し22, 24),ファゴソーム膜でも脂質ラフトを形成する38).ファゴソームを形成する過程で,さまざまな顆粒膜がファゴソームに融合する.ヒト好中球にPILAMあるいはManLAMをコートしたビーズを貪食させた後,LAMコートビーズを含むファゴソームを分離し,それらを可溶化後,抗LacCer抗体で免疫沈降を行うと,PILAMコートビーズを含むファゴソームからリン酸化されたHckが免疫沈降される(図7B).その一方で,HckはManLAMコートビーズを含むファゴソームからは免疫沈降されない.さらに,抗酸菌を貪食した好中球のファゴソームを観察すると,非病原性抗酸菌であるM. gordonaeを含むファゴソームでLacCerは大きなクラスターを形成し,Hckと共局在するのに対して,結核菌を含むファゴソームでは,LacCerの大きなクラスター形成が阻止され,Hckとも共局在しない(図7C).このように,好中球はLynと会合したLacCerの脂質ラフトとLAMのマンナンコアとの結合を使って抗酸菌を貪食する一方で,病原性抗酸菌はファゴソーム膜形成過程で新たに作られるHcK会合型のLacCerの脂質ラフト形成を阻害し,ファゴソームにリソソームが融合するための情報を遮断することでファゴソーム成熟を回避している.
LacCerの関与する生物機能は自然免疫にとどまらない.最近,慢性中枢神経系炎症として知られる多発性硬化症のモデルマウスにおいて,アストロサイトでのLacCer合成酵素B4GALT6によるLacCer産生能の亢進が,炎症の惹起や神経変性に関与することが示されている102).さらに,脳脊髄根末梢神経炎のような抗LacCer抗体による自己免疫疾患が観察されている40).しかしながら,これらの論文では実際のLacCerの存在形態については解析されておらず,これらの現象の背景にあるメカニズムが一体どのようなものであるかは不明であり,今後の研究の展開が待たれる.糖鎖の違いや脂質構造の違いがあると,GSLは異なる脂質ラフトとして細胞膜上に存在する32, 47).また,人工膜上で作った同じGSLが形成する同一のドメインにおいても,ドメインの部位によって抗原特異性が異なることは39),さまざまな分子が会合する細胞膜の脂質ラフトでは,より複雑な抗原特異性を示すと考えられる.したがって,GSLが関与する生命現象や疾患を解明するためには,脂質ラフトに会合する分子群が示す立体構造や相互作用を生理的な状態で解析する必要があり,それら分子間の相互作用や役割を解析するさらなる工夫が求められている.
謝辞Acknowledgments
この総説で紹介しました我々の研究は,ワシントン大学の箱守仙一郎先生のご教示から始まった研究であり,多くの共同研究者,研究支援者の皆様のご尽力,ご協力いただいた成果です.心から感謝申し上げます.また,一連の研究は文部科学省や諸財団のご援助の下,順天堂大学環境医学研究所において行うことができましたことに深謝申し上げます.
引用文献References
1) Degroote, S., Wolthoorn, J., & van Meer, G. (2004) Semin. Cell Dev. Biol., 15, 375–387.
2) Hakomori, S. (2003) Curr. Opin. Hematol., 10, 16–24.
3) Kaga, N., Kazuno, S., Taka, H., Iwabuchi, K., & Murayama, K. (2005) Anal. Biochem., 337, 316–324.
4) Sonnino, S., Prinetti, A., Mauri, L., Chigorno, V., & Tettamanti, G. (2006) Chem. Rev., 106, 2111–2125.
5) Brown, D.A. & London, E. (1997) Biophys. Res. Commun., 240, 1–7.
6) Iwabuchi, K., Handa, K., & Hakomori, S. (1998) J. Biol. Chem., 273, 33766–33773.
7) Iwabuchi, K., Yamamura, S., Prinetti, A., Handa, K., & Hakomori, S. (1998) J. Biol. Chem., 273, 9130–9138.
8) Yamamura, S., Handa, K., & Hakomori, S. (1997) Biochem. Biophys. Res. Commun., 236, 218–222.
9) Kabayama, K., Sato, T., Saito, K., Loberto, N., Prinetti, A., Sonnino, S., Kinjo, M., Igarashi, Y., & Inokuchi, J. (2007) Proc. Natl. Acad. Sci. USA, 104, 13678–13683.
10) Ringerike, T., Blystad, F.D., Levy, F.O., Madshus, I.H., & Stang, E. (2002) J. Cell Sci., 115, 1331–1340.
11) Roepstorff, K., Thomsen, P., Sandvig, K., & van Deurs, B. (2002) J. Biol. Chem., 277, 18954–18960.
12) Mutoh, T., Tokuda, A., Miyadai, T., Hamaguchi, M., & Fujiki, N. (1995) Proc. Natl. Acad. Sci. USA, 92, 5087–5091.
13) Ichikawa, N., Iwabuchi, K., Kurihara, H., Ishii, K., Kobayashi, T., Sasaki, T., Hattori, N., Mizuno, Y., Hozumi, K., Yamada, Y., & Arikawa-Hirasawa, E. (2009) J. Cell Sci., 122, 289–299.
14) Abul-Milh, M., Paradis, S.E., Dubreuil, J.D., & Jacques, M. (1999) Infect. Immun., 67, 4983–4987.
15) Angstrom, J., Teneberg, S., Milh, M.A., Larsson, T., Leonardsson, I., Olsson, B.M., Halvarsson, M.O., Danielsson, D., Naslund, I., Ljungh, A., Wadstrom, T., & Karlsson, K.A. (1998) Glycobiology, 8, 297–309.
16) Hahn, P.Y., Evans, S.E., Kottom, T.J., Standing, J.E., Pagano, R.E., & Limper, A.H. (2003) J. Biol. Chem., 278, 2043–2050.
17) Karlsson, K.A. (1986) Chem. Phys. Lipids, 42, 153–172.
18) Sato, T., Iwabuchi, K., Nagaoka, I., Adachi, Y., Ohno, N., Tamura, H., Seyama, K., Fukuchi, Y., Nakayama, H., Yoshizaki, F., Takamori, K., & Ogawa, H. (2006) J. Leukoc. Biol., 80, 204–211.
19) Saukkonen, K., Burnette, W.N., Mar, V.L., Masure, H.R., & Tuomanen, E.I. (1992) Proc. Natl. Acad. Sci. USA, 89, 118–122.
20) Zimmerman, J.W., Lindermuth, J., Fish, P.A., Palace, G.P., Stevenson, T.T., & DeMong, D.E. (1998) J. Biol. Chem., 273, 22014–22020.
21) Brackman, D., Lund-Johansen, F., & Aarskog, D. (1995) J. Leukoc. Biol., 58, 547–555.
22) Kniep, B. & Skubitz, K.M. (1998) J. Leukoc. Biol., 63, 83–88.
23) Spychalska, J., Smolenska-Sym, G., Zdebska, E., Wozniak, J., & Koscielak, J. (2003) Cell. Mol. Biol. Lett., 8, 911–917.
24) Iwabuchi, K. & Nagaoka, I. (2002) Blood, 100, 1454–1464.
25) Murate, M., Abe, M., Kasahara, K., Iwabuchi, K., Umeda, M., & Kobayashi, T. (2015) J. Cell Sci., 128, 1627–1638.
26) Iwabuchi, K., Nakayama, H., Masuda, H., Kina, K., Ogawa, H., & Takamori, K. (2012) Biofactors, 38, 275–283.
27) Hakomori, S., Handa, K., Iwabuchi, K., Yamamura, S., & Prinetti, A. (1998) Glycobiology, 8, xi–xix.
28) Simons, K. & Ikonen, E. (1997) Nature, 387, 569–572.
29) Pike, L.J. (2003) J. Lipid Res., 1, 1.
30) Singh, R.D., Puri, V., Valiyaveettil, J.T., Marks, D.L., Bittman, R., & Pagano, R.E. (2003) Mol. Biol. Cell, 14, 3254–3265.
31) Iwabuchi, K., Prinetti, A., Sonnino, S., Mauri, L., Kobayashi, T., Ishii, K., Kaga, N., Murayama, K., Kurihara, H., Nakayama, H., Yoshizaki, F., Takamori, K., Ogawa, H., & Nagaoka, I. (2008) Glycoconj. J., 25, 357–374.
32) Fujita, A., Cheng, J., & Fujimoto, T. (2009) Biochim. Biophys. Acta, 1791, 388–396.
33) Suzuki, K.G., Fujiwara, T.K., Sanematsu, F., Iino, R., Edidin, M., & Kusumi, A. (2007) J. Cell Biol., 177, 717–730.
34) Pralle, A., Keller, P., Florin, E.L., Simons, K., & Horber, J.K. (2000) J. Cell Biol., 148, 997–1008.
35) Saxena, K., Zimmermann, P., Schmidt, R.R., & Shipley, G.G. (2000) Biophys. J., 78, 306–312.
36) Ferraretto, A., Pitto, M., Palestini, P., & Masserini, M. (1997) Biochemistry, 36, 9232–9236.
37) Ariga, T. & Yu, R.K. (2005) J. Neurosci. Res., 80, 1–17.
38) Nakayama, H., Yoshizaki, F., Prinetti, A., Sonnino, S., Mauri, L., Takamori, K., Ogawa, H., & Iwabuchi, K. (2008) J. Leukoc. Biol., 83, 728–741.
39) Iwabuchi, K., Masuda, H., Kaga, N., Nakayama, H., Matsumoto, R., Iwahara, C., Yoshizaki, F., Tamaki, Y., Kobayashi, T., Hayakawa, T., Ishii, K., Yanagida, M., Ogawa, H., & Takamori, K. (2015) Glycobiology, 25, 655–668.
40) Shima, S., Kawamura, N., Ishikawa, T., Masuda, H., Iwahara, C., Niimi, Y., Ueda, A., Iwabuchi, K., & Mutoh, T. (2014) Neurology, 82, 114–118.
41) Levy, M. & Futerman, A.H. (2010) IUBMB Life, 62, 347–356.
42) Grant, C.W., Mehlhorn, I.E., Florio, E., & Barber, K.R. (1987) Biochim. Biophys. Acta, 902, 169–177.
43) Morrow, M.R., Singh, D., Lu, D., & Grant, C.W. (1995) Biophys. J., 68, 179–186.
44) Li, X.M., Momsen, M.M., Brockman, H.L., & Brown, R.E. (2002) Biophys. J., 83, 1535–1546.
45) Chiricozzi, E., Ciampa, M.G., Brasile, G., Compostella, F., Prinetti, A., Nakayama, H., Ekyalongo, R.C., Iwabuchi, K., Sonnino, S., & Mauri, L. (2015) J. Lipid Res., 56, 129–141.
46) Ullrich, A. & Schlessinger, J. (1990) Cell, 61, 203–212.
47) Kina, K., Masuda, H., Nakayama, H., Nagatsuka, Y., Nabetani, T., Hirabayashi, Y., Takahashi, Y., Shimada, K., Daida, H., Ogawa, H., Takamori, K., & Iwabuchi, K. (2011) J. Immunol., 186, 5323–5332.
48) Nagatsuka, Y., Hara-Yokoyama, M., Kasama, T., Takekoshi, M., Maeda, F., Ihara, S., Fujiwara, S., Ohshima, E., Ishii, K., Kobayashi, T., Shimizu, K., & Hirabayashi, Y. (2003) Proc. Natl. Acad. Sci. USA, 100, 7454–7459.
49) Nagatsuka, Y., Horibata, Y., Yamazaki, Y., Kinoshita, M., Shinoda, Y., Hashikawa, T., Koshino, H., Nakamura, T., & Hirabayashi, Y. (2006) Biochemistry, 45, 8742–8750.
50) Maeda, Y., Tashima, Y., Houjou, T., Fujita, M., Yoko-o, T., Jigami, Y., Taguchi, R., & Kinoshita, T. (2007) Mol. Biol. Cell, 18, 1497–1506.
51) Kusumi, A., Shirai, Y.M., Koyama-Honda, I., Suzuki, K.G., & Fujiwara, T.K. (2010) FEBS Lett., 584, 1814–1823.
52) Sarbu, M., Robu, A.C., Ghiulai, R.M., Vukelic, Z., Clemmer, D.E., & Zamfir, A.D. (2016) Anal. Chem., 88, 5166–5178.
53) Yoshizaki, F., Nakayama, H., Iwahara, C., Takamori, K., Ogawa, H., & Iwabuchi, K. (2008) Biochim. Biophys. Acta, 1780, 383–392.
54) Rabb, H., Michishita, M., Sharma, C.P., Brown, D., & Arnaout, M.A. (1993) J. Immunol., 151, 990–1002.
55) Piccardoni, P., Manarini, S., Federico, L., Bagoly, Z., Pecce, R., Martelli, N., Piccoli, A., Totani, L., Cerletti, C., & Evangelista, V. (2004) Biochem. J., 380, 57–65.
56) Medzhitov, R. & Janeway, C. Jr. (2000) Immunol. Rev., 173, 89–97.
57) Gordon, S. (2002) Cell, 111, 927–930.
58) Blander, J.M. & Medzhitov, R. (2004) Science, 304, 1014–1018.
59) Doyle, S.E., O’Connell, R.M., Miranda, G.A., Vaidya, S.A., Chow, E.K., Liu, P.T., Suzuki, S., Suzuki, N., Modlin, R.L., Yeh, W.C., Lane, T.F., & Cheng, G. (2004) J. Exp. Med., 199, 81–90.
60) Ezekowitz, R.A., Sastry, K., Bailly, P., & Warner, A. (1990) J. Exp. Med., 172, 1785–1794.
61) Jiang, Z., Georgel, P., Du, X., Shamel, L., Sovath, S., Mudd, S., Huber, M., Kalis, C., Keck, S., Galanos, C., Freudenberg, M., & Beutler, B. (2005) Nat. Immunol., 6, 565–570.
62) Elomaa, O., Kangas, M., Sahlberg, C., Tuukkanen, J., Sormunen, R., Liakka, A., Thesleff, I., Kraal, G., & Tryggvason, K. (1995) Cell, 80, 603–609.
63) Peiser, L., Gough, P.J., Kodama, T., & Gordon, S. (2000) Infect. Immun., 68, 1953–1963.
64) Herre, J., Marshall, A.S., Caron, E., Edwards, A.D., Williams, D.L., Schweighoffer, E., Tybulewicz, V., Reis e Sousa, C., Gordon, S., & Brown, G.D. (2004) Blood, 104, 4038–4045.
65) Brown, E.J. (1991) Curr. Opin. Immunol., 3, 76–82.
66) Naroeni, A. & Porte, F. (2002) Infect. Immun., 70, 1640–1644.
67) Tsai, B., Gilbert, J.M., Stehle, T., Lencer, W., Benjamin, T.L., & Rapoport, T.A. (2003) EMBO J., 22, 4346–4355.
68) Schengrund, C.L. (2003) Biochem. Pharmacol., 65, 699–707.
69) Nagafuku, M., Okuyama, K., Onimaru, Y., Suzuki, A., Odagiri, Y., Yamashita, T., Iwasaki, K., Fujiwara, M., Takayanagi, M., Ohno, I., & Inokuchi, J. (2012) Proc. Natl. Acad. Sci. USA, 109, E336–E342.
70) Lingwood, C.A. (1996) Trends Microbiol., 4, 147–153.
71) Louise, C.B., Kaye, S.A., Boyd, B., Lingwood, C.A., & Obrig, T.G. (1995) Infect. Immun., 63, 2766–2769.
72) Takenouchi, H., Kiyokawa, N., Taguchi, T., Matsui, J., Katagiri, Y.U., Okita, H., Okuda, K., & Fujimoto, J. (2004) J. Cell Sci., 117, 3911–3922.
73) Liang, J., Melican, D., Cafro, L., Palace, G., Fisette, L., Armstrong, R., & Patchen, M.L. (1998) Int. J. Immunopharmacol., 20, 595–614.
74) Ohno, N., Uchiyama, M., Tsuzuki, A., Tokunaka, K., Miura, N.N., Adachi, Y., Aizawa, M.W., Tamura, H., Tanaka, S., & Yadomae, T. (1999) Carbohydr. Res., 316, 161–172.
75) Miura, N.N., Adachi, Y., Yadomae, T., Tamura, H., Tanaka, S., & Ohno, N. (2003) Microbiol. Immunol., 47, 173–182.
76) Hanashima, S., Ikeda, A., Tanaka, H., Adachi, Y., Ohno, N., Takahashi, T., & Yamaguchi, Y. (2014) Glycoconj. J., 31, 199–207.
77) Manes, S., del Real, G., & Martinez, A.C. (2003) Nat. Rev. Immunol., 3, 557–568.
78) Baorto, D.M., Gao, Z., Malaviya, R., Dustin, M.L., van der Merwe, A., Lublin, D.M., & Abraham, S.N. (1997) Nature, 389, 636–639.
79) Brumell, J.H., Kujat-Choy, S., Brown, N.F., Vallance, B.A., Knodler, L.A., & Finlay, B.B. (2003) Traffic, 4, 36–48.
80) Gatfield, J. & Pieters, J. (2000) Science, 288, 1647–1650.
81) Gekara, N.O. & Weiss, S. (2004) Biochem. Soc. Trans., 32, 712–714.
82) Harrison, R.E., Brumell, J.H., Khandani, A., Bucci, C., Scott, C.C., Jiang, X., Finlay, B.B., & Grinstein, S. (2004) Mol. Biol. Cell, 15, 3146–3154.
83) Knodler, L.A., Vallance, B.A., Hensel, M., Jackel, D., Finlay, B.B., & Steele-Mortimer, O. (2003) Mol. Microbiol., 49, 685–704.
84) Stuart, E.S., Webley, W.C., & Norkin, L.C. (2003) Exp. Cell Res., 287, 67–78.
85) Tamilselvam, B. & Daefler, S. (2008) J. Immunol., 180, 8262–8271.
86) Watarai, M., Makino, S., Fujii, Y., Okamoto, K., & Shirahata, T. (2002) Cell. Microbiol., 4, 341–355.
87) Eum, S.Y., Kong, J.H., Hong, M.S., Lee, Y.J., Kim, J.H., Hwang, S.H., Cho, S.N., Via, L.E., & Barry, C.E. 3rd. (2010) Chest, 137, 122–128.
88) Appelberg, R., Castro, A.G., Gomes, S., Pedrosa, J., & Silva, M.T. (1995) Infect. Immun., 63, 3381–3387.
89) Saunders, B.M. & Cheers, C. (1996) Infect. Immun., 64, 4236–4241.
90) Nakayama, H., Kurihara, H., Morita, Y.S., Kinoshita, T., Mauri, L., Prinetti, A., Sonnino, S., Yokoyama, N., Ogawa, H., Takamori, K., & Iwabuchi, K. (2016) Sci. Signal., 9, ra101.
91) Kaur, D., Obregon-Henao, A., Pham, H., Chatterjee, D., Brennan, P.J., & Jackson, M. (2008) Proc. Natl. Acad. Sci. USA, 105, 17973–17977.
92) Kang, P.B., Azad, A.K., Torrelles, J.B., Kaufman, T.M., Beharka, A., Tibesar, E., DesJardin, L.E., & Schlesinger, L.S. (2005) J. Exp. Med., 202, 987–999.
93) Fratti, R.A., Chua, J., Vergne, I., & Deretic, V. (2003) Proc. Natl. Acad. Sci. USA, 100, 5437–5442.
94) Shukla, S., Richardson, E.T., Athman, J.J., Shi, L., Wearsch, P.A., McDonald, D., Banaei, N., Boom, W.H., Jackson, M., & Harding, C.V. (2014) PLoS Pathog., 10, e1004471.
95) Gaur, R.L., Ren, K., Blumenthal, A., Bhamidi, S., Gonzalez-Nilo, F.D., Jackson, M., Zare, R.N., Ehrt, S., Ernst, J.D., & Banaei, N. (2014) PLoS Pathog., 10, e1004376.
96) Appelmelk, B.J., den Dunnen, J., Driessen, N.N., Ummels, R., Pak, M., Nigou, J., Larrouy-Maumus, G., Gurcha, S.S., Movahedzadeh, F., Geurtsen, J., Brown, E.J., Eysink Smeets, M.M., Besra, G.S., Willemsen, P.T., Lowary, T.L., van Kooyk, Y., Maaskant, J.J., Stoker, N.G., van der Ley, P., Puzo, G., Vandenbroucke-Grauls, C.M., Wieland, C.W., van der Poll, T., Geijtenbeek, T.B., van der Sar, A.M., & Bitter, W. (2008) Cell. Microbiol., 10, 930–944.
97) Mishra, A.K., Driessen, N.N., Appelmelk, B.J., & Besra, G.S. (2011) FEMS Microbiol. Rev., 35, 1126–1157.
98) Sena, C.B., Fukuda, T., Miyanagi, K., Matsumoto, S., Kobayashi, K., Murakami, Y., Maeda, Y., Kinoshita, T., & Morita, Y.S. (2010) J. Biol. Chem., 285, 13326–13336.
99) Dell’Angelica, E.C., Mullins, C., Caplan, S., & Bonifacino, J.S. (2000) FASEB J., 14, 1265–1278.
100) Mohn, H., Le Cabec, V., Fischer, S., & Maridonneau-Parini, I. (1995) Biochem. J., 309, 657–665.
101) Peyron, P., Maridonneau-Parini, I., & Stegmann, T. (2001) J. Biol. Chem., 276, 35512–35517.
102) Rostami, A. & Ciric, B. (2014) Nat. Med., 20, 1092–1093.
著者紹介Author Profile
 中山 仁志(なかやま ひとし)
中山 仁志(なかやま ひとし)順天堂大学医療看護学部准教授.医学博士.
略歴千葉県に生まれる.2008年順天堂大学大学院医学研究科修了(医学博士).同年同大学院医学研究科環境医学研究所博士研究員.09年順天堂大学医療看護学部助教(医学研究科環境医学研究所併任).16年順天堂大学医療看護学部准教授.
研究テーマと抱負抗酸菌感染におけるスフィンゴ糖脂質の役割を多面的なアプローチから明らかにしていきたい.
趣味研究についてあれこれ考えること,美味しいお酒を飲むこと,子供と遊ぶこと.
 岩渕 和久(いわぶち かずひさ)
岩渕 和久(いわぶち かずひさ)順天堂大学医療看護学部教授.医学博士.
略歴大阪府に生まれる.東京理科大学薬学部卒業,1986年順天堂大学医学部助手,97年ワシントン大学上級研究員,ノースウェストリサーチ研究所客員研究員,2002年順天堂大学医学部講師,04年順天堂大学助教授(医療看護学部・医学研究科),06年順天堂大学医療看護学部教授,07年順天堂大学医療看護学研究科感染制御看護学分野教授.
研究テーマと抱負生命現象における糖脂質の脂質ラフトの機能とその分子メカニズムを解明したい.
ウェブサイトhttp://www.juntendo.ac.jp/graduate/laboratory/labo/kankyo_igaku/k4_iwabuchi.html
趣味良い音に浸ること,自然に浸ること