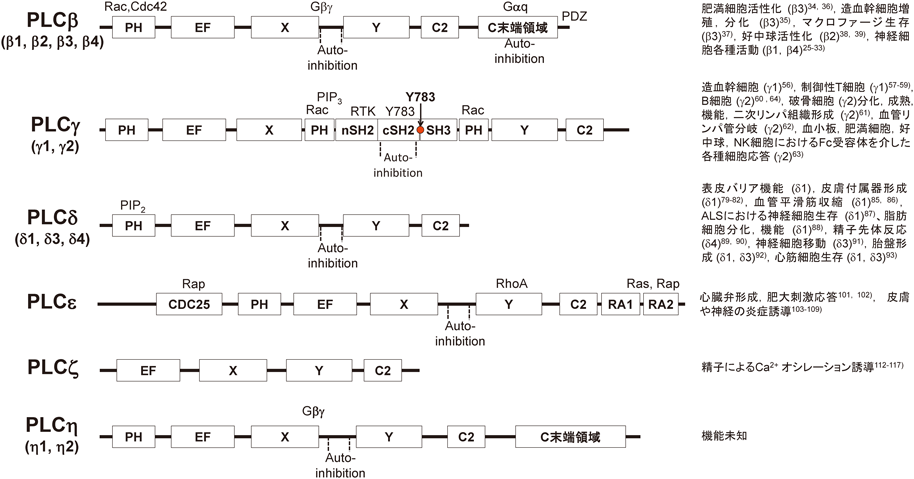
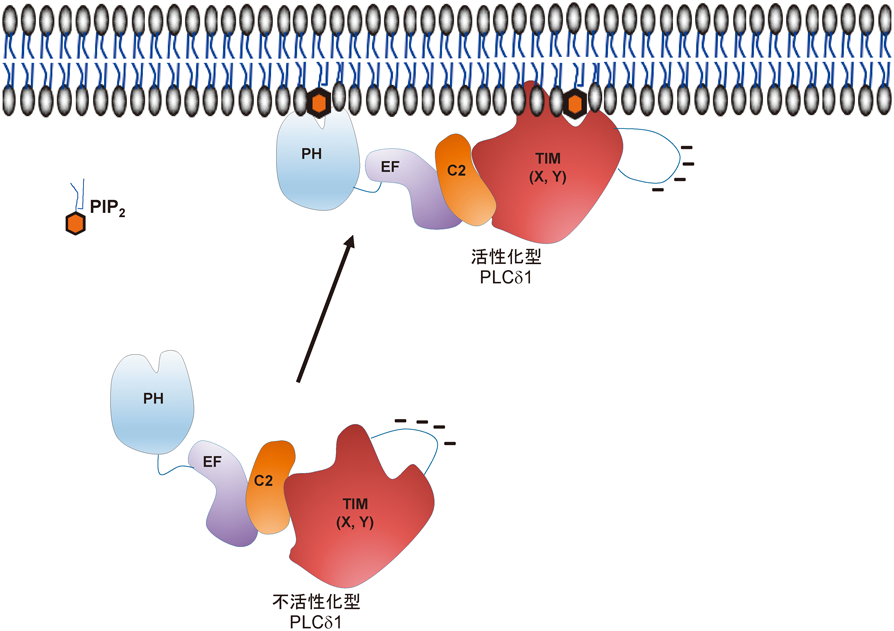
イノシトールリン脂質代謝系は細胞骨格や細胞内輸送など多様な細胞機能を調節する細胞内情報伝達系である.ホスホリパーゼC(phospholipase C:PLC)はイノシトールリン脂質代謝系においてセカンドメッセンジャー産生のトリガーとなる酵素であり,細胞膜に存在するホスファチジルイノシトール4,5-二リン酸(phosphatidylinositol 4,5-bisphosphate:PIP2)を加水分解し,二つのセカンドメッセンジャーであるイノシトール1,4,5-三リン酸(inositol 1,4,5-trisphosphate:IP3)とジアシルグリセロール(diacylglycerol:DAG)を産生する.前者は小胞体からのCa2+の遊離を促し,後者は,プロテインキナーゼCを活性化することで,さまざまな細胞応答を促す1, 2).また,PLCの基質であるPIP2自体もアクチン細胞骨格やエンドサイトーシス,イオンチャネルの活性を制御する.さらにPIP2はホスファチジルイノシトール3,4,5-三リン酸(PIP3)の前駆物質であり,PIP2の増減は細胞増殖や細胞生存などPIP3が担うさまざまな細胞機能調節にも関与する.哺乳動物では13種類のPLCが存在しており,それらは,構造および活性化機構によって六つのサブタイプ(β, γ, δ, ε, ζ, η)に大別される(図1)3).これらのPLCは共通してpleckstrin homology(PH)ドメイン(ζを除く),EFハンドモチーフ,TIMバレルドメイン(XドメインとYドメイン),C2ドメインといった構造を有している.PLCは各種刺激に応じて活性化される必要があるため,刺激のない状態では不活性化型に保たれている.PLCのXドメインとYドメインの間に存在する配列(X–Yリンカー)はPLCの活性中心をマスクすることでPLCを不活性化状態に保っている.X–Yリンカーは負に荷電しており,PLCが各種刺激により細胞膜に移行するとX–Yリンカーが細胞膜の負電荷と反発し活性中心から離れ,活性中心が基質であるPIP2に接触することが可能になる(図2)4).PLC各サブタイプはそれぞれ異なる細胞内情報伝達系と共役しており,PLCβはGタンパク質共役型受容体(G protein-coupled receptor:GPCR),PLCγは受容体型チロシンキナーゼ,PLCδは細胞内Ca2+濃度上昇,PLCεはRasやRapなどの低分子量Gタンパク質により活性化される.このようなサブタイプ特異的な活性制御機構には各サブタイプの持つ特徴的な構造が深く関与している5, 6).本稿ではPLCの活性制御機構と個体レベルでの生理機能について筆者らの研究成果も含め概説する.
1)組織分布,構造,活性化機構
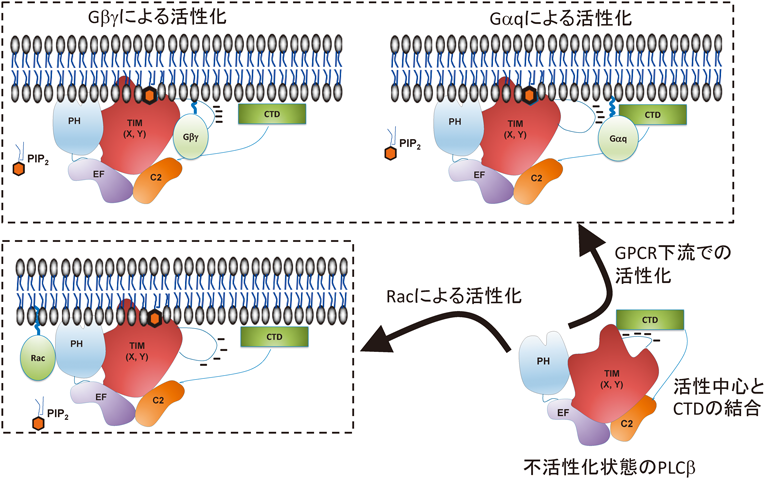
PLCβにはβ1, β2, β3, β4の四つのアイソザイムが存在している.PLCβ3は多くの組織に発現するPLCアイソザイムであるが,それ以外のPLCβアイソザイムは特徴的な組織分布を示しており,PLCβ1とPLCβ4は神経系組織において発現が高く,PLCβ2は血球系細胞に高い発現がみられる.PLCβは他のPLCサブタイプと共通のドメイン構造に加えて,約400アミノ酸からなるC末端領域とPDZ結合モチーフという特徴的な構造を持つ.PLCβのPHドメインはRhoファミリー低分子量Gタンパク質であるRacやCdc42と結合し,これらの分子によるPLCβの活性化に関与する7–12).C末端領域はPLCβの細胞膜への結合に重要であることに加え,Gαqと結合することによりGPCRの下流でPLCβの活性化を誘導する13–18).また,GβγがPLCβのPHドメインまたはTIMバレルへ結合することもGPCRの下流におけるPLCβの活性化に関与する19).PLCβはGαqに対してGTPase活性化タンパク質(GTPase-activating protein:GAP)として働き20–22),グアノシン三リン酸(guanosine triphosphate:GTP)の分解を促進していることも報告されており,Gαqの過剰な活性化を抑制する働きも持つと考えられる.さらに最近になりPLCβ1のC末端領域は細胞膜の曲率を高める働きを持つこと,PLCβ1は高曲率膜に存在するPIP2を効率よく切断することが報告された23).これはPLCβ1のC末端領域が細胞膜曲率を変化させるというユニークなメカニズムによってPLCβの活性化を促進することを示唆するものであり興味深い.ここまで述べてきたようにPLCβのC末端領域は刺激に応じたPLCβの活性化に重要であるが,休止状態の細胞においてはC末端領域が活性中心へ分子内結合することでPLCβを不活性化状態に保つ役割も有している16).PLCβの持つPDZ結合モチーフはPDZドメインを持つ足場タンパク質との結合を介してGPCRとの選択的な共役に寄与しているものと考えられる24).以上のようにPLCβはGαq, Gβγ, Rhoファミリー低分子量Gタンパク質などにより活性制御を受ける(図3).
2)生理機能
a.PLCβ1, PLCβ4による神経系組織の機能制御
PLCβ1やβ4は前述のように脳,神経系に多く発現しており,これらの組織の正常な機能維持に重要である.PLCβ1のノックアウト(KO)マウスはてんかん様症状を示すことが報告されている25).またヒトにおいても早期発症てんかん性脳症の患者でPLCβ1の機能欠失型変異が確認されている26).統合失調症や躁うつ病患者の眼窩前頭皮質検体でPLCβ1が検出されないことも報告されており,PLCβ1 KOマウスでは成体神経新生の異常を伴う統合失調症様の行動異常が観察されている27–29).PLCβ4 KOマウスは運動失調,不安症,視覚異常,小脳における長期抑制の異常が観察される25, 30, 31).またPLCβ4の欠損は欠心発作を引き起こすことも報告されている32).さらにPLCβ4はC繊維侵害受容神経にも多く発現しておりPLCβ4 KOマウスではヒスタミン投与時の掻痒行動が抑制されることが報告されている33).
b.PLCβ2, PLCβ3による血球系細胞の機能制御
アレルギー反応に重要な細胞である肥満細胞はIgE高親和性Fc受容体であるFcεRIを介し活性化される.PLCβ3は肥満細胞におけるFcεRIシグナルに関与することが報告されている.PLCβ3 KOマウスの肥満細胞はFcεRI刺激によるサイトカイン産生が低下しており,遅延型アナフィラキシーや気道炎症が抑制される34).肥満細胞においてPLCβ3はFcεRIおよび,Lyn, Src homology region 2 domain-containing phosphatase-1(SHP-1)と結合する.この結合はFcεRIシグナルに対し抑制的に働くLynをSHP-1により脱リン酸化(不活性化)する反応を促進するため,PLCβ3欠損下ではLynが不活性化されずFcεRIシグナルが抑制されることが示唆されている.また,PLCβ3は造血幹細胞の増殖,生存,自己複製等の調節にも関与している.PLCβ3 KOマウスでは,骨髄中の造血幹細胞や骨髄系前駆細胞の数が増加しており,骨髄,脾臓,末梢血中における顆粒球の数が増加するという骨髄増殖性症候群様の症状を示す35).PLCβ3はC末端領域を介して,PLCβ3, SHP-1, STAT5(signal transducers and activator of transcription 5)三者複合体を形成し,この複合体中でSHP-1によるSTAT5の脱リン酸化が行われ,STAT5の過剰活性化が抑制されている.興味深いことにPLCβ3の酵素活性はこの現象には必要ではなく,PLCβ3はタンパク質複合体形成の足場として機能していると考えられている.PLCβ3 KOマウスでは,上記の三者複合体が形成されないため,STAT5が過剰活性化しており,これがPLCβ3 KOマウスが示す骨髄増殖性症候群様の異常の原因であることが示唆されている.PLCβ3 KOマウスではSTAT5の過剰活性化により肥満細胞やその前駆細胞の増殖も亢進しており,アトピー性皮膚炎に類似した皮膚炎症を自然発症することも報告されている36).さらにPLCβ3はマクロファージの生存にも関与している.PLCβ3 KOマウスではBcl-xLの発現低下により,マクロファージがアポトーシス刺激に対して過敏になっており,動脈硬化モデルマウスにおいてPLCβ3を欠損させると動脈硬化部位でのマクロファージのアポトーシスが亢進し,動脈硬化巣の大きさが抑えられることが報告されている37).PLCβ2はGタンパク質共役型化学誘引物質(ケモアトラクタント)受容体の下流で活性化されケモアトラクタントによるシグナル伝達を調節しており,PLCβ2 KOマウスの好中球ではケモアトラクタント刺激によるCa2+濃度上昇やスーパーオキシド産生が抑制されている38, 39).
1)組織分布,構造,活性化機構
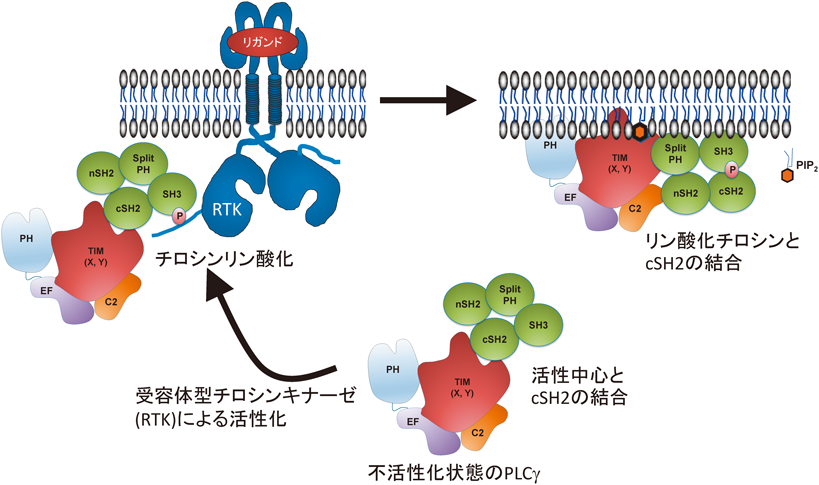
PLCγにはγ1, γ2の二つのアイソザイムが存在している.PLCγ1は多くの組織に発現がみられる一方,PLCγ2は主に血球系細胞に発現がみられる.PLCγはXドメインとYドメインの間に,スプリットPHドメインと二つのSrc homology(SH)2ドメイン,一つのSH3ドメインからなる特徴的な構造を有している.PLCγのC末端側のSH2ドメインはPLCγの活性中心と結合しており,PLCγを不活性化状態に保つ役割を持っている40–42).一方で,PLCγはN末端側のSH2ドメインを介して受容体型チロシンキナーゼと結合し43),PLCγの保存されたチロシン残基がリン酸化を受ける44, 45).このようにしてリン酸化を受けたPLCγのチロシン残基はC末端側のSH2ドメインと結合するため,C末端側のSH2ドメインが活性中心から除かれ,PLCγが活性化する46)(図4).PLCγ2のスプリットPHドメインはRac1, Rac2と結合することによりPLCγ2の細胞膜移行を促し,PLCγ2を活性化する47–49).PLCγはPIP3との結合によっても活性化される.PIP3によるPLCγの活性化にはスプリットPHドメインのN末端側の部分とSH2ドメインが必要であり,PIP3はPLCγを細胞膜のPIP3が豊富に存在する領域にリクルートし活性化するものと考えられる50–52).以上のようにPLCγの活性調節にはXドメインとYドメインの間に存在する特徴的な構造が重要な役割を担っている53).
2)生理機能
a.PLCγ1によるT細胞受容体(T cell receptor:TCR)シグナルを介したT細胞の発生,機能制御
PLCγ1 KOマウスは赤血球形成と脈管形成の異常を伴い54)胎生9日目において致死となる55).またPLCγ1のキメラKOマウスでは,PLCγ1を欠損した細胞がリンパや造血系組織に寄与できないことが判明しており,PLCγ1が造血幹細胞の発生や維持に関与することが示唆されている56).T細胞特異的なPLCγ1 KOマウスでは胸腺におけるT細胞の発生,分化に異常が生じ,末梢T細胞の減少がみられる57).これらのマウスのT細胞では,TCRシグナル依存性の細胞増殖やサイトカイン産生が抑制されている.さらに,このマウスでは制御性T細胞の発達や機能が阻害され,自己免疫疾患様の表現型がみられる.PLCγ1はTCR刺激によりリン酸化を受けたlinker for activation of T cell(LAT)に結合する.PLCγ1との結合ができないLAT変異体のノックインマウスにおいても制御性T細胞の減少や機能低下を伴う自己免疫疾患の発症が観察されており58, 59),PLCγ1のLATとの結合が制御性T細胞の発生に必要であると考えられる.
b.PLCγ2によるB細胞受容体(B cell receptor:BCR)やFc受容体シグナルを介した血球系細胞の機能制御
PLCγ2はB細胞の分化において重要な役割を担っている.PLCγ2 KOマウスB細胞ではプレBCRシグナルが抑制されており,B細胞の分化がproBからpreBの段階で阻害されている60).PLCγ2の欠損により血球系細胞におけるreceptor activator of NFκB ligandシグナルの抑制もみられ,二次リンパ組織の形成や破骨細胞の分化が抑制されることも報告されている61).また,骨髄由来細胞におけるPLCγ2の欠損は血管とリンパ管の分岐異常を引き起こすことも判明している62).PLCγ2はFc受容体を介したシグナルの制御も行っており,PLCγ2 KOマウスでは血小板の凝集,肥満細胞の脱顆粒,好中球の活性化,ナチュラルキラー(natural killer:NK)細胞の細胞傷害性などのFc受容体を介したシグナルが関与する現象が抑制されていることも判明している63).PLCγ2の機能獲得型のマウスも報告されている64).N-エチル-N-ニトロソ尿素変異導入により自己免疫疾患様の表現型を示すPLCγ2の機能獲得型変異マウスが見いだされた.このマウスでは,PLCγ2の993番目のアスパラギン酸がグリシンに変化することによりPLCγ2の細胞膜への結合能が増加し,B細胞や自然免疫細胞の過剰活性化が誘導されていることが示唆されている.
1)組織分布,構造,活性化機構
PLCδはδ1, δ3, δ4の三つのアイソザイムが存在している.ウシにおいてPLCδ2の遺伝子のクローニングが報告されていたが,この遺伝子はラット,マウス,ヒトにおいてクローニングされ機能解析が進んでいたPLCδ4遺伝子のオーソログであることが,後になり明らかになった65).これら三つのPLCδアイソザイムは多くの組織に発現がみられる.PLCδは各サブタイプが持つ共通のドメイン構造のみからなっており,PLCδが他のサブタイプのプロトタイプであると考えられる.PLCδ,特にPLCδ1のPHドメインは他のPLCサブタイプのPHドメインと比較して基質であるPIP2と高い親和性を持っており,PHドメインを介したPIP2との結合はPLCδ1の細胞膜移行に特に重要である(図2)66–72).C2ドメインやTIMバレルへのCa2+イオンの結合もPLCδの細胞膜への移行を促進し,PLCδの活性化に寄与している73–77).このようにPLCδは基質やCa2+イオンとの結合により活性化される.
2)生理機能
a.PLCδ1による多様な組織の機能制御
PLCδ1は広い組織に分布しているため,PLCδ1はさまざまな組織の機能調節に関与する.我々はPLCδ1が皮膚の最外層に存在する表皮角化細胞に多く発現しており,表皮角化細胞の分化を調節していることを明らかにしてきた78).表皮角化細胞は分化により皮膚表面に角質層を形成し,外界と体内を隔てる強固なバリアとして機能する.PLCδ1は分化した表皮角化細胞に多く発現しており,PLCδ1 ΚΟマウスでは角質層の形成が正常に行われずバリア機能の低下が観察される.またPLCδ1を欠損したマウスでは,表皮角化細胞が特殊な様式の分化をすることで形成される皮膚付属器官である毛包において,毛を構成するケラチンが正常に産生されず毛の構造が異常になり,毛が体表まで出てこなくなることも明らかにしてきた79).PLCδ1 ΚΟマウスと同様な毛包異常を示すマウスとして免疫不全モデルマウスであるヌードマウスが知られており,我々の解析によりヌードマウスで機能欠損している転写因子FoxN1によりPLCδ1の転写が誘導されることが明らかになった.さらにPLCδ1 KOマウスにおいてFoxN1プロモーターの制御下でPLCδ1を発現させることによりPLCδ1 KOマウスの毛の形成が正常化されることも判明し,PLCδ1がFoxN1の下流で働き毛の正常な形成に不可欠な働きをしていることを明らかにした80).また,自然発生変異マウスであるOligotricheマウスの体毛形成異常の原因遺伝子がPLCδ1であることも判明している81).爪も表皮角化細胞の分化により生じる組織であり,その分化不全は爪白斑の原因となる.遺伝性爪白斑においてPLCδ1遺伝子の変異が報告されており,この変異はPLCδ1の酵素活性を低下させるものであった82).以上のようにPLCδ1は角質層や毛,爪といった表皮角化細胞の分化により作られる組織の正常な形成に必須なPLCアイソザイムである.表皮角化細胞にはPLCδ1以外のPLCアイソザイムも存在しているにも関わらず,PLCδ1が欠損,変異するのみで上述のような表皮角化細胞の分化異常が生じるのは意外であった.そこでPLCδ1 KOマウスの表皮細胞における全PLC活性を調べたところPLCδ1を欠損したのみで全PLC活性の90%以上が失われており,PLCδ1が表皮のPLC活性の大半を占める主要なPLCアイソザイムであることが判明した83).表皮角化細胞は分化に伴い細胞内のCa2+濃度が上昇する.また表皮角化細胞の分化にはPLC下流シグナルが重要な役割をしている.そのためCa2+濃度の上昇を鋭敏に感知し活性化されるPLCδ1は表皮角化細胞の分化調節に適したPLCアイソザイムであると考えられる.表皮角化細胞は角質バリアの破壊や外部からの各種の刺激に応じてさまざまなサイトカインやケモカインを産生することで免疫反応を調節しており,その調節異常は各種炎症性皮膚疾患の一因となる.我々は表皮角化細胞特異的にPLCδ1を欠損したマウスが白血球の浸潤や炎症性サイトカインのインターロイキン(IL)-23,IL-17の発現増加を伴う自然発生的な皮膚炎症を示すことも明らかにした83, 84).皮膚におけるIL-23やIL-17の増加はヒト尋常性乾癬の特徴の一つである.実際に尋常性乾癬患者表皮においてPLCδ1の減少が観察され,ヒトにおいてもPLCδ1の減少が尋常性乾癬の発症や悪化に関与している可能性が考えられる.また,表皮角化細胞特異的にPLCδ1を欠損したマウスではIL-17依存的に接触皮膚炎の増悪も観察される.驚いたことに表皮角化細胞特異的にPLCδ1を欠損したマウスでは上記の皮膚の異常に加え末梢血,脾臓,骨髄において顆粒球増多を示しており,皮膚におけるIL-17の過剰産生がその原因であることも筆者らは示唆してきた83).
PLCδ1は血管平滑筋の収縮調節に関与することも報告されている.冠動脈の血管平滑筋の過剰な収縮が一因となる疾患である冠動脈攣縮患者の一部ではPLCδ1の257番目のアルギニンがヒスチジンへと変異しており,この変異はPLCδ1の酵素活性を上昇させる85).実際に平滑筋特異的に上記変異体PLCδ1を発現するトランスジェニックマウスはエルゴメトリン投与により局所的な冠動脈攣縮や冠動脈灌流圧の上昇を示しており,PLCδ1の変異が冠動脈攣縮に関与することが強く示唆されている86).
筋萎縮性側索硬化症(ALS)との関連に着目したマイクロサテライト解析においてPLCδ1遺伝子中に存在するマイクロサテライトマーカーがALSと関連していることが報告されている.興味深いことにALSモデルマウスの脊髄神経細胞ではPLCδ1の発現が増加している一方で,PLCδ1を欠損したALSマウスの運動神経細胞では核萎縮が抑制され生存期間の延長が認められる.このことからPLCδ1がALSの進行に関与している可能性が示唆されている87).我々はPLCδ1が脂肪細胞の機能調節においても重要な役割を果たしていることも明らかにしてきた.PLCδ1 KOマウスでは高脂肪食による体重増加やインスリン抵抗性が抑制される.PLCδ1の減少により白色脂肪細胞における脂肪産生が抑制される一方,褐色脂肪細胞における熱産生は促進されることが明らかになっている88).
b.PLCδ4による受精時の精子先体反応制御
PLCδ4は精子に多く存在しており受精に必須なPLCアイソザイムである.PLCδ4を欠損したマウスは受精において精子が透明帯を通過し卵と融合する際に必須な精子先体反応が抑制されており雄性不妊となる89).精子先体反応の開始には精子内のCa2+濃度上昇が必要であり,PLCδ4欠損マウスではこのCa2+濃度上昇が阻害されている90).
c.PLCδ1とPLCδ3による胎盤や心臓における細胞生存の制御
マウス胎仔の脳でPLCδ3を急性的に発現抑制することにより発生過程において大脳皮質の神経細胞の移動に遅れが観察された91).PLCδ3はRhoAの発現を負に調節することにより神経突起の形成を促進しており,PLCδ3の減少によりこの現象が阻害されることが大脳皮質の神経細胞の移動の遅れと関連していることが示唆されている.PLCδ3はPLCδ1同様に多くの組織に存在しているにも関わらず,PLCδ3を欠損したマウスでは目立った異常は観察されなかった.これはPLCδ1がPLCδ3の欠損を補なってしまっているためではないかと考え,PLCδ1とPLCδ3を同時に欠損したマウスを作製した.これらのマウスでは母体胎仔間の栄養交換を担う組織である胎盤の栄養膜細胞においてアポトーシスが亢進し,胎盤ラビリンス層の血管形成不全により胎生中期に致死となる92).さらに胎仔のみでPLCδ1とPLCδ3両遺伝子を欠損し,胎盤を含む胎仔外組織では両遺伝子が存在しているマウスを作製したところ,これらのマウスでは心筋細胞の過剰なアポトーシスにより心臓の線維化や心機能の低下が観察された93).これらのことからPLCδ1とPLCδ3は胎盤や心臓といった個体の発生や生存に不可欠である組織における細胞生存に必須な酵素であることが明らかになった.
1)組織分布,構造,活性化機構
PLCεは心臓において最も発現が高いが,その他のさまざまな組織においても発現がみられる.PLCεはPLC各サブタイプに共通なドメイン構造に加えてCDC25相同ドメイン,Ras-Associating(RA)1ドメイン,RA2ドメインを持つ.このうちRA2ドメインはGTP結合型のRapやRasと結合し,PLCεを活性化する94–96).RasやRapとの結合はPLCεの細胞内での局在を変化させており,RasはPLCεを形質膜に移行させる一方95),RapはPLCεを核周辺部に移行させる97).GTP結合型のRhoAもPLCεのYドメインと結合し,PLCεを活性化する98).リゾホスファチジン酸(lysophosphatidic acid:LPA),トロンビン,スフィンゴシン1-リン酸(sphingosine 1-phosphate:S1P)などのリガンドがGα12やGα13と共役した受容体に結合するとRhoグアニンヌクレオチド交換因子(guanine nucleotide exchange factor:GEF)の活性化を介し,GTP結合型のRhoAが増加し,PLCεは活性化される.またPLCεはRap1やRasのGEFとして働きRap1やRasの活性化を促進する.前述のようにGTP結合型のRap1はPLCεを活性化するため,Rap1–PLCε経路には正のフィードバック機構が存在し,Rap1はPLCεを持続的に活性化する97, 99, 100).以上のようにPLCεは低分子量Gタンパク質との間の双方向制御によって活性が調節されている.
2)生理機能
PLCεの組織分布からも推測できるようにPLCεは心臓の発生や機能の調節において重要な役割をしている.PLCεの酵素活性領域を欠く遺伝子改変マウスでは心臓弁の形成異常により軽度の血管狭窄を伴う弁閉鎖不全が引き起こされる101).また,PLCεを発現抑制したラット新生仔心筋細胞では心肥大誘導刺激に対する反応性が低下する.さらに心筋細胞特異的にPLCεを欠損したマウスでも圧負荷により誘導される心肥大の抑制がみられる102).心筋細胞においてPLCεは刺激に応じて核周辺のゴルジ体のホスファチジルイノシトール4-リン酸(PI4P)を分解しDAGを産生することで心肥大誘導に重要な役割を果たすプロテインキナーゼD(protein kinase D:PKD)を活性化する.興味深いことにヒトの特発性拡張型心筋症患者の心臓ではPLCεのmRNAが増加しており102),ヒトの心疾患にもPLCεが関与している可能性が高い.PLCεの酵素活性領域を欠く遺伝子改変マウスの皮膚では2,4-ジニトロフルオロベンゼンに対する接触皮膚炎103)や紫外線104),12-O-tetradecanoylphorbol-13-acetate(TPA)処理による皮膚炎105)が抑制されており,7,12-ジメチルベンズ[a]アントラセンとTPAによる皮膚二段階発がんに対しては抵抗性を示す106).一方,表皮角化細胞特異的にPLCεを過剰発現したマウスでは炎症性サイトカインIL-23, IL-22の産生増加がみられ,ヒト尋常性乾癬と一部類似した皮膚炎症が観察される107).PLCεは表皮角化細胞においてNFκBシグナルを活性化するため,このことがPLCεの炎症誘導作用の一因であることが示唆されている108).PLCεは神経組織の炎症にも関与することが報告されており,PLCεを欠損したマウスでは脳の損傷時に炎症性遺伝子の発現が抑制される109).以上のようにPLCεは皮膚や脳における炎症反応を正に制御する役割を持つと考えられる.
1)組織分布,構造,活性化機構
PLCζは精子に存在しており,すべてのPLCの中で唯一,PHドメインを持たないPLCアイソザイムである.PLCζはCa2+に対する感受性が高く,30 nM程度のCa2+濃度下においても活性化される.このようなPLCζの高いCa2+感受性にはEFハンドモチーフが重要な役割をしている110).通常,PLCのX–Yリンカー部位は負に荷電しており,PLCの活性中心をマスクし,不活性化状態に保つ役割をしているが,PLCζのX–Yリンカーは他のPLCサブタイプと異なり正に荷電している.そのため,PLCζのX–YリンカーはPLCζの基質であり負電荷を持つPIP2との結合を促進しPLCζを恒常的に活性化状態にしていると考えられている111).
2)生理機能
組織分布が示すようにPLCζは精子の機能調節において重要な役割を果たしている.受精の瞬間には卵でCa2+オシレーションと呼ばれる周期的なCa2+の上昇が観察される.PLCζは精子由来のCa2+オシレーション誘導因子の有力な候補として同定された112).PLCζに対するshRNAを発現するトランスジェニックマウスが作製されており,これらのマウスの精子の受精時には,Ca2+オシレーションの持続性が低下し卵の活性化を効率的に誘導できないことが判明している113).また,全身でPLCζを発現するトランスジェニックマウスも作製されており,これらのマウスでは,未受精卵において自発的なCa2+オシレーションが生じ,極体が放出され,前核形成が行われる114).ヒトの男性不妊患者の精子においてもPLCζの欠損や局在異常が報告されている115).また男性不妊患者ではPLCζの398番目のヒスチジンのプロリンへの変異や233番目のヒスチジンのロイシンへの変異がみられており,これらの変異型PLCζはマウス卵へ注入した際に正常なCa2+オシレーションを誘導できないことが判明している116, 117).
引用文献References
1) Berridge, M.J. & Irvine, R.F. (1984) Nature, 312, 315–321.
2) Nishizuka, Y. (1988) Nature, 334, 661–665.
3) Fukami, K., Inanobe, S., Kanemaru, K., & Nakamura, Y. (2010) Prog. Lipid Res., 49, 429–437.
4) Hicks, S.N., Jezyk, M.R., Gershburg, S., Seifert, J.P., Harden, T.K., & Sondek, J. (2008) Mol. Cell, 31, 383–394.
5) Suh, P.G., Park, J.I., Manzoli, L., Cocco, L., Peak, J.C., Katan, M., Fukami, K., Kataoka, T., Yun, S., & Ryu, S.H. (2008) BMB Rep., 41, 415–434.
6) Kadamur, G. & Ross, E.M. (2013) Annu. Rev. Physiol., 75, 127–154.
7) Jiang, H., Wu, D., & Simon, M.J. (1994) J. Biol. Chem., 269, 7593–7596.
8) Lee, C.W., Lee, K.H., Lee, S.B., Park, D., & Rhee, S.G. (1994) J. Biol. Chem., 269, 25335–25338.
9) Wang, T., Dowal, L., El-Maghrabi, M.R., Rebecchi, M., & Scarlata, S. (2000) J. Biol. Chem., 275, 7466–7469.
10) Illenberger, D., Schwald, F., Pimmer, D., Binder, W., Maier, G., Dietrich, A., & Gierschik, P. (1998) EMBO J., 17, 6241–6249.
11) Snyder, J.T., Singer, A.U., Wing, M.R., Harden, T.K., & Sondek, J. (2003) J. Biol. Chem., 278, 21099–21104.
12) Jezyk, M.R., Snyder, J.T., Gershberg, S., Worthylake, D.K., Harden, T.K., & Sondek, J. (2006) Nat. Struct. Mol. Biol., 13, 1135–1140.
13) Lee, S.B., Shin, S.H., Hepler, J.R., Gilman, A.G., & Rhee, S.G. (1993) J. Biol. Chem., 268, 25952–25957.
14) Kim, C.G., Park, D., & Rhee, S.G. (1996) J. Biol. Chem., 271, 21187–21192.
15) Waldo, G.L., Ricks, T.K., Hicks, S.N., Cheever, M.L., Kawano, T., Tsuboi, K., Wang, X., Montell, C., Kozasa, T., Sondek, J., & Harden, T.K. (2010) Science, 330, 974–980.
16) Lyon, A.M., Tesmer, V.M., Dhamsania, V.D., Thal, D.M., Gutierrez, J., Chowdhury, S., Suddala, K.C., Northup, J.K., & Tesmer, J.J. (2011) Nat. Struct. Mol. Biol., 18, 999–1005.
17) Lyon, A.M., Dutta, S., Boguth, C.A., Skiniotis, G., & Tesmer, J.J. (2013) Nat. Struct. Mol. Biol., 20, 355–362.
18) Singer, A.U., Waldo, G.L., Harden, T.K., & Sondek, J. (2002) Nat. Struct. Biol., 9, 32–36.
19) Lyon, A.M. & Tesmer, J.J. (2013) Mol. Pharmacol., 84, 488–500.
20) Wu, D., Jiang, H., Katz, A., & Simon, M.I. (1993) J. Biol. Chem., 268, 3704–3709.
21) Ilkaeva, O., Kinch, L.N., Paulssen, R.H., & Ross, E.M. (2002) J. Biol. Chem., 277, 4294–4300.
22) Paulssen, R.H., Woodson, J., Liu, Z., & Ross, E.M. (1996) J. Biol. Chem., 271, 26622–26629.
23) Inaba, T., Kishimoto, T., Murate, M., Tajima, T., Sakai, S., Abe, M., Makino, A., Tomishige, N., Ishitsuka, R., Ikeda, Y., Takeoka, S., & Kobayashi, T. (2016) Proc. Natl. Acad. Sci. USA, 113, 7834–7839.
24) Kim, J.K., Lim, S., Kim, J., Kim, S., Kim, J.H., Ryu, S.H., & Suh, P.G. (2011) Adv. Enzyme Regul., 51, 138–151.
25) Kim, D., Jun, K.S., Lee, S.B., Kang, N.G., Min, D.S., Kim, Y.H., Ryu, S.H., Suh, P.G., & Shin, H.S. (1997) Nature, 389, 290–293.
26) Kurian, M.A., Meyer, E., Vassallo, G., Morgan, N.V., Prakash, N., Pasha, S., Hai, N.A., Shuib, S., Rahman, F., Wassmer, E., Cross, J.H., O’Callaghan, F.J., Osborne, J.P., Scheffer, I.E., Gissen, P., & Maher, E.R. (2010) Brain, 133, 2964–2970.
27) Lo Vasco, V.R., Cardinale, G., & Polonia, P. (2012) J. Cell. Mol. Med., 16, 844–851.
28) Lo Vasco, V.R., Longo, L., & Polonia, P. (2013) J. Cell Commun. Signal., 7, 25–29.
29) Manning, E.E., Ransome, M.I., Burrows, E.L., & Hannan, A.J. (2012) Hippocampus, 22, 309–319.
30) Jiang, H., Lyubarsky, A., Dodd, R., Vardi, N., Pugh, E., Baylor, D., Simon, M.I., & Wu, D. (1996) Proc. Natl. Acad. Sci. USA, 93, 14598–14601.
31) Hirono, M., Sugiyama, T., Kishimoto, Y., Sakai, I., Miyazawa, T., Kishio, M., Inoue, H., Nakao, K., Ikeda, M., Kawahara, S., Kirino, Y., Katsuki, M., Horie, H., Ishikawa, Y., & Yoshioka, T. (2001) J. Biol. Chem., 276, 45236–45242.
32) Cheong, E., Zheng, Y., Lee, K., Lee, J., Kim, S., Sanati, M., Lee, S., Kim, Y.S., & Shin, H.S. (2009) Proc. Natl. Acad. Sci. USA, 106, 21912–21917.
33) Han, S.K., Mancino, V., & Simon, M.I. (2006) Neuron, 52, 691–703.
34) Xiao, W., Kashiwakura, J., Hong, H., Yasudo, H., Ando, T., Maeda-Yamamoto, M., Wu, D., Kawakami, Y., & Kawakami, T. (2011) Immunity, 34, 893–904.
35) Xiao, W., Hong, H., Kawakami, Y., Kato, Y., Wu, D., Yasudo, H., Kimura, A., Kubagawa, H., Bertoli, L.F., Davis, R.S., Chau, L.A., Madrenas, J., Hsia, C.C., Xenocostas, A., Kipps, T.J., Hennighausen, L., Iwama, A., Nakauchi, H., & Kawakami, T. (2009) Cancer Cell, 16, 161–171.
36) Ando, T., Xiao, W., Gao, P., Namiranian, S., Matsumoto, K., Tomimori, Y., Hong, H., Yamashita, H., Kimura, M., Kashiwakura, J., Hata, T.R., Izuhara, K., Gurish, M.F., Roers, A., Rafaels, N.M., Barnes, K.C., Jamora, C., Kawakami, Y., & Kawakami, T. (2014) Cell Reports, 6, 366–376.
37) Wang, Z., Liu, B., Wang, P., Dong, X., Fernandez-Hernando, C., Li, Z., Hla, T., Li, Z., Claffey, K., Smith, J.D., & Wu, D. (2008) J. Clin. Invest., 118, 195–204.
38) Jiang, H., Kuang, Y., Wu, Y., Xie, W., Simon, M.I., & Wu, D. (1997) Proc. Natl. Acad. Sci. USA, 94, 7971–7975.
39) Li, Z., Jiang, H., Xie, W., Zhang, Z., Smrcka, A.V., & Wu, D. (2000) Science, 287, 1046–1049.
40) Everett, K.L., Buehler, A., Bunney, T.D., Margineanu, A., Baxendale, R.W., Vatter, P., Retlich, M., Walliser, C., Manning, H.B., Neil, M.A., Dunsby, C., French, P.M., Gierschik, P., & Katan, M. (2011) Mol. Cell. Biol., 31, 1240–1251.
41) Gresset, A., Hicks, S.N., Harden, T.K., & Sondek, J. (2010) J. Biol. Chem., 285, 35836–35847.
42) DeBell, K., Graham, L., Reischl, I., Serrano, C., Bonvini, E., & Rellahan, B. (2007) Mol. Cell. Biol., 27, 854–863.
43) Bae, J.H., Lew, E.D., Yuzawa, S., Tomé, F., Lax, I., & Schlessinger, J. (2009) Cell, 138, 514–524.
44) Kim, H.K., Kim, J.W., Zilberstein, A., Margolis, B., Kim, J.G., Schlessinger, J., & Rhee, S.G. (1991) Cell, 65, 435–441.
45) Wahl, M.I., Daniel, T.O., & Carpenter, G. (1988) Science, 241, 968–970.
46) Poulin, B., Sekiya, F., & Rhee, S.G. (2005) Proc. Natl. Acad. Sci. USA, 102, 4276–4281.
47) Piechulek, T., Rehlen, T., Walliser, C., Vatter, P., Moepps, B., & Gierschik, P. (2005) J. Biol. Chem., 280, 38923–38931.
48) Walliser, C., Retlich, M., Harris, R., Everett, K.L., Josephs, M.B., Vatter, P., Esposito, D., Driscoll, P.C., Katan, M., Gierschik, P., & Bunney, T.D. (2008) J. Biol. Chem., 283, 30351–30362.
49) Everett, K.L., Buehler, A., Bunney, T.D., Margineanu, A., Baxendale, R.W., Vatter, P., Retlich, M., Walliser, C., Manning, H.B., Neil, M.A., Dunsby, C., French, P.M., Gierschik, P., & Katan, M. (2011) Mol. Cell. Biol., 31, 1240–1251.
50) Bae, Y.S., Cantley, L.G., Chen, C.S., Kim, S.R., Kwon, K.S., & Rhee, S.G. (1998) J. Biol. Chem., 273, 4465–4469.
51) Falasca, M., Logan, S.K.L., Lehto, V.P., Baccante, G., Lemmon, M.A., & Schlessinger, J. (1998) EMBO J., 17, 414–422.
52) Gratacap, M.-P., Payrastre, B., Viala, C., Mauco, G., Plantavid, M., & Chap, H. (1998) J. Biol. Chem., 273, 24314–24321.
53) Koss, H., Bunney, T.D., Behjati, S., & Katan, M. (2014) Trends Biochem. Sci., 39, 603–611.
54) Liao, H.J., Kume, T., McKay, C., Xu, M.J., Ihle, J.N., & Carpenter, G. (2002) J. Biol. Chem., 277, 9335–9341.
55) Ji, Q.S., Winnier, G.E., Niswender, K.D., Horstman, D., Wisdom, R., Magnuson, M.A., & Carpenter, G. (1997) Proc. Natl. Acad. Sci. USA, 94, 2999–3003.
56) Shirane, M., Sawa, H., Kobayashi, Y., Nakano, T., Kitajima, K., Shinkai, Y., Nagashima, K., & Negishi, I. (2001) Development, 128, 5173–5180.
57) Fu, G., Chen, Y., Yu, M., Podd, A., Schuman, J., He, Y., Di, L., Yassai, M., Haribhai, D., North, P.E., Gorski, J., Williams, C.B., Wang, D., & Wen, R. (2010) J. Exp. Med., 207, 309–318.
58) Koonpaew, S., Shen, S., Flowers, L., & Zhang, W. (2006) J. Exp. Med., 203, 119–129.
59) Chuck, M.I., Zhu, M., Shen, S., & Zhang, W. (2010) J. Immunol., 184, 2476–2486.
60) Hashimoto, A., Takeda, K., Inaba, M., Sekimata, M., Kaisho, T., Ikehara, S., Homma, Y., Akira, S., & Kurosaki, T. (2000) J. Immunol., 165, 1738–1742.
61) Chen, Y., Wang, X., Di, L., Fu, G., Chen, Y., Bai, L., Liu, J., Feng, X., McDonald, J.M., Michalek, S., He, Y., Yu, M., Fu, Y.X., Wen, R., Wu, H., & Wang, D. (2008) J. Biol. Chem., 283, 29593–29601.
62) Ichise, H., Ichise, T., Ohtani, O., & Yoshida, N. (2009) Development, 136, 191–195.
63) Caraux, A., Kim, N., Bell, S.E., Zompi, S., Ranson, T., Lesjean-Pottier, S., Garcia-Ojeda, M.E., Turner, M., & Colucci, F. (2006) Blood, 107, 994–1002.
64) Yu, P., Constien, R., Dear, N., Katan, M., Hanke, P., Bunney, T.D., Kunder, S., Quintanilla-Martinez, L., Huffstadt, U., Schröder, A., Jones, N.P., Peters, T., Fuchs, H., de Angelis, M.H., Nehls, M., Grosse, J., Wabnitz, P., Meyer, T.P., Yasuda, K., Schiemann, M., Schneider-Fresenius, C., Jagla, W., Russ, A., Popp, A., Josephs, M., Marquardt, A., Laufs, J., Schmittwolf, C., Wagner, H., Pfeffer, K., & Mudde, G.C. (2005) Immunity, 22, 451–465.
65) Irino, Y., Cho, H., Nakamura, Y., Nakahara, M., Furutani, M., Suh, P.G., Takenawa, T., & Fukami, K. (2004) Biochem. Biophys. Res. Commun., 320, 537–543.
66) Lomasney, J.W., Cheng, H.F., Wang, L.P., Kuan, Y.S., Liu, S.M., Fesik, S.W., & King, K. (1996) J. Biol. Chem., 271, 25316–25326.
67) Lemmon, M.A., Ferguson, K.M., O’Brien, R., Sigler, P.B., & Schlessinger, J. (1995) Proc. Natl. Acad. Sci. USA, 92, 10472–10476.
68) Cifuentes, M.E., Honkanen, L., & Rebecchi, M.J. (1993) J. Biol. Chem., 268, 11586–11593.
69) Ferguson, K.M., Lemmon, M.A., Schlessinger, J., & Sigler, P.B. (1995) Cell, 83, 1037–1046.
70) Yagisawa, H., Sakuma, K., Paterson, H.F., Cheung, R., Allen, V., Hirata, H., Watanabe, Y., Hirata, M., Williams, R.L., & Katan, M. (1998) J. Biol. Chem., 273, 417–424.
71) Yagisawa, H., Hirata, M., Kanematsu, T., Watanabe, Y., Ozaki, S., Sakuma, K., Tanaka, H., Yabuta, N., Kamata, H., Hirata, H., et al. (1994) J. Biol. Chem., 269, 20179–20188.
72) Fujii, M., Ohtsubo, M., Ogawa, T., Kamata, H., Hirata, H., & Yagisawa, H. (1999) Biochem. Biophys. Res. Commun., 254, 284–291.
73) Allen, V., Swigart, P., Cheung, R., Cockcroft, S., & Katan, M. (1997) Biochem. J., 327, 545–552.
74) Kim, Y.H., Park, T.J., Lee, Y.H., Baek, K.J., Suh, P.G., Ryu, S.H., & Kim, K.T. (1999) J. Biol. Chem., 274, 26127–26134.
75) Ananthanarayanan, B., Das, S., Rhee, S.G., Murray, D., & Cho, W. (2002) J. Biol. Chem., 277, 3568–3575.
76) Lomasney, J.W., Cheng, H.F., Roffler, S.R., & King, K. (1999) J. Biol. Chem., 274, 21995–22001.
77) Lomasney, J.W., Cheng, H.F., Kobayashi, M., & King, K. (2012) Biochemistry, 51, 2246–2257.
78) Nakamura, Y., Kanemaru, K., & Fukami, K. (2013) Adv. Biol. Regul., 53, 356–362.
79) Nakamura, Y., Fukami, K., Yu, H., Takenaka, K., Kataoka, Y., Shirakata, Y., Nishikawa, S.I., Hashimoto, K., Yoshida, N., & Takenawa, T. (2003) EMBO J., 22, 2981–2991.
80) Nakamura, Y., Ichinohe, M., Hirata, M., Matsuura, H., Fujiwara, T., Igarashi, T., Nakahara, M., Yamaguchi, H., Yasugi, S., Takenawa, T., & Fukami, K. (2008) FASEB J., 22, 841–849.
81) Runkel, F., Aubin, I., Simon-Chazottes, D., Büssow, H., Stingl, R., Miething, A., Fukami, K., Nakamura, Y., Guénet, J.L., & Franz, T. (2008) Mamm. Genome, 19, 691–702.
82) Kiuru, M., Kurban, M., Itoh, M., Petukhova, L., Shimomura, Y., Wajid, M., & Christiano, A.M. (2011) Am. J. Hum. Genet., 88, 839–844.
83) Kanemaru, K., Nakamura, Y., Sato, K., Kojima, R., Takahashi, S., Yamaguchi, M., Ichinohe, M., Kiyonari, H., Shioi, G., Kabashima, K., Nakahigashi, K., Asagiri, M., Jamora, C., Yamaguchi, H., & Fukami, K. (2012) Nat. Commun., 3, 963.
84) Ichinohe, M., Nakamura, Y., Sai, K., Nakahara, M., Yamaguchi, H., & Fukami, K. (2007) Biochem. Biophys. Res. Commun., 356, 912–918.
85) Nakano, T., Osanai, T., Tomita, H., Sekimata, M., Homma, Y., & Okumura, K. (2002) Circulation, 105, 2024–2029.
86) Shibutani, S., Osanai, T., Ashitate, T., Sagara, S., Izumiyama, K., Yamamoto, Y., Hanada, K., Echizen, T., Tomita, H., Fujita, T., Miwa, T., Matsubara, H., Homma, Y., & Okumura, K. (2012) Circulation, 125, 1027–1036.
87) Staats, K.A., Van Helleputte, L., Jones, A.R., Bento-Abreu, A., Van Hoecke, A., Shatunov, A., Simpson, C.L., Lemmens, R., Jaspers, T., Fukami, K., Nakamura, Y., Brown, R.H. Jr., Van Damme, P., Liston, A., Robberecht, W., Al-Chalabi, A., & Van Den Bosch, L. (2013) Neurobiol. Dis., 60, 11–17.
88) Hirata, M., Suzuki, M., Ishii, R., Satow, R., Uchida, T., Kitazumi, T., Sasaki, T., Kitamura, T., Yamaguchi, H., Nakamura, Y., & Fukami, K. (2011) Diabetes, 60, 1926–1937.
89) Fukami, K., Nakao, K., Inoue, T., Kataoka, Y., Kurokawa, M., Fissore, R.A., Nakamura, K., Katsuki, M., Mikoshiba, K., Yoshida, N., & Takenawa, T. (2001) Science, 292, 920–923.
90) Fukami, K., Yoshida, M., Inoue, T., Kurokawa, M., Fissore, R.A., Yoshida, N., Mikoshiba, K., & Takenawa, T. (2003) J. Cell Biol., 161, 79–88.
91) Kouchi, Z., Igarashi, T., Shibayama, N., Inanobe, S., Sakurai, K., Yamaguchi, H., Fukuda, T., Yanagi, S., Nakamura, Y., & Fukami, K. (2011) J. Biol. Chem., 286, 8459–8471.
92) Nakamura, Y., Hamada, Y., Fujiwara, T., Enomoto, H., Hiroe, T., Tanaka, S., Nose, M., Nakahara, M., Yoshida, N., Takenawa, T., & Fukami, K. (2005) Mol. Cell. Biol., 25, 10979–10988.
93) Nakamura, Y., Kanemaru, K., Kojima, R., Hashimoto, Y., Marunouchi, T., Oka, N., Ogura, T., Tanonaka, K., & Fukami, K. (2014) Cell Death Dis., 5, e1215.
94) Kelley, G.G., Reks, S.E., Ondrako, J.M., & Smrcka, A.V. (2001) EMBO J., 20, 743–754.
95) Song, C., Hu, C.D., Masago, M., Kariya, K., Yamawaki-Kataoka, Y., Shibatohge, M., Wu, D., Satoh, T., & Kataoka, T. (2001) J. Biol. Chem., 276, 2752–2757.
96) Bunney, T.D., Harris, R., Gandarillas, N.L., Josephs, M.B., Roe, S.M., Sorli, S.C., Paterson, H.F., Rodrigues-Lima, F., Esposito, D., Ponting, C.P., Gierschik, P., Pearl, L.H., Driscoll, P.C., & Katan, M. (2006) Mol. Cell, 21, 495–507.
97) Jin, T.G., Satoh, T., Liao, Y., Song, C., Gao, X., Kariya, K., Hu, C.D., & Kataoka, T. (2001) J. Biol. Chem., 276, 30301–30307.
98) Seifert, J.P., Wing, M.R., Snyder, J.T., Gershburg, S., Sondek, J., & Harden, T.K. (2004) J. Biol. Chem., 279, 47992–47997.
99) Lopez, I., Mak, E.C., Ding, J., Hamm, H.E., & Lomasney, J.W. (2001) J. Biol. Chem., 276, 2758–2765.
100) Smrcka, A.V., Brown, J.H., & Holz, G.G. (2012) Cell. Signal., 24, 1333–1334.
101) Tadano, M., Edamatsu, H., Minamisawa, S., Yokoyama, U., Ishikawa, Y., Suzuki, N., Saito, H., Wu, D., Masago-Toda, M., Yamawaki-Kataoka, Y., Setsu, T., Terashima, T., Maeda, S., Satoh, T., & Kataoka, T. (2005) Mol. Cell. Biol., 25, 2191–2199.
102) Zhang, L., Malik, S., Pang, J., Wang, H., Park, K.M., Yule, D.I., Blaxall, B.C., & Smrcka, A.V. (2013) Cell, 153, 216–227.
103) Hu, L., Edamatsu, H., Takenaka, N., Ikuta, S., & Kataoka, T. (2010) J. Immunol., 184, 993–1002.
104) Oka, M., Edamatsu, H., Kunisada, M., Hu, L., Takenaka, N., Sakaguchi, M., Kataoka, T., & Nishigori, C. (2011) Lab. Invest., 91, 711–718.
105) Ikuta, S., Edamatsu, H., Li, M., Hu, L., & Kataoka, T. (2008) Cancer Res., 68, 64–72.
106) Bai, Y., Edamatsu, H., Maeda, S., Saito, H., Suzuki, N., Satoh, T., & Kataoka, T. (2004) Cancer Res., 64, 8808–8810.
107) Takenaka, N., Edamatsu, H., Suzuki, N., Saito, H., Inoue, Y., Oka, M., Hu, L., & Kataoka, T. (2011) Eur. J. Immunol., 41, 202–213.
108) Harada, Y., Edamatsu, H., & Kataoka, T. (2011) Biochem. Biophys. Res. Commun., 414, 106–111.
109) Dusaban, S.S., Purcell, N.H., Rockenstein, E., Masliah, E., Cho, M.K., Smrcka, A.V., & Brown, J.H. (2013) Proc. Natl. Acad. Sci. USA, 110, 3609–3614.
110) Kouchi, Z., Shikano, T., Nakamura, Y., Shirakawa, H., Fukami, K., & Miyazaki, S. (2005) J. Biol. Chem., 280, 21015–21021.
111) Nomikos, M., Elgmati, K., Theodoridou, A., Georgillis, A., Gonzalez-Garcia, J.R., Nounesis, G., Swann, K., & Lai, F.A. (2011) Biochem. J., 438, 427–432.
112) Saunders, C.M., Larman, M.G., Parrington, J., Cox, L.J., Royse, J., Blayney, L.M., Swann, K., & Lai, F.A. (2002) Development, 129, 3533–3544.
113) Knott, J.G., Kurokawa, M., Fissore, R.A., Schultz, R.M., & Williams, C.J. (2005) Biol. Reprod., 72, 992–996.
114) Yoshida, N., Amanai, M., Fukui, T., Kajikawa, E., Brahmajosyula, M., Iwahori, A., Nakano, Y., Shoji, S., Diebold, J., Hessel, H., Huss, R., & Perry, A.C. (2007) Development, 134, 3941–3952.
115) Yoon, S.Y., Jellerette, T., Salicioni, A.M., Lee, H.C., Yoo, M.S., Coward, K., Parrington, J., Grow, D., Cibelli, J.B., Visconti, P.E., Mager, J., & Fissore, R.A. (2008) J. Clin. Invest., 118, 3671–3681.
116) Heytens, E., Parrington, J., Coward, K., Young, C., Lambrecht, S., Yoon, S.Y., Fissore, R.A., Hamer, R., Deane, C.M., Ruas, M., Grasa, P., Soleimani, R., Cuvelier, C.A., Gerris, J., Dhont, M., Deforce, D., Leybaert, L., & De Sutter, P. (2009) Hum. Reprod., 24, 2417–2428.
117) Kashir, J., Konstantinidis, M., Jones, C., Lemmon, B., Lee, H.C., Hamer, R., Heindryckx, B., Deane, C.M., De Sutter, P., Fissore, R.A., Parrington, J., Wells, D., & Coward, K. (2012) Hum. Reprod., 27, 222–231.
118) Nakahara, M., Shimozawa, M., Nakamura, Y., Irino, Y., Morita, M., Kudo, Y., & Fukami, K. (2005) J. Biol. Chem., 280, 29128–29134.
119) Zhou, Y., Sondek, J., & Harden, T.K. (2008) Biochemistry, 47, 4410–4417.
120) Kanemaru, K., Nakahara, M., Nakamura, Y., Hashiguchi, Y., Kouchi, Z., Yamaguchi, H., Oshima, N., Kiyonari, H., & Fukami, K. (2010) Gene Expr. Patterns, 10, 119–126.