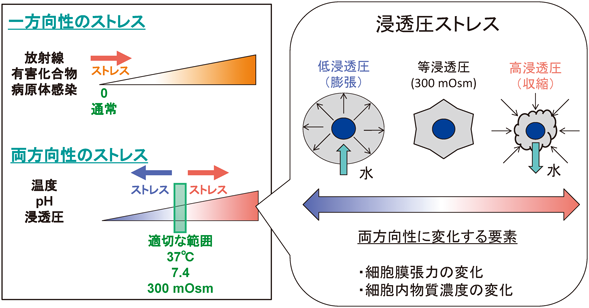
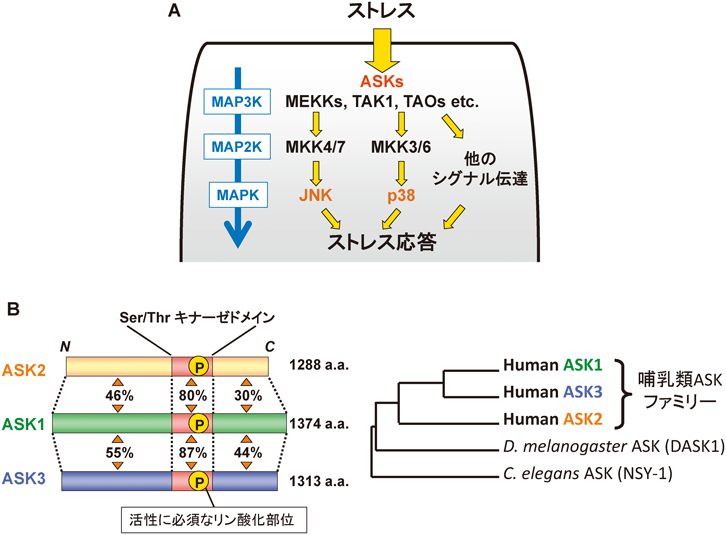
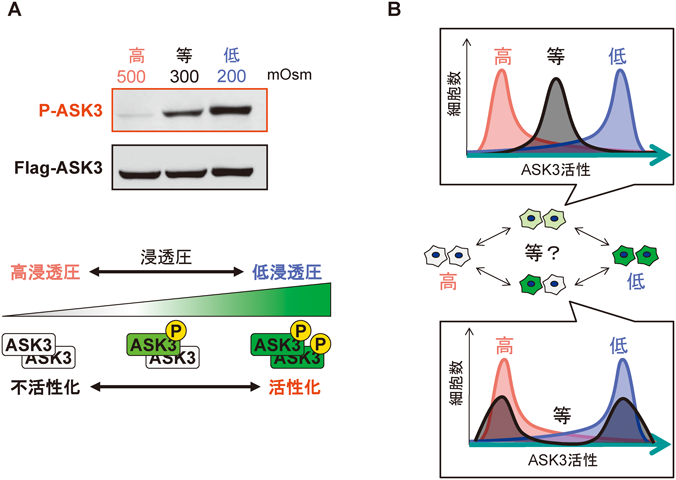
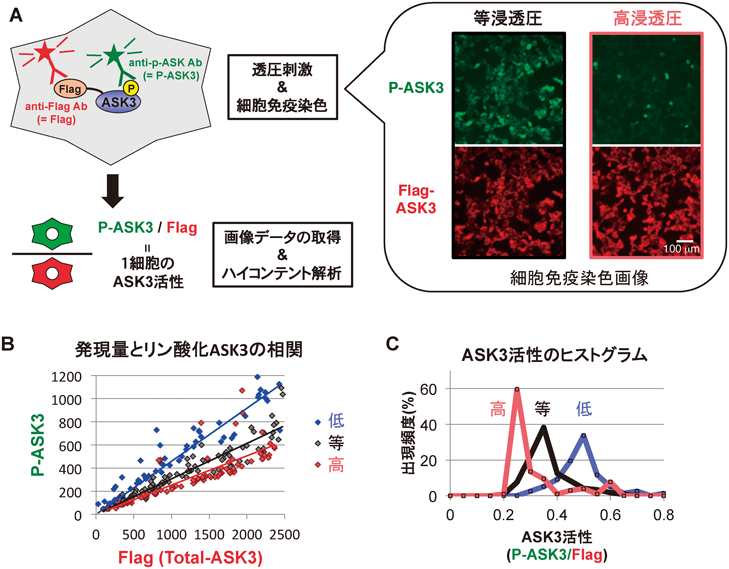
20) Iriyama, T., Takeda, K., Nakamura, H., Morimoto, Y., Kuroiwa, T., Mizukami, J., Umeda, T., Noguchi, T., Naguro, I., Nishitoh, H., Saegusa, K., Tobiume, K., Homma, T., Shimada, Y., Tsuda, H., Aiko, S., Imoto, I., Inazawa, J., Chida, K., Kamei, Y., Kozuma, S., Taketani, Y., Matsuzawa, A., & Ichijo, H. (2009) EMBO J., 28, 843–853.
34) Kahle, K.T., Schmouth, J.F., Lavastre, V., Latremoliere, A., Zhang, J., Andrews, N., Omura, T., Laganiere, J., Rochefort, D., Hince, P., Castonguay, G., Gaudet, R., Mapplebeck, J.C., Sotocinal, S.G., Duan, J., Ward, C., Khanna, A.R., Mogil, J.S., Dion, P.A., Woolf, C.J., Inquimbert, P., & Rouleau, G.A. (2016) Sci. Signal., 9, ra32.
38) Boyden, L.M., Choi, M., Choate, K.A., Nelson-Williams, C.J., Farhi, A., Toka, H.R., Tikhonova, I.R., Bjornson, R., Mane, S.M., Colussi, G., Lebel, M., Gordon, R.D., Semmekrot, B.A., Poujol, A., Valimaki, M.J., De Ferrari, M.E., Sanjad, S.A., Gutkin, M., Karet, F.E., Tucci, J.R., Stockigt, J.R., Keppler-Noreuil, K.M., Porter, C.C., Anand, S.K., Whiteford, M.L., Davis, I.D., Dewar, S.B., Bettinelli, A., Fadrowski, J.J., Belsha, C.W., Hunley, T.E., Nelson, R.D., Trachtman, H., Cole, T.R., Pinsk, M., Bockenhauer, D., Shenoy, M., Vaidyanathan, P., Foreman, J.W., Rasoulpour, M., Thameem, F., Al-Shahrouri, H.Z., Radhakrishnan, J., Gharavi, A.G., Goilav, B., & Lifton, R.P. (2012) Nature, 482, 98–102.
45) McCormick, J.A., Yang, C.L., Zhang, C., Davidge, B., Blankenstein, K.I., Terker, A.S., Yarbrough, B., Meermeier, N.P., Park, H.J., McCully, B., West, M., Borschewski, A., Himmerkus, N., Bleich, M., Bachmann, S., Mutig, K., Argaiz, E.R., Gamba, G., Singer, J.D., & Ellison, D.H. (2014) J. Clin. Invest., 124, 4723–4736.
46) Yamada, K., Park, H.M., Rigel, D.F., DiPetrillo, K., Whalen, E.J., Anisowicz, A., Beil, M., Berstler, J., Brocklehurst, C.E., Burdick, D.A., Caplan, S.L., Capparelli, M.P., Chen, G., Chen, W., Dale, B., Deng, L., Fu, F., Hamamatsu, N., Harasaki, K., Herr, T., Hoffmann, P., Hu, Q.Y., Huang, W.J., Idamakanti, N., Imase, H., Iwaki, Y., Jain, M., Jeyaseelan, J., Kato, M., Kaushik, V.K., Kohls, D., Kunjathoor, V., LaSala, D., Lee, J., Liu, J., Luo, Y., Ma, F., Mo, R., Mowbray, S., Mogi, M., Ossola, F., Pandey, P., Patel, S.J., Raghavan, S., Salem, B., Shanado, Y.H., Trakshel, G.M., Turner, G., Wakai, H., Wang, C., Weldon, S., Wielicki, J.B., Xie, X., Xu, L., Yagi, Y.I., Yasoshima, K., Yin, J., Yowe, D., Zhang, J.H., Zheng, G., & Monovich, L. (2016) Nat. Chem. Biol., 12, 896–898.
48) Wang, G., Yang, E., Smith, K.J., Zeng, Y., Ji, G., Connon, R., Fangue, N.A., & Cai, J.J. (2014) Front. Genet., 5, 312.
50) Lein, E.S., Hawrylycz, M.J., Ao, N., Ayres, M., Bensinger, A., Bernard, A., Boe, A.F., Boguski, M.S., Brockway, K.S., Byrnes, E.J., Chen, L., Chen, L., Chen, T.M., Chin, M.C., Chong, J., Crook, B.E., Czaplinska, A., Dang, C.N., Datta, S., Dee, N.R., Desaki, A.L., Desta, T., Diep, E., Dolbeare, T.A., Donelan, M.J., Dong, H.W., Dougherty, J.G., Duncan, B.J., Ebbert, A.J., Eichele, G., Estin, L.K., Faber, C., Facer, B.A., Fields, R., Fischer, S.R., Fliss, T.P., Frensley, C., Gates, S.N., Glattfelder, K.J., Halverson, K.R., Hart, M.R., Hohmann, J.G., Howell, M.P., Jeung, D.P., Johnson, R.A., Karr, P.T., Kawal, R., Kidney, J.M., Knapik, R.H., Kuan, C.L., Lake, J.H., Laramee, A.R., Larsen, K.D., Lau, C., Lemon, T.A., Liang, A.J., Liu, Y., Luong, L.T., Michaels, J., Morgan, J.J., Morgan, R.J., Mortrud, M.T., Mosqueda, N.F., Ng, L.L., Ng, R., Orta, G.J., Overly, C.C., Pak, T.H., Parry, S.E., Pathak, S.D., Pearson, O.C., Puchalski, R.B., Riley, Z.L., Rockett, H.R., Rowland, S.A., Royall, J.J., Ruiz, M.J., Sarno, N.R., Schaffnit, K., Shapovalova, N.V., Sivisay, T., Slaughterbeck, C.R., Smith, S.C., Smith, K.A., Smith, B.I., Sodt, A.J., Stewart, N.N., Stumpf, K.R., Sunkin, S.M., Sutram, M., Tam, A., Teemer, C.D., Thaller, C., Thompson, C.L., Varnam, L.R., Visel, A., Whitlock, R.M., Wohnoutka, P.E., Wolkey, C.K., Wong, V.Y., Wood, M., Yaylaoglu, M.B., Young, R.C., Youngstrom, B.L., Yuan, X.F., Zhang, B., Zwingman, T.A., & Jones, A.R. (2007) Nature, 445, 168–176.