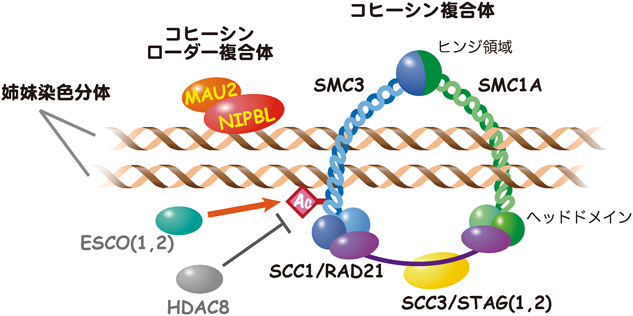
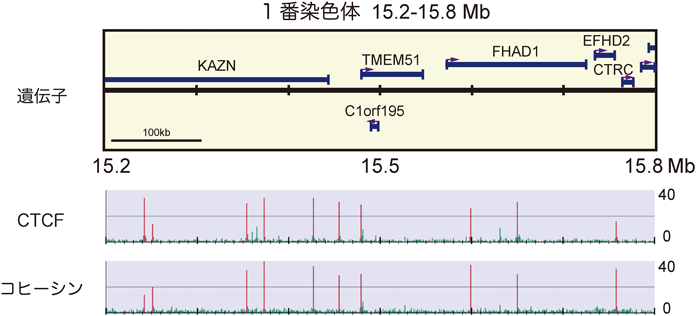
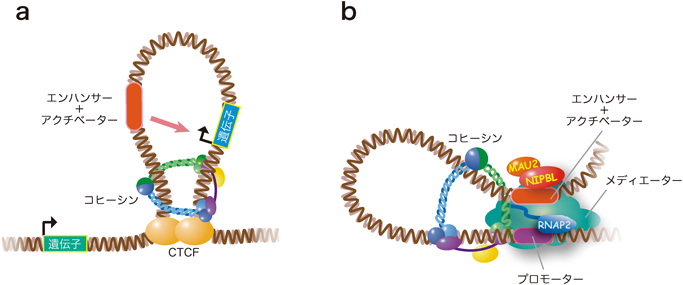
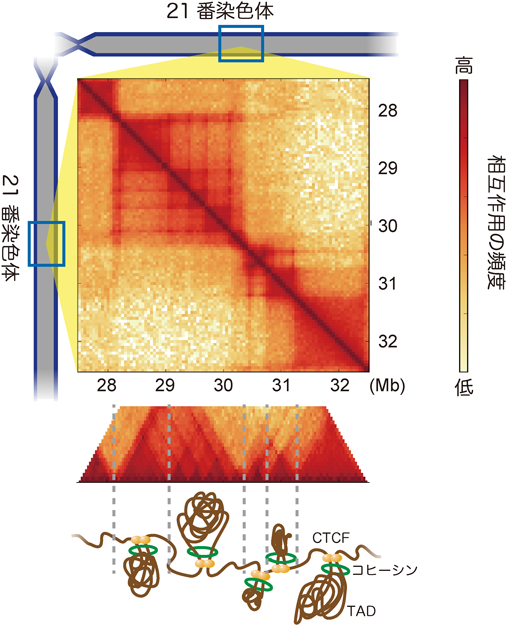
9) Deardorff, M.A., Clark, D.M., & Krantz, I.D. (1993–2013) in: GeneReviews™ (Pagon, R.A., Adam, M.P., Bird, T.D., Dolan, C.R., Fong, C.T., Stephens, K., eds.), University of Washington, Seattle.
19) Rollins, R.A., Morcillo, P., & Dorsett, D. (1999) Genetics, 152, 577–593.
41) Stunnenberg, H.G., Abrignani, S., Adams, D., de Almeida, M., Altucci, L., Amin, V., Amit, I., Antonarakis, S.E., Aparicio, S., Arima, T., Arrigoni, L., Arts, R., Asnafi, V., Esteller, M., Bae, J.-B., Bassler, K., Beck, S., Berkman, B., Bernstein, B.E., Bilenky, M., Bird, A., Bock, C., Boehm, B., Bourque, G., Breeze, C.E., Brors, B., Bujold, D., Burren, O., Bussemakers, M.J., Butterworth, A., Campo, E., Carrillo-de-Santa-Pau, E., Chadwick, L., Chan, K.M., Chen, W., Cheung, T.H., Chiapperino, L., Choi, N.H., Chung, H.-R., Clarke, L., Connors, J.M., Cronet, P., Danesh, J., Dermitzakis, M., Drewes, G., Durek, P., Dyke, S., Dylag, T., Eaves, C.J., Ebert, P., Eils, R., Eils, J., Ennis, C.A., Enver, T., Feingold, E.A., Felder, B., Ferguson-Smith, A., Fitzgibbon, J., Flicek, P., Foo, R.S.-Y., Fraser, P., Frontini, M., Furlong, E., Gakkhar, S., Gasparoni, N., Gasparoni, G., Geschwind, D.H., Glažar, P., Graf, T., Grosveld, F., Guan, X.-Y., Guigo, R., Gut, I.G., Hamann, A., Han, B.-G., Harris, R.A., Heath, S., Helin, K., Hengstler, J.G., Heravi-Moussavi, A., Herrup, K., Hill, S., Hilton, J.A., Hitz, B.C., Horsthemke, B., Hu, M., Hwang, J.-Y., Ip, N.Y., Ito, T., Javierre, B.-M., Jenko, S., Jenuwein, T., Joly, Y., Jones, S.J.M., Kanai, Y., Kang, H.G., Karsan, A., Kiemer, A.K., Kim, S.C., Kim, B.-J., Kim, H.-H., Kimura, H., Kinkley, S., Klironomos, F., Koh, I.-U., Kostadima, M., Kressler, C., Kreuzhuber, R., Kundaje, A., Küppers, R., Larabell, C., Lasko, P., Lathrop, M., Lee, D.H.S., Lee, S., Lehrach, H., Leitão, E., Lengauer, T., Lernmark, Å., Leslie, R.D., Leung, G.K.K., Leung, D., Loeffler, M., Ma, Y., Mai, A., Manke, T., Marcotte, E.R., Marra, M.A., Martens, J.H.A., Martin-Subero, J.I., Maschke, K., Merten, C., Milosavljevic, A., Minucci, S., Mitsuyama, T., Moore, R.A., Müller, F., Mungall, A.J., Netea, M.G., Nordström, K., Norstedt, I., Okae, H., Onuchic, V., Ouellette, F., Ouwehand, W., Pagani, M., Pancaldi, V., Pap, T., Pastinen, T., Patel, R., Paul, D.S., Pazin, M.J., Pelicci, P.G., Phillips, A.G., Polansky, J., Porse, B., Pospisilik, J.A., Prabhakar, S., Procaccini, D.C., Radbruch, A., Rajewsky, N., Rakyan, V., Reik, W., Ren, B., Richardson, D., Richter, A., Rico, D., Roberts, D.J., Rosenstiel, P., Rothstein, M., Salhab, A., Sasaki, H., Satterlee, J.S., Sauer, S., Schacht, C., Schmidt, F., Schmitz, G., Schreiber, S., Schröder, C., Schübeler, D., Schultze, J.L., Schulyer, R.P., Schulz, M., Seifert, M., Shirahige, K., Siebert, R., Sierocinski, T., Siminoff, L., Sinha, A., Soranzo, N., Spicuglia, S., Spivakov, M., Steidl, C., Strattan, J.S., Stratton, M., Südbeck, P., Sun, H., Suzuki, N., Suzuki, Y., Tanay, A., Torrents, D., Tyson, F.L., Ulas, T., Ullrich, S., Ushijima, T., Valencia, A., Vellenga, E., Vingron, M., Wallace, C., Wallner, S., Walter, J., Wang, H., Weber, S., Weiler, N., Weller, A., Weng, A., Wilder, S., Wiseman, S.M., Wu, A.R., Wu, Z., Xiong, J., Yamashita, Y., Yang, X., Yap, D.Y., Yip, K.Y., Yip, S., Yoo, J.-I., Zerbino, D., Zipprich, G., & Hirst, M. (2016) Cell, 167, 1145–1149.
42) Andersson, R., Gebhard, C., Miguel-Escalada, I., Hoof, I., Bornholdt, J., Boyd, M., Chen, Y., Zhao, X., Schmidl, C., Suzuki, T., Ntini, E., Arner, E., Valen, E., Li, K., Schwarzfischer, L., Glatz, D., Raithel, J., Lilje, B., Rapin, N., Bagger, F.O., Jørgensen, M., Andersen, P.R., Bertin, N., Rackham, O., Burroughs, A.M., Baillie, J.K., Ishizu, Y., Shimizu, Y., Furuhata, E., Maeda, S., Negishi, Y., Mungall, C.J., Meehan, T.F., Lassmann, T., Itoh, M., Kawaji, H., Kondo, N., Kawai, J., Lennartsson, A., Daub, C.O., Heutink, P., Hume, D.A., Jensen, T.H., Suzuki, H., Hayashizaki, Y., Müller, F., Forrest, A.R., Carninci, P., Rehli, M., & Sandelin, A. (2014) Nature, 507, 455–461.