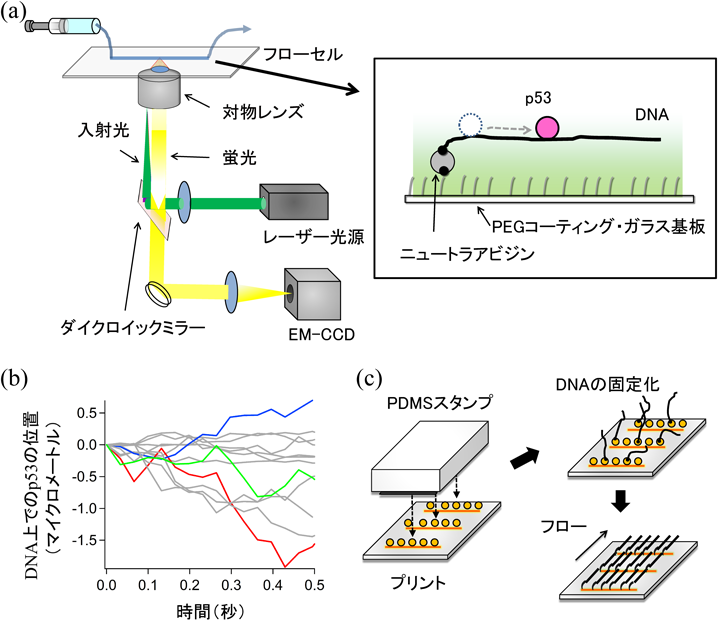
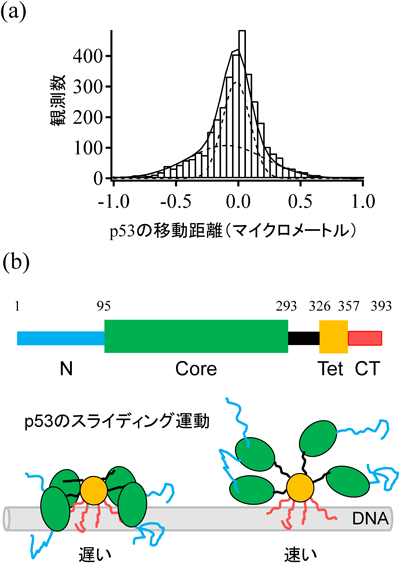
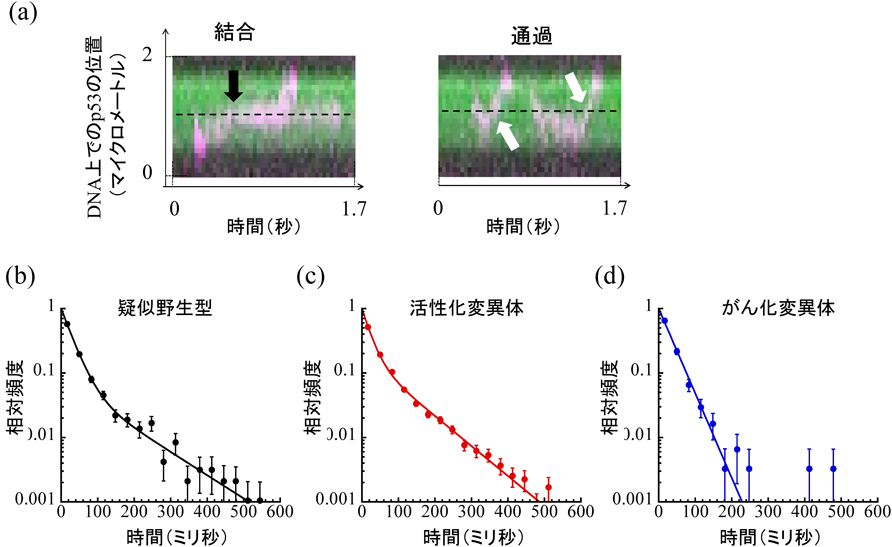
生物物理学的アプローチによるがん抑制タンパク質p53のDNA認識・結合機構の解明DNA-binding regulation of tumor suppressor p53 revealed by biophysical approach
1 東北大学多元物質科学研究所Institute for Multidisciplinary Research for Advanced Materials, Tohoku University ◇ 〒980–8577 宮城県仙台市青葉区片平2–1–1 ◇ Katahira 2–1–1, Aoba-ku, Sendai, Miyagi 980–8577, Japan
2 東北大学大学院理学研究科化学専攻Department of Chemistry, Graduate School of Science, Tohoku University ◇ 〒980–8577 宮城県仙台市青葉区片平2–1–1 ◇ Katahira 2–1–1, Aoba-ku, Sendai, Miyagi 980–8577, Japan