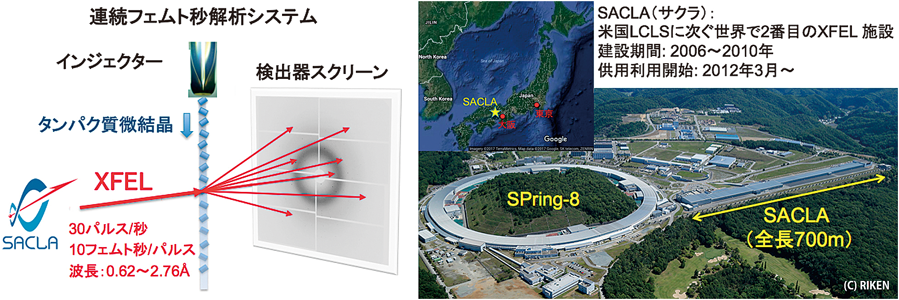
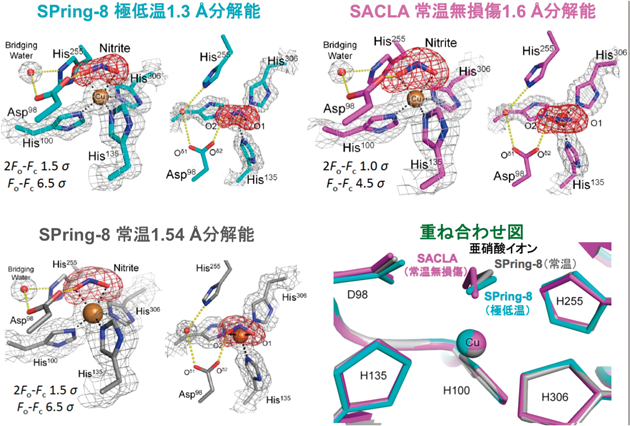
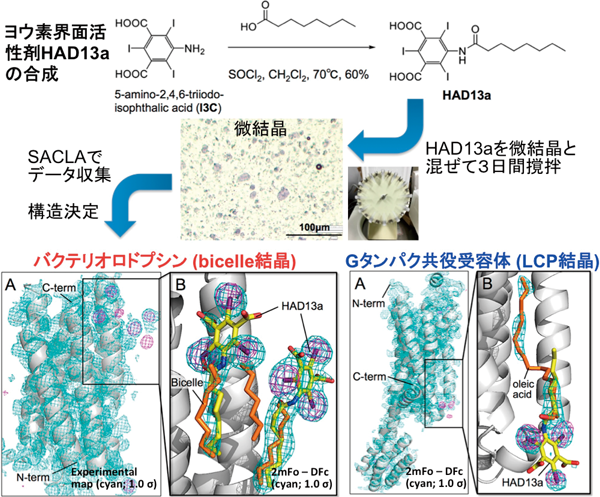
1) Fukuda, Y., Tse, K.M., Nakane, T., Nakatsu, T., Suzuki, M., Sugahara, M., Inoue, S., Masuda, T., Yumoto, F., Matsugaki, N., Nango, E., Tono, K., Joti, Y., Kameshima, T., Song, C., Hatsui, T., Yabashi, M., Nureki, O., Murphy, M.E., Inoue, T., Iwata, S., & Mizohata, E. (2016) Proc. Natl. Acad. Sci. USA, 113, 2928–2933.
2) Fukuda, Y., Tse, K.M., Suzuki, M., Diederichs, K., Hirata, K., Nakane, T., Sugahara, M., Nango, E., Tono, K., Joti, Y., Kameshima, T., Song, C., Hatsui, T., Yabashi, M., Nureki, O., Matsumura, H., Inoue, T., Iwata, S., & Mizohata, E. (2016) J. Biochem., 159, 527–538.
3) Barends, T.R., Foucar, L., Botha, S., Doak, R.B., Shoeman, R.L., Nass, K., Koglin, J.E., Williams, G.J., Boutet, S., Messerschmidt, M., & Schlichting, I. (2014) Nature, 505, 244–247.
4) Nakane, T., Song, C., Suzuki, M., Nango, E., Kobayashi, J., Masuda, T., Inoue, S., Mizohata, E., Nakatsu, T., Tanaka, T., Tanaka, R., Shimamura, T., Tono, K., Joti, Y., Kameshima, T., Hatsui, T., Yabashi, M., Nureki, O., Iwata, S., & Sugahara, M. (2015) Acta Crystallogr. D Biol. Crystallogr., 71, 2519–2525.
5) Nass, K., Meinhart, A., Barends, T.R., Foucar, L., Gorel, A., Aquila, A., Botha, S., Doak, R.B., Koglin, J., Liang, M., Shoeman, R.L., Williams, G., Boutet, S., & Schlichting, I. (2016) IUCrJ, 3, 180–191.
6) Batyuk, A., Galli, L., Ishchenko, A., Han, G.W., Gati, C., Popov, P.A., Lee, M.Y., Stauch, B., White, T.A., Barty, A., Aquila, A., Hunter, M.S., Liang, M., Boutet, S., Pu, M., Liu, Z.J., Nelson, G., James, D., Li, C., Zhao, Y., Spence, J.C., Liu, W., Fromme, P., Katritch, V., Weierstall, U., Stevens, R.C., & Cherezov, V. (2016) Sci. Adv., 2, e1600292.
7) Nakane, T., Hanashima, S., Suzuki, M., Saiki, H., Hayashi, T., Kakinouchi, K., Sugiyama, S., Kawatake, S., Matsuoka, S., Matsumori, N., Nango, E., Kobayashi, J., Shimamura, T., Kimura, K., Mori, C., Kunishima, N., Sugahara, M., Takakyu, Y., Inoue, S., Masuda, T., Hosaka, T., Tono, K., Joti, Y., Kameshima, T., Hatsui, T., Yabashi, M., Inoue, T., Nureki, O., Iwata, S., Murata, M., & Mizohata, E. (2016) Proc. Natl. Acad. Sci. USA, 113, 13039–13044.
8) Yamashita, K., Pan, D., Okuda, T., Sugahara, M., Kodan, A., Yamaguchi, T., Murai, T., Gomi, K., Kajiyama, N., Mizohata, E., Suzuki, M., Nango, E., Tono, K., Joti, Y., Kameshima, T., Park, J., Song, C., Hatsui, T., Yabashi, M., Iwata, S., Kato, H., Ago, H., Yamamoto, M., & Nakatsu, T. (2015) Sci. Rep., 5, 14017.
9) Sugahara, M., Nakane, T., Masuda, T., Suzuki, M., Inoue, S., Song, C., Tanaka, R., Nakatsu, T., Mizohata, E., Yumoto, F., Tono, K., Joti, Y., Kameshima, T., Hatsui, T., Yabashi, M., Nureki, O., Numata, K., Nango, E., & Iwata, E. (2017) Sci. Rep., in press.
10) Ginn, H.M., Brewster, A.S., Hattne, J., Evans, G., Wagner, A., Grimes, J.M., Sauter, N.K., Sutton, G., & Stuart, D.I. (2015) Acta Crystallogr. D Biol. Crystallogr., 71, 1400–1410.
11) Uervirojnangkoorn, M., Zeldin, O.B., Lyubimov, A.Y., Hattne, J., Brewster, A.S., Sauter, N.K., Brunger, A.T., & Weis, W.I. (2015) eLife, 4, e05421.
12) Colletier, J.P., Sawaya, M.R., Gingery, M., Rodriguez, J.A., Cascio, D., Brewster, A.S., Michels-Clark, T., Hice, R.H., Coquelle, N., Boutet, S., Williams, G.J., Messerschmidt, M., DePonte, D.P., Sierra, R.G., Laksmono, H., Koglin, J.E., Hunter, M.S., Park, H.W., Uervirojnangkoorn, M., Bideshi, D.K., Brunger, A.T., Federici, B.A., Sauter, N.K., & Eisenberg, D.S. (2016) Nature, 539, 43–47.
13) Nango, E., Royant, A., Kubo, M., Nakane, T., Wickstrand, C., Kimura, T., Tanaka, T., Tono, K., Song, C., Tanaka, R., Arima, T., Yamashita, A., Kobayashi, J., Hosaka, T., Mizohata, E., Nogly, P., Sugahara, M., Nam, D., Nomura, T., Shimamura, T., Im, D., Fujiwara, T., Yamanaka, Y., Jeon, B., Nishizawa, T., Oda, K., Fukuda, M., Andersson, R., Båth, P., Dods, R., Davidsson, J., Matsuoka, S., Kawatake, S., Murata, M., Nureki, O., Owada, S., Kameshima, T., Hatsui, T., Joti, Y., Schertler, G., Yabashi, M., Bondar, A.N., Standfuss, J., Neutze, R., & Iwata, S. (2016) Science, 354, 1552–1557.
14) Suga, M., Akita, F., Sugahara, M., Kubo, M., Nakajima, Y., Nakane, T., Yamashita, K., Umena, Y., Nakabayashi, M., Yamane, T., Nakano, T., Suzuki, M., Masuda, T., Inoue, S., Kimura, T., Nomura, T., Yonekura, S., Yu, L.J., Sakamoto, T., Motomura, T., Chen, J.H., Kato, Y., Noguchi, T., Tono, K., Joti, Y., Kameshima, T., Hatsui, T., Nango, E., Tanaka, R., Naitow, H., Matsuura, Y., Yamashita, A., Yamamoto, M., Nureki, O., Yabashi, M., Ishikawa, T., Iwata, S., & Shen, J.R. (2017) Nature, 543, 131–135.