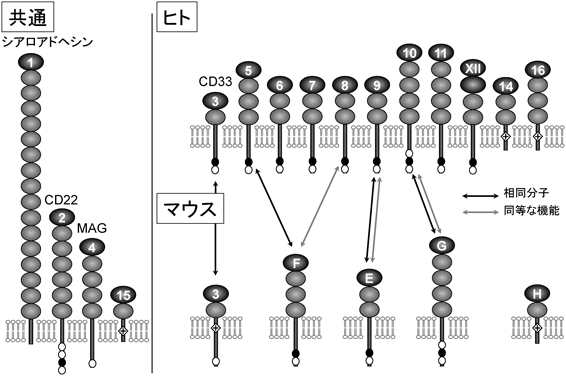
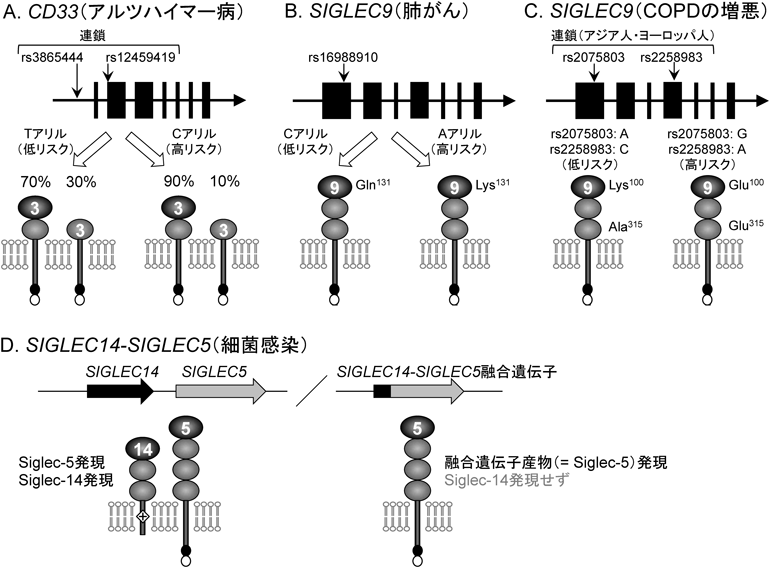
4) Bull, C., den Brok, M.H., & Adema, G.J. (2014) Biochim. Biophys. Acta, 1846, 238–246.
6) Naj, A., Jun, G., Beecham, G., Wang, L., Vardarajan, B., Buros, J., Gallins, P.J., Buxbaum, J.D., Jarvik, G.P., Crane, P.K., Larson, E.B., Bird, T.D., Boeve, B.F., Graff-Radford, N.R., De Jager, P.L., Evans, D., Schneider, J.A., Carrasquillo, M.M., Ertekin-Taner, N., Younkin, S.G., Cruchaga, C., Kauwe, J.S., Nowotny, P., Kramer, P., Hardy, J., Huentelman, M.J., Myers, A.J., Barmada, M.M., Demirci, F.Y., Baldwin, C.T., Green, R.C., Rogaeva, E., St George-Hyslop, P., Arnold, S.E., Barber, R., Beach, T., Bigio, E.H., Bowen, J.D., Boxer, A., Burke, J.R., Cairns, N.J., Carlson, C.S., Carney, R.M., Carroll, S.L., Chui, H.C., Clark, D.G., Corneveaux, J., Cotman, C.W., Cummings, J.L., DeCarli, C., DeKosky, S.T., Diaz-Arrastia, R., Dick, M., Dickson, D.W., Ellis, W.G., Faber, K.M., Fallon, K.B., Farlow, M.R., Ferris, S., Frosch, M.P., Galasko, D.R., Ganguli, M., Gearing, M., Geschwind, D.H., Ghetti, B., Gilbert, J.R., Gilman, S., Giordani, B., Glass, J.D., Growdon, J.H., Hamilton, R.L., Harrell, L.E., Head, E., Honig, L.S., Hulette, C.M., Hyman, B.T., Jicha, G.A., Jin, L.W., Johnson, N., Karlawish, J., Karydas, A., Kaye, J.A., Kim, R., Koo, E.H., Kowall, N.W., Lah, J.J., Levey, A.I., Lieberman, A.P., Lopez, O.L., Mack, W.J., Marson, D.C., Martiniuk, F., Mash, D.C., Masliah, E., McCormick, W.C., McCurry, S.M., McDavid, A.N., McKee, A.C., Mesulam, M., Miller, B.L., Miller, C.A., Miller, J.W., Parisi, J.E., Perl, D.P., Peskind, E., Petersen, R.C., Poon, W.W., Quinn, J.F., Rajbhandary, R.A., Raskind, M., Reisberg, B., Ringman, J.M., Roberson, E.D., Rosenberg, R.N., Sano, M., Schneider, L.S., Seeley, W., Shelanski, M.L., Slifer, M.A., Smith, C.D., Sonnen, J.A., Spina, S., Stern, R.A., Tanzi, R.E., Trojanowski, J.Q., Troncoso, J.C., Van Deerlin, V.M., Vinters, H.V., Vonsattel, J.P., Weintraub, S., Welsh-Bohmer, K.A., Williamson, J., Woltjer, R.L., Cantwell, L.B., Dombroski, B.A., Beekly, D., Lunetta, K.L., Martin, E.R., Kamboh, M.I., Saykin, A.J., Reiman, E.M., Bennett, D.A., Morris, J.C., Montine, T.J., Goate, A.M., Blacker, D., Tsuang, D.W., Hakonarson, H., Kukull, W.A., Foroud, T.M., Haines, J.L., Mayeux, R., Pericak-Vance, M.A., Farrer, L.A., & Schellenberg, G.D. (2011) Nat. Genet., 43, 436–441.
7) Hollingworth, P., Harold, D., Sims, R., Gerrish, A., Lambert, J., Carrasquillo, M.M., Abraham, R., Hamshere, M.L., Pahwa, J.S., Moskvina, V., Dowzell, K., Jones, N., Stretton, A., Thomas, C., Richards, A., Ivanov, D., Widdowson, C., Chapman, J., Lovestone, S., Powell, J., Proitsi, P., Lupton, M.K., Brayne, C., Rubinsztein, D.C., Gill, M., Lawlor, B., Lynch, A., Brown, K.S., Passmore, P.A., Craig, D., McGuinness, B., Todd, S., Holmes, C., Mann, D., Smith, A.D., Beaumont, H., Warden, D., Wilcock, G., Love, S., Kehoe, P.G., Hooper, N.M., Vardy, E.R., Hardy, J., Mead, S., Fox, N.C., Rossor, M., Collinge, J., Maier, W., Jessen, F., Rüther, E., Schürmann, B., Heun, R., Kölsch, H., van den Bussche, H., Heuser, I., Kornhuber, J., Wiltfang, J., Dichgans, M., Frölich, L., Hampel, H., Gallacher, J., Hüll, M., Rujescu, D., Giegling, I., Goate, A.M., Kauwe, J.S., Cruchaga, C., Nowotny, P., Morris, J.C., Mayo, K., Sleegers, K., Bettens, K., Engelborghs, S., De Deyn, P.P., Van Broeckhoven, C., Livingston, G., Bass, N.J., Gurling, H., McQuillin, A., Gwilliam, R., Deloukas, P., Al-Chalabi, A., Shaw, C.E., Tsolaki, M., Singleton, A.B., Guerreiro, R., Mühleisen, T.W., Nöthen, M.M., Moebus, S., Jöckel, K.H., Klopp, N., Wichmann, H.E., Pankratz, V.S., Sando, S.B., Aasly, J.O., Barcikowska, M., Wszolek, Z.K., Dickson, D.W., Graff-Radford, N.R., Petersen, R.C., van Duijn, C.M., Breteler, M.M., Ikram, M.A., DeStefano, A.L., Fitzpatrick, A.L., Lopez, O., Launer, L.J., Seshadri, S., Berr, C., Campion, D., Epelbaum, J., Dartigues, J.F., Tzourio, C., Alpérovitch, A., Lathrop, M., Feulner, T.M., Friedrich, P., Riehle, C., Krawczak, M., Schreiber, S., Mayhaus, M., Nicolhaus, S., Wagenpfeil, S., Steinberg, S., Stefansson, H., Stefansson, K., Snaedal, J., Björnsson, S., Jonsson, P.V., Chouraki, V., Genier-Boley, B., Hiltunen, M., Soininen, H., Combarros, O., Zelenika, D., Delepine, M., Bullido, M.J., Pasquier, F., Mateo, I., Frank-Garcia, A., Porcellini, E., Hanon, O., Coto, E., Alvarez, V., Bosco, P., Siciliano, G., Mancuso, M., Panza, F., Solfrizzi, V., Nacmias, B., Sorbi, S., Bossù, P., Piccardi, P., Arosio, B., Annoni, G., Seripa, D., Pilotto, A., Scarpini, E., Galimberti, D., Brice, A., Hannequin, D., Licastro, F., Jones, L., Holmans, P.A., Jonsson, T., Riemenschneider, M., Morgan, K., Younkin, S.G., Owen, M.J., O’Donovan, M., Amouyel, P., & Williams, J., Alzheimer’s Disease Neuroimaging Initiative, CHARGE consortium, EADI1 consortium (2011) Nat. Genet., 43, 429–435.
14) Munz, M., Willenborg, C., Richter, G.M., Jockel-Schneider, Y., Graetz, C., Staufenbiel, I., Wellmann, J., Berger, K., Krone, B., Hoffmann, P., Velde, N.V., Uitterlinden, A.G., Groot, L.C.P.G.M., Sawalha, A., Direskeneli, H., Saruhan-Direskeneli, G., Guzeldemir-Akcakanat, E., Keceli, G., Laudes, M., Noack, B., Teumer, A., Holtfreter, B., Kocher, T., Eickholz, P., Meyle, J., Doerfer, C., Bruckmann, C., Lieb, W., Franke, A., Schreiber, S., Nohutcu, R.M., Erdmann, J., Loos, B.G., Jepsen, S., Dommisch, H., & Schaefer, A.S. (2017) Hum. Mol. Genet., 26, 2577–2588.
17) Sun, C., Molineros, J.E., Looger, L.L., Zhou, X.J., Kim, K., Okada, Y., Ma, J., Qi, Y.Y., Kim-Howard, X., Motghare, P., Bhattarai, K., Adler, A., Bang, S.Y., Lee, H.S., Kim, T.H., Kang, Y.M., Suh, C.H., Chung, W.T., Park, Y.B., Choe, J.Y., Shim, S.C., Kochi, Y., Suzuki, A., Kubo, M., Sumida, T., Yamamoto, K., Lee, S.S., Kim, Y.J., Han, B.G., Dozmorov, M., Kaufman, K.M., Wren, J.D., Harley, J.B., Shen, N., Chua, K.H., Zhang, H., Bae, S.C., & Nath, S.K. (2016) Nat. Genet., 48, 323–330.