4) Bernstein, B.E., Mikkelsen, T.S., Xie, X., Kamal, M., Huebert, D.J., Cuff, J., Fry, B., Meissner, A., Wernig, M., Plath, K., Jaenisch, R., Wagschal, A., Feil, R., Schreiber, S.L., & Lander, E.S. (2006) Cell, 125, 315–326.
5) Mikkelsen, T.S., Xu, Z., Zhang, X., Wang, L., Gimble, J.M., Lander, E.S., & Rosen, E.D. (2010) Cell, 143, 156–169.
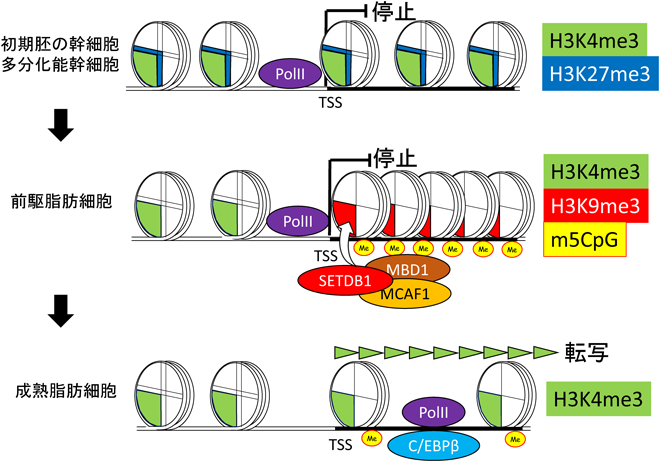
6) Matsumura, Y., Nakaki, R., Inagaki, T., Yoshida, A., Kano, Y., Kimura, H., Tanaka, T., Tsutsumi, S., Nakao, M., Doi, T., Fukami, K., Osborne, T.F., Kodama, T., Aburatani, H., & Sakai, J. (2015) Mol. Cell, 60, 584–596.
7) Yamane, K., Toumazou, C., Tsukada, Y., Erdjument-Bromage, H., Tempst, P., Wong, J., & Zhang, Y. (2006) Cell, 125, 483–495.
8) Okada, Y., Scott, G., Ray, M.K., Mishina, Y., & Zhang, Y. (2007) Nature, 450, 119–123.
9) Kuroki, S., Matoba, S., Akiyoshi, M., Matsumura, Y., Miyachi, H., Mise, N., Abe, K., Ogura, A., Wilhelm, D., Koopman, P., Nozaki, M., Kanai, Y., Shinkai, Y., & Tachibana, M. (2013) Science, 341, 1106–1109.
10) Inagaki, T., Tachibana, M., Magoori, K., Kudo, H., Tanaka, T., Okamura, M., Naito, M., Kodama, T., Shinkai, Y., & Sakai, J. (2009) Genes Cells, 14, 991–1001.
11) Tateishi, K., Okada, Y., Kallin, E.M., & Zhang, Y. (2009) Nature, 458, 757–761.
12) Abe, Y., Rozqie, R., Matsumura, Y., Kawamura, T., Nakaki, R., Tsurutani, Y., Tanimura-Inagaki, K., Shiono, A., Magoori, K., Nakamura, K., Ogi, S., Kajimura, S., Kimura, H., Tanaka, T., Fukami, K., Osborne, T.F., Kodama, T., Aburatani, H., Inagaki, T., & Sakai, J. (2015) Nat. Commun., 6, 7052.
13) Inagaki, T., Iwasaki, S., Matsumura, Y., Kawamura, T., Tanaka, T., Abe, Y., Yamasaki, A., Tsurutani, Y., Yoshida, A., Chikaoka, Y., Nakamura, K., Magoori, K., Nakaki, R., Osborne, T.F., Fukami, K., Aburatani, H., Kodama, T., & Sakai, J. (2015) J. Biol. Chem., 290, 4163–4177.
14) Cheng, M.B., Zhang, Y., Cao, C.Y., Zhang, W.L., Zhang, Y., & Shen, Y.F. (2014) PLoS Biol., 12, e1002026.
15) Sun, Z., Feng, D., Fang, B., Mullican, S.E., You, S.H., Lim, H.W., Everett, L.J., Nabel, C.S., Li, Y., Selvakumaran, V., Won, K.J., & Lazar, M.A. (2013) Mol. Cell, 52, 769–782.
16) Baba, A., Ohtake, F., Okuno, Y., Yokota, K., Okada, M., Imai, Y., Ni, M., Meyer, C.A., Igarashi, K., Kanno, J., Brown, M., & Kato, S. (2011) Nat. Cell Biol., 13, 668–675.
17) Mimura, I., Nangaku, M., Kanki, Y., Tsutsumi, S., Inoue, T., Kohro, T., Yamamoto, S., Fujita, T., Shimamura, T., Suehiro, J., Taguchi, A., Kobayashi, M., Tanimura, K., Inagaki, T., Tanaka, T., Hamakubo, T., Sakai, J., Aburatani, H., Kodama, T., & Wada, Y. (2012) Mol. Cell. Biol., 32, 3018–3032.
18) Claussnitzer, M., Dankel, S.N., Kim, K.H., Quon, G., Meuleman, W., Haugen, C., Glunk, V., Sousa, I.S., Beaudry, J.L., Puviindran, V., Abdennur, N.A., Liu, J., Svensson, P.A., Hsu, Y.H., Drucker, D.J., Mellgren, G., Hui, C.C., Hauner, H., & Kellis, M. (2015) N. Engl. J. Med., 373, 895–907.
19) Smemo, S., Tena, J.J., Kim, K.H., Gamazon, E.R., Sakabe, N.J., Gómez-Marín, C., Aneas, I., Credidio, F.L., Sobreira, D.R., Wasserman, N.F., Lee, J.H., Puviindran, V., Tam, D., Shen, M., Son, J.E., Vakili, N.A., Sung, H.K., Naranjo, S., Acemel, R.D., Manzanares, M., Nagy, A., Cox, N.J., Hui, C.C., Gomez-Skarmeta, J.L., & Nóbrega, M.A. (2014) Nature, 507, 371–375.
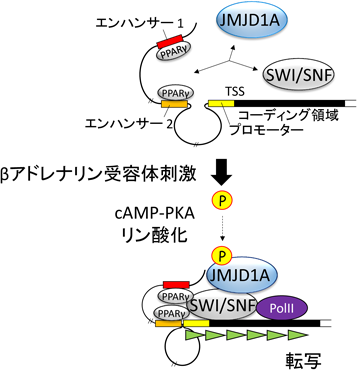
20) Roqueta-Rivera, M., Esquejo, R.M., Phelan, P.E., Sandor, K., Daniel, B., Foufelle, F., Ding, J., Li, X., Khorasanizadeh, S., & Osborne, T.F. (2016) Cell Metab., 24, 474–484.