多数の神経細胞を生み出す神経前駆領域内において,神経幹細胞は多様な神経細胞を多数生み出し,複雑かつ多様な神経回路を形成する.このような神経多様性を生み出す過程において空間軸および時間軸に沿った制御機構が重要な役割を果たす.空間的なパターン形成は主に分泌性タンパク質によるモルフォゲン濃度勾配によって支配される1).一方,時間的なパターン形成はTemporal Factorと呼ばれる一連の転写因子が時間とともに発現を変化させ,これらの因子の神経幹細胞における発現変化が生み出される神経細胞の性質を決定する.また,転写因子以外にもエピジェネティックなメカニズムの関与も知られている2, 3).これら空間的および時間的パターン形成機構はさまざまな動物のさまざまな脳領域において研究されてきた.たとえば神経前駆細胞の空間パターン形成機構はトリやマウスの脊髄,さらにマウスの視床を用いて精力的に研究された1, 4).神経前駆細胞の時間的パターン形成はカエルやマウスの網膜,またマウスの大脳皮質を用いて研究された5–8).これらの時空間パターン形成メカニズムは脊椎動物に特有なわけではなく,ショウジョウバエなどの無脊椎動物においてもみられる.神経前駆細胞の空間パターン形成は胚期の脳や腹部神経節および視覚中枢において明らかにされている9–13).時間的パターン形成機構については胚期における腹部神経節,蛹期における脳において示された3, 14–18).哺乳類とショウジョウバエの脳の間にはさまざまな形態学的また機能的な類似性が指摘されているが,空間的・時間的パターン形成についても共通点が多くみられるのである19, 20).最後に,空間的パターン形成と時間的パターン形成の組み合わせによって一つの神経前駆領域から生み出される神経細胞の多様性がさらに拡張されることも脊椎動物および無脊椎動物を用いた研究から明らかにされている2, 3, 13).
上記のように個々の神経前駆領域から多様な神経細胞が生み出されるメカニズムについてはよく知られているが,我々はここで“inter-progenitor pool wiring”と名づけた神経発生戦略に注目したい.一つの神経前駆領域から生まれた神経細胞の一群は細胞移動によって離れた脳領域に到達し,別の神経前駆領域に由来する神経細胞群に統合され,はるかに複雑な神経回路を形成する(図1)7, 8, 10–12, 18).空間的パターン形成,時間的パターン形成,そして神経細胞移動の組み合わせによって神経回路の多様性が飛躍的に増大し,脳の情報処理能力の向上にも大きく寄与すると考えられる.以下に議論するように,inter-progenitor pool wiringは哺乳類の大脳皮質とショウジョウバエ視覚中枢において見いだされている.本稿では哺乳類とショウジョウバエの脳においてinter-progenitor pool wiringに関わると考えられる分子機構および進化的な共通性について議論する.
2. 大脳皮質における神経前駆領域の空間的パターン形成
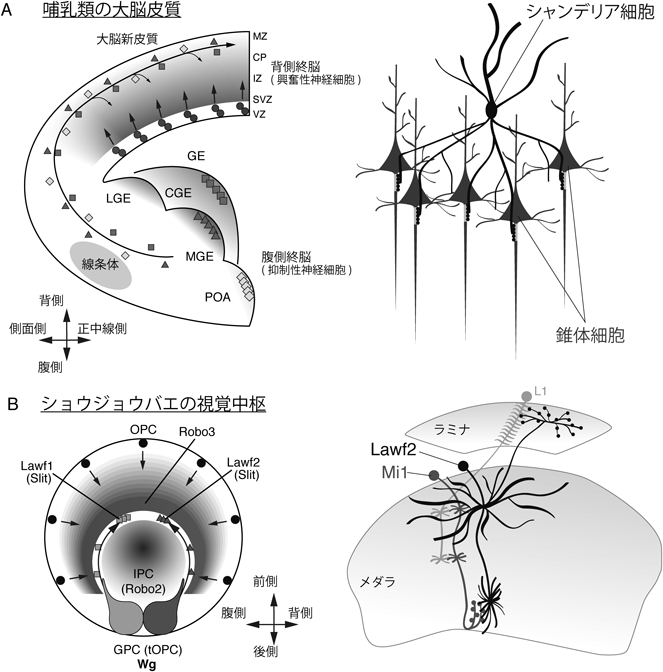
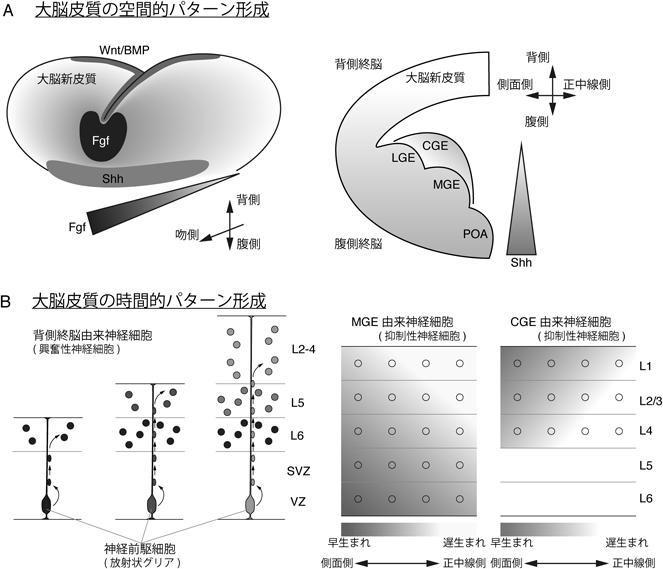
発生過程において哺乳類の脳は空間的に区画化され,それぞれの領域は異なる神経前駆領域を形成する.マウスの大脳皮質において,興奮性神経細胞は背側終脳の脳室帯(VZ)および脳室下帯(SVZ)において産生され,放射方向に移動することで大脳新皮質の特徴的な構造である6層構造(L1~L6)を形成する(図1A,図2B)21).早生まれの神経細胞は深層側に,遅生まれの神経細胞は早生まれの細胞を追い越し表層側に到達することで,いわゆる“inside-out”の配置パターンを形成する.抑制性神経細胞については腹側終脳において産生される(図1A,図2A)7, 8).この領域はさらに外側基底核原基(LGE),内側基底核原基(MGE),尾側基底核原基(CGE),そして視索前野(POA)の四つの領域に細分化される(POAは本来視床下部の一部であるが,簡単のため基底核原基と区別せずに議論する).抑制性神経細胞はMGE, CGE, POAにおいて産生された後,接線方向の細胞移動によって背側終脳の大脳皮質まで到達し,大脳皮質の神経回路を形成する7, 8).このように,背側終脳と腹側終脳はまったく異なる神経細胞群を生み出すが,それらが協調して神経回路を形成するのである(図2A).
これらの神経前駆領域はどのようにして独自の性質を獲得するのだろうか? 分泌性タンパク質である線維芽増殖因子(FGF)やSonic Hedgehog(Shh)のモルフォゲンとしての働きによって終脳の大まかな空間的パターン形成が制御される22).Fgf8は背側終脳において大脳新皮質の前後軸方向のパターン形成を制御する(図2A)23, 24).さらに,Fgf17, Wnt,骨形成タンパク質(BMP)そして上皮増殖因子(EGF)ファミリーの分泌性タンパク質も背側終脳において空間的位置情報を与える(図2A).背側終脳のVZはこれら分泌性因子の制御下において発現するさまざまな転写因子の働きによってさらに細かい領域に細分化される25, 26).一方,腹側終脳においてはShhがMGEおよびCGEの運命決定において重要な役割を果たす(図2A)22, 27, 28).Shhの発現は神経産生の間も持続するが,神経前駆領域の運命決定も制御する29).さらに,転写因子Nkx2.1の発現制御を介してMGEにおける神経細胞の運命決定を制御する30–32).Nkx2.1の変異マウスではMGEはLGEへと運命転換し,大脳皮質へ抑制性介在神経を供給しなくなる33, 34).MGEは主にパルブアルブミン(PV)およびソマトスタチン(SST)陽性神経細胞を生み,CGEは血管作動性腸管ペプチド(VIP)およびReelin陽性神経を生み出す.このように,腹側終脳の空間パターンは実際に神経前駆領域ごとの異なる神経タイプ産生に寄与しているのである.また,MGE, CEGおよびPOAはさらに細かい領域に細分化されることがわかっている35, 36).このように,大脳皮質の神経細胞は背側終脳および腹側終脳に由来し,それぞれの神経前駆領域の空間パターン形成は主にFGF, Shh, Wnt, BMP, EGFといった分泌性因子によって制御されるのである22, 25, 26).
哺乳類大脳皮質において時間的パターンに従って神経タイプを決定する分子機構は背側終脳を用いて精力的に研究された3, 25).背側終脳のVZやSVZに位置する神経前駆細胞から生み出された神経細胞は神経前駆細胞である放射状グリアに由来する放射状突起に沿って放射方向に移動する(図2B,図3B).遅生まれの神経細胞は早生まれの神経細胞を追い抜いて脳の表層側に移動するため,最終的に早生まれの神経細胞ほど脳の深層側に位置し,inside-outの層構造を形成する.この過程に関わる多くの遺伝子のうち,Fezf2とIkarosは大脳新皮質における時間的パターン形成を制御する転写因子として知られる.どちらもSVZにおける若い神経前駆細胞において一過的に発現し,早生まれの5~6層に位置する神経細胞の運命を決定する3, 37, 38).Ikarosはショウジョウバエの胚期中枢神経系において時間的パターン形成を制御する転写因子Hunchbackのホモログであることは興味深い.両者はどちらも早い時期に生まれる神経細胞タイプを決定するのである37).さらに,Brn1/2およびOtx1は背側終脳のVZにおいて時間的に特異的な発現パターンを示す39).また,ポリコーム因子の一つであるRing1Bが5層に見られる神経タイプの産生時期の終結を制御することが示されており,エピジェネティックな制御が背側終脳における時間的パターン形成に関与することが示唆される40).
背側終脳と同様に,腹側終脳においても時間的パターン形成機構が働く(図2B)41, 42).MGEおよびCGEに由来する介在神経の産生時期を調べた解析結果から,これらの神経細胞は背側終脳において側方-正中方向の勾配に従った分布を示す,つまり早生まれの神経細胞ほど脳の側部に早く到達することがわかった.さらに,産生時期に従ってMGE由来の神経細胞は新皮質の深層側から表層側へ,CGE由来の神経細胞は表層側から深層側へ向かった勾配に沿って分布する42).しかし,CGE由来神経細胞は産生時期とは無関係に表層側に分布するとも報告されている41, 43, 44).
神経前駆細胞の時間的パターン形成は腹側終脳に由来する介在神経のサブタイプ決定にも関与することがわかっている.MGEおよびCGEにおいて,時間とともに生理学的に異なる介在神経のサブタイプが産生される43, 44).シャンデリア細胞は軸索投射やシナプス結合のパターンに関してきわめてステレオタイプな介在神経サブタイプである45).一つのシャンデリア細胞は複数の興奮性錐体細胞の軸索起始部(AIS)に投射する(図1A).AISは活動電位が発生する部位なので,シャンデリア細胞は一群の錐体細胞の神経活動を統合的に支配していると考えられる.また,シャンデリア細胞は発生後期においてMGEの神経前駆細胞から産生されることが示された46).このように,背側終脳および腹側終脳の神経前駆細胞は空間的そして時間的なパターンに従って制御されていることがわかる.
4. 大脳皮質における興奮性神経細胞と抑制性神経細胞によるinter-progenitor pool wiring
このように,興奮性と抑制性という異なるタイプの神経細胞がそれぞれ背側および腹側終脳の独立した神経前駆領域から生み出される.さらに,抑制性神経細胞は背側終脳に向かって移動し,背側終脳に由来する興奮性神経細胞とともに神経回路を形成する(図1A).このように,大脳皮質はinter-progenitor pool wiringの代表的な例である.個々の神経前駆領域から多様な神経細胞が生み出された後,以下の三つのステップによってinter-progenitor pool wiringが実現されると考えられる.1)腹側終脳から抑制性介在神経が離脱し,2)背側終脳まで移動し,3)移動完了後に大脳新皮質の興奮性神経細胞とシナプス結合する42).
このうち最初の二つのステップについては比較的よく研究されており,さまざまなガイダンス分子に制御されることが明らかにされている.たとえばEphrin-A5とEphA4が基底核原基由来の介在神経と基底核原基との間の反発性の相互作用を制御し,これによって抑制性神経細胞が基底核原基の神経前駆領域から脱出する47).他にもSema3A/3FとNeuropilin1/2がMGE由来介在神経と線条体間の反発性相互作用を制御し,これによって介在神経が線条体に向かわずに大脳新皮質に向かって移動する経路を選択することになる(図1A)48).一方,Neuregulin1とその受容体ErbB4による抑制性介在神経の誘引が背側終脳の大脳新皮質への移動を制御する49, 50).さらに,環境中のGABA,グリシン,グルタミン酸,ドーパミンなどの神経伝達物質が介在神経の大脳新皮質への移動を制御する42).
基底核原基由来の介在神経は辺縁帯(MZ),中間帯(IZ),SVZといった背側終脳内の特定の経路を通って移動するが8),その経路は以下のようにして制御される.大脳新皮質のMZにおいて産生されるNetrin1がα4β1インテグリンと結合することによって介在神経がMZを通って移動する.Netrin1とα4β1インテグリンの二重変異体では抑制性介在神経が異所的にVZを通過するようになる51).カハールレチウス細胞(CR)も介在神経のMZ内の移動を制御しているかもしれない42).細胞移動の最後のステップとして,基底核原基由来の神経細胞は移動方向を接線方向から放射方向に切り換え,移動経路であるMZを脱出し,放射方向の移動によって皮質板(CP)へと進入する.最終的にこれらの介在神経は産生順に従って大脳新皮質の各層に分布するようになる.MGEおよびCGEに由来する神経細胞はそれぞれinside-outおよびoutside-inの分布パターンを示す(図2B)42).接線方向から放射方向への切換えタイミングはCKCL12とその受容体CXCR4/7によって制御されている.CXCR4/7の機能欠損によって,基底核原基由来の神経細胞は通常よりも早く移動経路を脱出し,未成熟なCPに進入する52–58).
Inter-progenitor pool wiringの最後のステップは背側終脳と腹側終脳に由来するそれぞれの神経細胞が互いを認識する過程である.この相互作用の詳細はほとんどわかっていないが,MGEに由来する神経細胞の分布が大脳新皮質の錐体細胞によって直接的な影響を受けることがわかっている.Reelin変異マウスにおいて興奮性錐体細胞の分布が乱れると,抑制性介在神経細胞の層状の分布も異常を来す.この表現型は抑制性介在神経細胞内のReelinシグナルの異常によって細胞自律的に生じているわけではない点は重要である59, 60).Fezf2は5~6層に位置する神経細胞の運命を決定するが,Fezf2変異マウスにおいて錐体細胞のサブタイプが脳梁投射神経に転換する.このとき,大脳新皮質における抑制性介在神経の分布が異常を示す61).さらに,脳梁投射神経の細胞タイプを直接的に別のサブタイプである皮質遠心投射神経に転換させることで,抑制性介在神経が大脳新皮質の異所的な部位に生じる62).このように,背側終脳由来の興奮性神経細胞が実際に腹側終脳由来の抑制性介在神経の分布を制御しているのである.
興奮性錐体細胞と顕著に相互作用する抑制性介在神経の例としてシャンデリア細胞が挙げられる.シャンデリア細胞は後期MGEにおいて産生され,出生後に大脳新皮質まで移動し,最終的に第2・5・6層に分布する46).そして,興奮性錐体細胞のAISに並置した結節状のシナプス様構造を形成する(図1A)63).
5. inter-progenitor pool wiringのモデルとしてのショウジョウバエ視覚中枢
神経発生における空間的および時間的パターン形成はハエの中枢神経系を用いて精力的に研究されてきた.しかし,ハエの神経発生においては神経細胞の移動は非常にまれでほとんどみられないため,inter-progenitor pool wiringのような複雑な発生プロセスが存在するとは考えられていなかった.最近になって,我々を含む複数の研究グループによってハエ視覚中枢の細胞が発生過程において大幅に細胞移動することが見いだされた11–13, 18, 64).重要なことに,視覚中枢の後方に位置するGPC領域(glia precursor cells, tOPCとも呼ばれる)は接線方向の細胞移動によって主にOPC領域に由来するメダラの神経回路に加わるのである(図1B).このように,ハエの視覚中枢は哺乳類の大脳皮質と同様,inter-progenitor pool wiringの優れたモデル系であるといえる.
6. ショウジョウバエ視覚中枢における時間的パターン形成
メダラのほとんどの神経細胞は神経幹細胞(NB)から産生される.これらNBはメダラ前駆体の前側に位置するouter proliferation center(OPC)と呼ばれる領域の表層に位置する(図1B,図3).OPCのNBは神経上皮細胞(NE)から分化するが,すべてのNBが同時に分化するわけではなくproneural waveと呼ばれる分化の波によってNE領域の端から順番に産生する.bHLH転写因子Lethal of scute(Lsc)によって可視化されるproneural waveはNEのシート上を正中線側から側面側に向かって進行する(図3A)65, 66).対称分裂を繰り返すNE中にproneural waveがやってくると,NEは1列ずつNBに分化する.proneural waveの後方ではapical-basal方向の非対称分裂によって神経母細胞(GMC)を脳の深層側に向かって生み出す.GMCはさらに二つの細胞に分裂し,これらが神経細胞もしくはグリア細胞となる67).
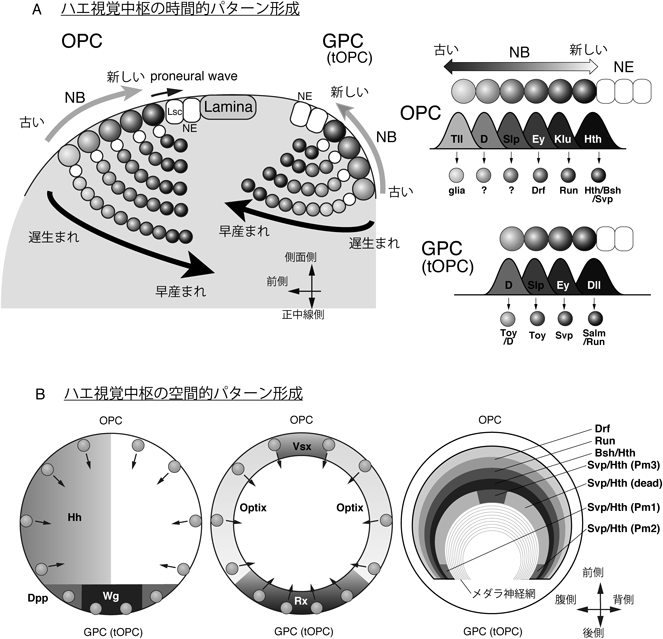
Proneural waveの後方で,NBは正中線側から側面側に向かって順に分化するため,正中線側ほど早く分化した古いNBが,側面側ほど遅く分化した新しいNBが位置することになる.このように,OPCのNBは正中線側から側面側に向かって産生時期に応じて並んでいるのである.一つ一つのNBは細胞系譜が共通した神経細胞群を脳の中心に向かって生み出し,初期に生まれた古い神経細胞はメダラの深層側に,後期に生まれた新しい神経細胞はメダラの表層に位置することになる(図1B,図3A)64).
我々と他のグループはOPCのNBにおいて一過的に発現するtemporal transcription factorを七つ同定した.Homothorax(Hth),Klumpfuss(Klu),Eyeless(Ey),Sloppy paired(Slp),Dichaete(D),Tailless(Tll)はこの順番にOPCのNBにおいて発現する(図3A)16, 17, 68, 69).HthはNEがNBに分化する前から発現を開始し,新しく分化したNBにおいても発現が維持される.その後,NBにおけるこれらの転写因子の発現は経時的にKlu, Ey, Slp, D, Tllと発現が変化していき,これらの転写因子の発現によってNBから生み出される神経細胞およびグリアの性質が決定される.このように,OPCのNBはハエの中枢神経系の他の部位とは異なるtemporal transcription factorを用いているのである3).
Hthが発現するNBはHthとBrain-specific-homeobox(Bsh)の両者を発現する神経細胞を生み出し,これがメダラ前駆体の最も深層側の領域に位置する.Kluを発現するNBはRunt(Run)陽性の神経細胞をHth/Bsh陽性神経細胞のすぐ表層側に産生する.NBは次にEyを発現し,Drifter(Drf)陽性神経細胞をRun陽性領域のすぐ表層側に産生する17, 68).このように,メダラのNBはHth, Klu, Eyと転写因子の発現を経時的に変化させ,それとともにHth/Bsh, Run, Drf陽性神経細胞を順に生み出すのである(図3).メダラのNBは放射方向にそして直線状にメダラ前駆体の深層側に向かって多数の神経細胞を生み出すので,その結果これらのメダラ神経細胞は同心円状の層構造を示すことになる(図3B)64).さらに,NotchシグナルによってGMCの非対称分裂が制御され,一つのGMCから生み出される二つの細胞運命が決定される16).
最近の研究によると,メダラの神経細胞はOPCだけでなく視覚中枢の後方に位置するGPC(tOPC)からも供給されることがわかっている11, 12, 18, 70).従来,GPCからはラミナグリアが生み出されると考えられていた70, 71).しかし,細胞系譜解析の結果から,GPCはラミナグリアだけでなくだけでなくメダラなど視覚中枢の各神経節に神経細胞を供給することがわかった11, 18, 72).
興味深いことに,GPCにおける神経発生もOPCと同様時間的パターン形成によって制御されている(図3A)18).GPCのNBはDistalless(Dll),Ey, Slp, Dと,経時的に異なる転写因子を発現し,次々と異なるタイプの神経細胞を産む.これら四つのtemporal transcription factorのうち,三つはOPCと共通しているが(Ey, Slp, D),DllはGPCに特異的となっている.GPCのNBから生まれたGMCは非対称分裂によって二つの細胞となる.ここでもNotchが非対称に活性化し,二つの細胞運命が制御される.二つの細胞のうち一つの細胞がアポトーシスによって除去されるので,一方の細胞のみが残る.Ey陽性NBから生まれる神経細胞はNotch-OFFのものが生き残るが,SlpおよびD陽性NBから生まれる神経細胞はNotch-ONのものが生き残る18).このような違いはあるが,OPCもGPCも同様に時間的パターン形成によって多様な神経細胞を生み出しているといえる.
後に述べるように,我々は2種類のlamina wide field neuron(Lawf1およびLawf2)がGPCに由来することを示したが,これらの神経細胞の運命がGPC内のどのような時間的パターン形成に従って決定されるのかは不明である(図1B)11).我々の結果はLawf神経細胞がWg陽性のGPC領域に由来することを示しているが,他のグループによってWg領域に隣接するDpp陽性領域に由来することが示唆されている(図1B)12).しかし,Wg領域とDpp領域はわずかにオーバーラップしているため,これらの結果は必ずしも矛盾するわけではない.Lawf神経細胞が生み出される詳細な分子機構は今後さらに解析される必要がある.
7. ショウジョウバエ視覚中枢における空間的パターン形成
WntおよびBMPリガンドをコードするwingless(wg)およびdecapentaplegic(dpp)遺伝子は視覚中枢の初期のパターン形成を制御すると考えられている(図3B)73).最近では,我々はWntシグナルがGPCの運命決定を制御することを示している10).GPCにおいてWntシグナルが抑制されるとGPC由来神経細胞が消失し,代わりにBsh陽性およびDrf陽性細胞といったOPC由来神経細胞が視覚中枢の後方に形成する.同様に,GPCにおけるWntシグナルの抑制によって,通常OPCにおいてのみ発現するHthが異所的に発現する.Wntシグナルの欠失によってGPCがOPCに変化したといえる.一方,OPCにおいてWntシグナルを異所的に活性化すると,OPCがGPCに変化し,GPCおよびGPC由来神経細胞が視覚中枢の前側の領域に形成する10).
GPCにおける空間パターン形成はよくわかっていないが,少なくとも背側と腹側のGPCは異なる神経細胞を生み出すことを我々は見いだしている.GPCから生まれるLawf神経細胞のうちLawf1とLawf2神経細胞はそれぞれ腹側および背側のGPCにおいて産生される(図1B)11).細胞系譜の解析の結果,背側GPCはOPCの背側に位置する神経細胞を生み,これらはLawf2に特異的な転写因子Lim1を発現する.このように,何らかの因子が背腹軸に沿ってGPCの空間パターン形成を制御していると考えられる.
GPCと比べて,OPCははるかに多くの神経細胞を生み出す.OPC側方側のNEから生まれるラミナは,Wntファミリーの分泌性タンパク質をコードするDWnt4がラミナの腹側特異的に発現することによって背腹軸方向に区画化されている74).一方,メダラNBはOPC正中線側のNEから生まれる.proneural waveによって開始するOPC NBの時間的パターン形成に加えて,OPCは空間的にも細分化されている(図3B)13).OPCは転写因子Vsx1, Optix, Rxの発現によって背腹軸に沿って三つの領域に分割される.それらに加えて分泌性因子をコードするhedgehog(hh)の発現が初期の視覚中枢において腹側半分の領域を規定する.このように,OPCにおいて同じ産生時期のNBから生まれる神経細胞であっても,これらの空間情報によって異なるタイプの神経細胞に分化するのである.たとえば,Hth陽性の最も新しいNBはBsh陽性およびSvp陽性神経細胞を生み出す(図3A).Bsh陽性細胞はOPC中の位置にかかわらず常にMi1神経細胞に分化する一方,Svp陽性細胞の運命はVsx1, Optix, Rxによって与えられる位置情報に影響される13).OPCの前側に位置するVsx陽性領域,および後方に位置するRx陽性領域において,Svp陽性細胞はそれぞれPm3およびPm1/2神経細胞に分化する.一方,背側もしくは腹側に位置するOptix陽性領域においてSvp陽性細胞はアポトーシスによって除去される.このように,ショウジョウバエ胚期の中枢神経系や脊椎動物の脊髄と同様,ハエの視覚中枢においても時間的および空間的パターン形成によって神経細胞の多様性が生み出されるのである2, 3).
8. ショウジョウバエ視覚中枢におけるinter-progenitor pool wiring
OPCはメダラ神経細胞の最も主要な供給源であるが,GPCもメダラに神経細胞を供給する11, 18, 72).しかし,GPC由来神経細胞がOPCに向かって移動するルートとOPCおよびGPC由来神経細胞によって形成される神経回路の実体は同定されていなかった.我々はGPC由来神経細胞がメダラ神経網に沿った移動経路を通って接線方向に移動し,幼虫期においてOPCの最も深層側の領域に到達することを見いだした(図1B)11).Slit/RoboシグナルがこれらGPC由来神経細胞の移動経路を確立するうえで重要な役割を果たすと考えられる.GPC由来神経細胞によって産生されたSlitはOPC由来神経細胞およびinner proliferating center(IPC)の細胞においてそれぞれ発現するRobo3およびRobo2によって受容されるが(図1B),Slit/Roboシグナルが阻害されると,GPC由来神経細胞の移動に異常が生じるのである11).これらのOPC由来神経細胞は蛹期においてもう一度大規模に移動するが13, 64),これらの細胞移動の生物学的な意義は不明である.
2種類のlamina wide field神経細胞であるLawf1とLawf2は幼虫期において最も深層側の同心円ゾーン上に位置する(図1B)64).すでに述べたように,細胞系譜解析の結果から,GPCの背側および腹側の領域からそれぞれLawf2およびLawf1神経細胞が生まれる11).これらの神経細胞は蛹期になると再び移動を開始し,メダラ内に均一に分布するようになる.
このような細胞移動の過程で,Lawf1/2神経細胞はラミナに向かって軸索を伸ばし,ラミナ中に広範囲の軸索終末を形成する.一方,メダラ内においても広範囲の樹状突起を伸長し,ラミナとメダラ両方において複数のカラムをカバーする傘のような特徴的な形態を示す(図1B)64, 75–77).これらの形態的特徴から,Lawf1/2神経細胞はメダラの複数のカラムにおける情報を統合し,その情報をラミナに対してフィードバックする役割を持つと考えられる.Lawf2細胞については,低周波数シグナルを減じることで動体認識回路の働きを補正することが示されている77).このように,GPCに由来するLawf神経細胞はラミナおよびメダラにおける他の神経細胞とともに神経回路を形成するため,inter-progenitor pool wiringの好例であるといえる.
Inter-progenitor pool wiringは哺乳類の大脳皮質とショウジョウバエの視覚中枢においてみられることから,類似したストラテジーが他の脳領域においても用いられ,神経回路の多様性向上に貢献している可能性が十分に考えられる.腹側終脳の一部であるPOAにおいてDbx1陽性の神経前駆領域から生まれた興奮性および抑制性神経細胞が扁桃体まで細胞移動することが示されている78).このように,背側終脳と扁桃体の間においてもinter-progenitor pool wiringがみられるのである.小脳も由来の異なる神経細胞からなる.GABA作動性の抑制性神経細胞であるプルキンエ細胞は中脳のVZから生まれ,顆粒細胞は外顆粒層から生まれる79).外顆粒層に位置する顆粒細胞の前駆細胞は菱脳唇に由来するが,菱脳唇から前駆細胞が移動するメカニズムはよくわかっていない.視床の神経細胞は尾側(pTH-C)と吻側(pTH-R)の2か所の前駆領域に由来する.pTH-Cの前駆細胞はグルタミン酸作動性の興奮性神経細胞を,pTH-Rの前駆細胞はGABA作動性の抑制性神経細胞を生み出す80).
ハエの視覚中枢はOPCとGPC以外にも,IPCと呼ばれる神経前駆領域を持つ(図1B)81).IPCの深部に位置するNE細胞はIPCの表層に向かって移動し,その後NBへと分化する.IPC深部に由来するNBはメダラやラミナに投射する神経細胞を生み出し,OPC由来の神経細胞とともに神経回路を構築する.分化した神経細胞ではなく神経前駆細胞が移動することから,IPCにおいてみられるこの現象は小脳の顆粒細胞前駆細胞が菱脳唇から外顆粒層に向かって移動する現象と類似している79).このように,inter-progenitor pool wiringは神経細胞の移動だけでなく神経前駆細胞の移動によっても実現されることがわかる.
ハエの脳は哺乳類の脳に比べてはるかに単純であると考えられてきたが,最近の研究からハエの脳の発生もinter-progenitor pool wiringのような複雑なメカニズムによって成立していることが明らかとなった.哺乳類の大脳皮質,小脳,視床,そしてハエの視覚中枢において類似した戦略が使われていることから,inter-progenitor pool wiringは神経回路の多様性を増すために使われる進化的に保存された発生戦略であるといえる.さらに,ハエの視覚中枢はinter-progenitor pool wiringの分子機構を研究するうえでの強力なモデル系であるということもできる.
大脳皮質はinter-progenitor pool wiringの好例であるが,その分子機構の大部分は未解明である.大脳新皮質と基底核原基においても,空間的パターン形成および時間的パターン形成のメカニズムに比べると,興奮性神経細胞と抑制性神経細胞による回路形成の過程についてははっきりしない点が多い.inter-progenitor pool wiringによる回路形成の最終ステップは細胞移動の終結と由来の異なる神経細胞どうしの認識の二つのプロセスに分けられる42).基底核原基に由来する神経細胞は接線方向の移動経路に沿った位置情報を認識し,移動を停止すると考えられる.これらの細胞はさらに最終的な標的神経を認識し,シャンデリア細胞と錐体細胞においてみられるような特異的なシナプス結合を形成すると考えられる(図1A).ただし,これらの二つのプロセスは完全に分離できるとは限らない.たとえば標的となる神経細胞が細胞移動終結のシグナルも同時に供給しているかもしれない.
ハエの視覚中枢においてGPC由来神経細胞はGPCの外に出た後,幼虫期において一過的に細胞集団を形成する(図1B)11).興味深いことに,これらの細胞は蛹期においてもう一度移動を開始し,メダラ全体に分散する.これら神経細胞のラミナおよびメダラにおける広範な投射パターンおよび動体認識における役割から,GPC由来のLawf神経細胞はラミナのL1神経やメダラのMi1神経細胞とともに動体認識回路を形成していると考えられる(図1B)77).しかし,GPC由来Lawf神経細胞とその他のOPC由来神経細胞が正確に神経回路を形成するメカニズムについてはほとんど何もわかっていない.興味深いことに,Lawf神経細胞は幼虫期においてすでに最終的な標的に向かって軸索を伸長している.これらの軸索終末は蛹期における2回目の細胞移動を開始する前からメダラ神経網の表面に突起を形成しているため,Lawf神経細胞は幼虫期の非常に早い時期において最終的な標的を認識していると考えられる11).大脳皮質においてinter-progenitor pool wiringの最終ステップは細胞移動の終結と由来の異なる神経細胞どうしの認識であると考えられるが42),ハエの脳においては後者の方が先に起きているのかもしれない.このように,Lawf神経細胞による標的認識の分子機構は特に興味深い.
このように,我々は脳の発生におけるinter-progenitor pool wiringの重要性を認識したばかりであり,その分子機構はほとんど未解明であるが,哺乳類とハエの脳において進化的に保存されたメカニズムに注目することによって,脳の神経回路形成における深遠な戦略の全貌を理解することができると期待される.
引用文献References
1) Jessell, T.M. (2000) Natl. Rev., 1, 20–29.
2) Guillemot, F. (2007) Development, 134, 3771–3780.
3) Kohwi, M. & Doe, C.Q. (2013) Nat. Rev. Neurosci., 14, 823–838.
4) Vue, T.Y., Aaker, J., Taniguchi, A., Kazemzadeh, C., Skidmore, J.M., Martin, D.M., Martin, J.F., Treier, M., & Nakagawa, Y. (2007) J. Comp. Neurol., 505, 73–91.
5) Livesey, F.J. & Cepko, C.L. (2001) Nat. Rev. Neurosci., 2, 109–118.
6) Kim, J., Wu, H.H., Lander, A.D., Lyons, K.M., Matzuk, M.M., & Calof, A.L. (2005) Science, 308, 1927–1930.
7) Anderson, S.A., Eisenstat, D.D., Shi, L., & Rubenstein, J.L. (1997) Science, 278, 474–476.
8) Kriegstein, A.R. & Noctor, S.C. (2004) Trends Neurosci., 27, 392–399.
9) Technau, G.M., Berger, C., & Urbach, R. (2006) Dev. Dyn., 235, 861–869.
10) Suzuki, T., Trush, O., Yasugi, T., Takayama, R., & Sato, M. (2016) J. Neurosci., 36, 6503–6513.
11) Suzuki, T., Hasegawa, E., Nakai, Y., Kaido, M., Takayama, R., & Sato, M. (2016) Cell Reports, 15, 499–509.
12) Chen, Z., Del Valle Rodriguez, A., Li, X., Erclik, T., Fernandes, V.M., & Desplan, C. (2016) Cell Reports, doi: 10.1016/j.celrep.2016.03.061.
13) Erclik, T., Li, X., Courgeon, M., Bertet, C., Chen, Z., Baumert, R., Ng, J., Koo, C., Arain, U., Behnia, R., del Valle Rodriguez, A., Senderowicz, L., Negre, N., White, K.P., & Desplan, C. (2017) Nature, 541, 365–370.
14) Isshiki, T., Pearson, B., Holbrook, S., & Doe, C.Q. (2001) Cell, 106, 511–521.
15) Bayraktar, O.A. & Doe, C.Q. (2013) Nature, 498, 449–455.
16) Li, X., Erclik, T., Bertet, C., Chen, Z., Voutev, R., Venkatesh, S., Morante, J., Celik, A., & Desplan, C. (2013) Nature, 498, 456–462.
17) Suzuki, T., Kaido, M., Takayama, R., & Sato, M. (2013) Dev. Biol., 380, 12–24.
18) Bertet, C., Li, X., Erclik, T., Cavey, M., Wells, B., & Desplan, C. (2014) Cell, 158, 1173–1186.
19) Cajal, S.R. & Sanchez, D. (1915) Trab. Lab. Invest. Biol., 13, 1–167.
20) Sanes, J.R. & Zipursky, S.L. (2010) Neuron, 66, 15–36.
21) Nobrega-Pereira, S. & Marin, O. (2009) Cereb. Cortex, 19(Suppl 1), i107–i113.
22) Hebert, J.M. & Fishell, G. (2008) Nat. Rev. Neurosci., 9, 678–685.
23) Fukuchi-Shimogori, T. & Grove, E.A. (2001) Science, 294, 1071–1074.
24) Toyoda, R., Assimacopoulos, S., Wilcoxon, J., Taylor, A., Feldman, P., Suzuki-Hirano, A., Shimogori, T., & Grove, E.A. (2010) Development, 137, 3439–3448.
25) Greig, L.C., Woodworth, M.B., Galazo, M.J., Padmanabhan, H., & Macklis, J.D. (2013) Nat. Rev. Neurosci., 14, 755–769.
26) Huang, Z.J. (2014) Neuron, 83, 1284–1302.
27) Rallu, M., Machold, R., Gaiano, N., Corbin, J.G., McMahon, A.P., & Fishell, G. (2002) Development, 129, 4963–4974.
28) Ericson, J., Muhr, J., Placzek, M., Lints, T., Jessell, T.M., & Edlund, T. (1995) Cell, 81, 747–756.
29) Xu, Q., Wonders, C.P., & Anderson, S.A. (2005) Development, 132, 4987–4998.
30) Gulacsi, A.A. & Anderson, S.A. (2008) Nat. Neurosci., 11, 1383–1391.
31) Butt, S.J., Sousa, V.H., Fuccillo, M.V., Hjerling-Leffler, J., Miyoshi, G., Kimura, S., & Fishell, G. (2008) Neuron, 59, 722–732.
32) Xu, Q., Guo, L., Moore, H., Waclaw, R.R., Campbell, K., & Anderson, S.A. (2010) Neuron, 65, 328–340.
33) Sussel, L., Marin, O., Kimura, S., & Rubenstein, J.L. (1999) Development, 126, 3359–3370.
34) Kessaris, N., Magno, L., Rubin, A.N., & Oliveira, M.G. (2014) Curr. Opin. Neurobiol., 26, 79–87.
35) Flames, N., Pla, R., Gelman, D.M., Rubenstein, J.L., Puelles, L., & Marín, O. (2007) J. Neurosci., 27, 9682–9695.
36) Gelman, D., Griveau, A., Dehorter, N., Teissier, A., Varela, C., Pla, R., Pierani, A., & Marín, O. (2011) J. Neurosci., 31, 16570–16580.
37) Alsio, J.M., Tarchini, B., Cayouette, M., & Livesey, F.J. (2013) Proc. Natl. Acad. Sci. USA, 110, E716–E725.
38) Srinivasan, K., Leone, D.P., Bateson, R.K., Dobreva, G., Kohwi, Y., Kohwi-Shigematsu, T., Grosschedl, R., & McConnell, S.K. (2012) Proc. Natl. Acad. Sci. USA, 109, 19071–19078.
39) Frantz, G.D., Weimann, J.M., Levin, M.E., & McConnell, S.K. (1994) J. Neurosci., 14, 5725–5740.
40) Morimoto-Suzki, N., Hirabayashi, Y., Tyssowski, K., Shinga, J., Vidal, M., Koseki, H., & Gotoh, Y. (2014) Development, 141, 4343–4353.
41) Bartolini, G., Ciceri, G., & Marin, O. (2013) Neuron, 79, 849–864.
42) Guo, J. & Anton, E.S. (2014) Trends Cell Biol., 24, 342–351.
43) Miyoshi, G., Butt, S.J., Takebayashi, H., & Fishell, G. (2007) J. Neurosci., 27, 7786–7798.
44) Miyoshi, G., Hjerling-Leffler, J., Karayannis, T., Sousa, V.H., Butt, S.J., Battiste, J., Johnson, J.E., Machold, R.P., & Fishell, G. (2010) J. Neurosci., 30, 1582–1594.
45) Inan, M. & Anderson, S.A. (2014) Curr. Opin. Neurobiol., 26, 142–148.
46) Taniguchi, H., Lu, J., & Huang, Z.J. (2013) Science, 339, 70–74.
47) Zimmer, G., Garcez, P., Rudolph, J., Niehage, R., Weth, F., Lent, R., & Bolz, J. (2008) Eur. J. Neurosci., 28, 62–73.
48) Marin, O., Yaron, A., Bagri, A., Tessier-Lavigne, M., & Rubenstein, J.L. (2001) Science, 293, 872–875.
49) Flames, N., Long, J.E., Garratt, A.N., Fischer, T.M., Gassmann, M., Birchmeier, C., Lai, C., Rubenstein, J.L., & Marín, O. (2004) Neuron, 44, 251–261.
50) Yau, H.J., Wang, H.F., Lai, C., & Liu, F.C. (2003) Cereb. Cortex, 13, 252–264.
51) Stanco, A., Szekeres, C., Patel, N., Rao, S., Campbell, K., Kreidberg, J.A., Polleux, F., & Anton, E.S. (2009) Proc. Natl. Acad. Sci. USA, 106, 7595–7600.
52) Li, G., Adesnik, H., Li, J., Long, J., Nicoll, R.A., Rubenstein, J.L., & Pleasure, S.J. (2008) J. Neurosci., 28, 1085–1098.
53) Lopez-Bendito, G., Sánchez-Alcañiz, J.A., Pla, R., Borrell, V., Picó, E., Valdeolmillos, M., & Marín, O. (2008) J. Neurosci., 28, 1613–1624.
54) Sanchez-Alcaniz, J.A., Haege, S., Mueller, W., Pla, R., Mackay, F., Schulz, S., López-Bendito, G., Stumm, R., & Marín, O. (2011) Neuron, 69, 77–90.
55) Stumm, R.K., Zhou, C., Ara, T., Lazarini, F., Dubois-Dalcq, M., Nagasawa, T., Höllt, V., & Schulz, S. (2003) J. Neurosci., 23, 5123–5130.
56) Tanaka, D.H., Mikami, S., Nagasawa, T., Miyazaki, J., Nakajima, K., & Murakami, F. (2010) Cereb. Cortex, 20, 2810–2817.
57) Tiveron, M.C., Rossel, M., Moepps, B., Zhang, Y.L., Seidenfaden, R., Favor, J., König, N., & Cremer, H. (2006) J. Neurosci., 26, 13273–13278.
58) Wang, Y., Li, G., Stanco, A., Long, J.E., Crawford, D., Potter, G.B., Pleasure, S.J., Behrens, T., & Rubenstein, J.L. (2011) Neuron, 69, 61–76.
59) Hevner, R.F., Daza, R.A., Englund, C., Kohtz, J., & Fink, A. (2004) Neuroscience, 124, 605–618.
60) Pla, R., Borrell, V., Flames, N., & Marin, O. (2006) J. Neurosci., 26, 6924–6934.
61) Lodato, S., Rouaux, C., Quast, K.B., Jantrachotechatchawan, C., Studer, M., Hensch, T.K., & Arlotta, P. (2011) Neuron, 69, 763–779.
62) Ye, Z., Mostajo-Radji, M.A., Brown, J.R., Rouaux, C., Tomassy, G.S., Hensch, T.K., & Arlotta, P. (2015) Neuron, 88, 475–483.
63) Somogyi, P. (1977) Brain Res., 136, 345–350.
64) Hasegawa, E., Kitada, Y., Kaido, M., Takayama, R., Awasaki, T., Tabata, T., & Sato, M. (2011) Development, 138, 983–993.
65) Yasugi, T., Umetsu, D., Murakami, S., Sato, M., & Tabata, T. (2008) Development, 135, 1471–1480.
66) Sato, M., Yasugi, T., Minami, Y., Miura, T., & Nagayama, M. (2016) Proc. Natl. Acad. Sci. USA, 113, E5153–E5162.
67) Egger, B., Boone, J.Q., Stevens, N.R., Brand, A.H., & Doe, C.Q. (2007) Neural Dev., 2, 1.
68) Suzuki, T. & Sato, M. (2014) Dev. Growth Differ., 56, 491–498.
69) Suzuki, T., Takayama, R., & Sato, M. (2016) Dev. Biol., 409, 343–353.
70) Perez, S.E. & Steller, H. (1996) J. Neurobiol., 30, 359–373.
71) Chotard, C. & Salecker, I. (2007) Neuron Glia Biol., 3, 17–25.
72) Dearborn, R. Jr. & Kunes, S. (2004) Development, 131, 2291–2303.
73) Kaphingst, K. & Kunes, S. (1994) Cell, 78, 437–448.
74) Sato, M., Umetsu, D., Murakami, S., Yasugi, T., & Tabata, T. (2006) Nat. Neurosci., 9, 67–75.
75) Fischbach, K.F. & Dittrich, A.P.M. (1989) Cell Tissue Res., 258, 441–475.
76) Takemura, S.Y., Bharioke, A., Lu, Z., Nern, A., Vitaladevuni, S., Rivlin, P.K., Katz, W.T., Olbris, D.J., Plaza, S.M., Winston, P., Zhao, T., Horne, J.A., Fetter, R.D., Takemura, S., Blazek, K., Chang, L.A., Ogundeyi, O., Saunders, M.A., Shapiro, V., Sigmund, C., Rubin, G.M., Scheffer, L.K., Meinertzhagen, I.A., & Chklovskii, D.B. (2013) Nature, 500, 175–181.
77) Tuthill, J.C., Nern, A., Rubin, G.M., & Reiser, M.B. (2014) Neuron, 82, 887–895.
78) Hirata, T., Li, P., Lanuza, G.M., Cocas, L.A., Huntsman, M.M., & Corbin, J.G. (2009) Nat. Neurosci., 12, 141–149.
79) Leto, K., Arancillo, M., Becker, E.B., Buffo, A., Chiang, C., Ding, B., Dobyns, W.B., Dusart, I., Haldipur, P., Hatten, M.E., Hoshino, M., Joyner, A.L., Kano, M., Kilpatrick, D.L., Koibuchi, N., Marino, S., Martinez, S., Millen, K.J., Millner, T.O., Miyata, T., Parmigiani, E., Schilling, K., Sekerková, G., Sillitoe, R.V., Sotelo, C., Uesaka, N., Wefers, A., Wingate, R.J., & Hawkes, R. (2016) Cerebellum, 15, 789–828.
80) Gezelius, H. & Lopez-Bendito, G. (2016) Dev. Neurobiol., 77, 830–843.
81) Apitz, H. & Salecker, I. (2015) Nat. Neurosci., 18, 46–55.
著者紹介Author Profile
 佐藤 純(さとう まこと)
佐藤 純(さとう まこと)金沢大学新学術創成研究機構教授.博士(理学).
略歴1995年東京大学理学部生物化学科卒業.2000年同大学院理学系研究科生物化学専攻博士課程修了(博士(理学)取得).00~02年カリフォルニア大学サンフランシスコ校Kornberg研究室博士研究員.02~08年東京大学分子細胞生物学研究所多羽田研究室助手および助教.08~12年金沢大学フロンティアサイエンス機構特任准教授(PI).12~15年金沢大学脳・肝インターフェースメディシン研究センター教授.15年より現職.
研究テーマと抱負ショウジョウバエの視覚中枢をモデル系として神経発生の研究を進めており,最近では特に脳のカラム構造の形成過程を制御する分子機構に着目しています.それと同時に,数理モデルを活用した手法に力を入れており,数理モデルから得られた予想を実験的に検証することによって,通常の生命科学実験だけでは理解できない生命現象を解明したいと考えています.
ウェブサイトhttp://fsosato.w3.kanazawa-u.ac.jp/index.html
趣味カラオケ,スキー,ジョギング,料理.