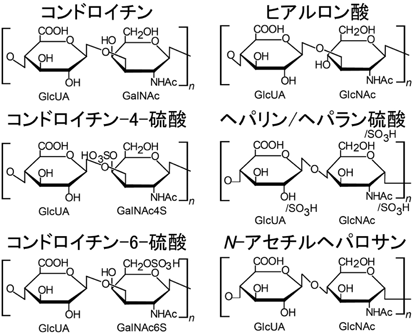
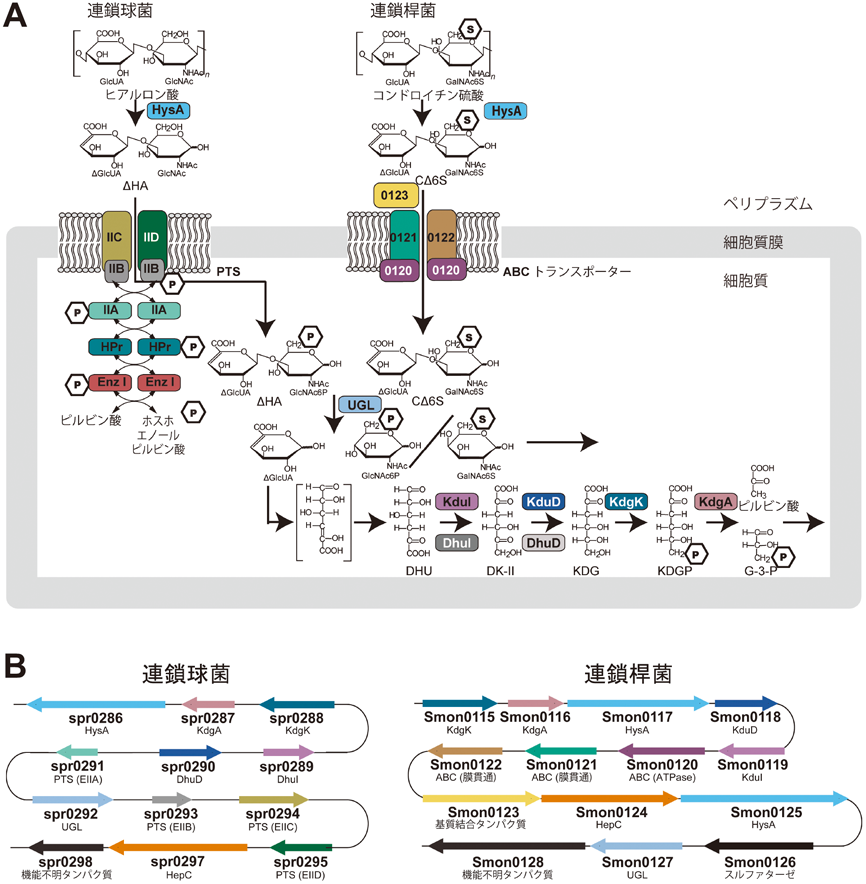
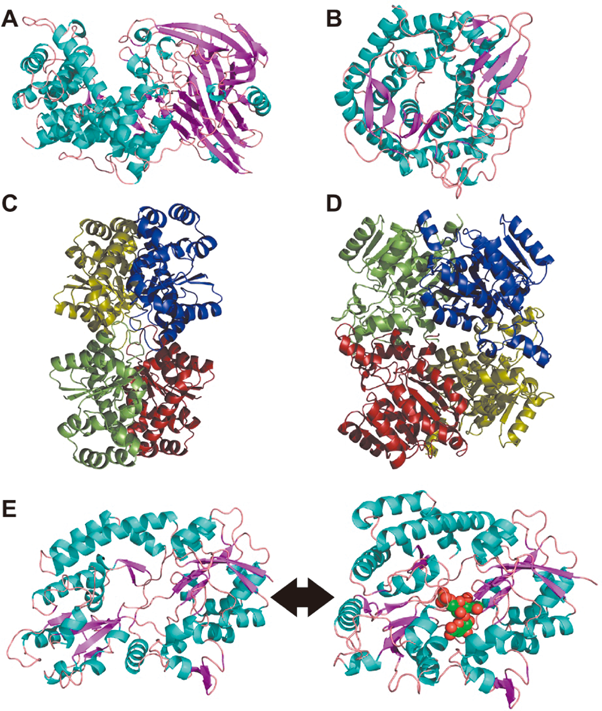
病原細菌による宿主細胞外グリコサミノグリカンの断片化・輸送・分解・代謝に関わる分子機構Bacterial molecular mechanism for depolymerization, import, degradation and metabolism of host extracellular matrices glycosaminoglycans
京都大学農学研究科Graduate School of Agriculture, Kyoto University ◇ 〒611–0011 京都府宇治市五ヶ庄 ◇ Gokasho, Uji, Kyoto 611–0011, Japan