我々の生活にもなじみ深い「酵母」は,1990年では597種類1),2000年には678種類2)と,次々と新種が同定されている真核微生物(子嚢菌類)である.これらのうち,細胞の一端から芽が出るように生育し,母細胞から離れて娘細胞として増殖する「出芽酵母」は,51属からなる大きな集団を形成しており,お酒の発酵などに関わる有名な出芽酵母Saccharomyces cerevisiaeだけでなく,Saccharomyces属には,この他に26種が存在する.
一方,動物細胞と同じように核が2個に分裂後,細胞の中央に隔壁を形成して分かれることで増殖する「分裂酵母」は,2010年に新たに1種類が報告された3)ものの,1属(Schizosaccharomyces属),4種(S. pombe, S. octosporus, S. japonicus, S. cryophilus)が同定されているのみである.
この中で,分裂酵母S. pombe(以降,分裂酵母と記す)は主に一倍体で生育し,その細胞分裂の制御機構は高等生物の機構と類似している.分裂酵母は出芽酵母よりゲノムに多くのイントロンを持ち,そのスプライシング機構が動物細胞と類似しており,遺伝子上でのpolyA添加シグナルやプロモーターに関しても類似点が多いことなどから4–6),旧来より基礎・応用研究ともに活発な解析が行われている菌株である7–10).分裂酵母のオンラインデータベース(PomBase:http://www.pombase.org/)11)では,2017年9月の時点で5118個のタンパク質が登録されており,これらの中で600個が出芽酵母Saccharomyces cerevisiaeにはないタンパク質としてあげられている.この600個のタンパク質のうち,343個(57%)がヒトのオルソログに相当する.さらに,この343個の中で,48個(14%)が42種類の疾患に関連しているタンパク質である(上位三つをあげると,がん関連が16個,代謝障害が10個,神経障害が10個).このように,分裂酵母は出芽酵母では解析しにくい,高等動物にも保存されているタンパク質の機能解析を行う宿主としての高いポテンシャルを持っていることを示唆している.本稿では,分裂酵母のオルガネラの中でも,分泌経路のゴルジ体を中心に,層板の特徴やそこで機能しているタンパク質から垣間みえる生命現象を紹介したい.
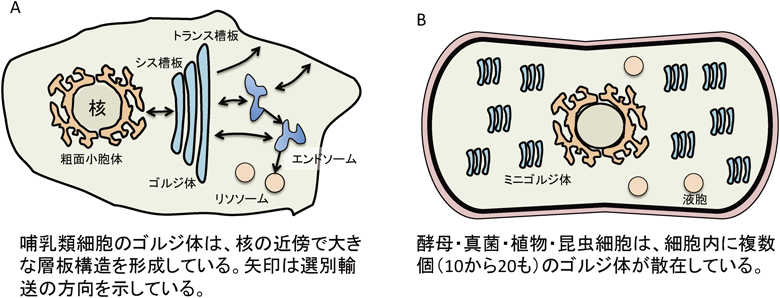
リボソームで合成されたタンパク質は通常,そのタンパク質が機能するオルガネラに輸送されるか,細胞外または細胞膜や細胞表層(細胞壁と細胞膜の間隙)に輸送される.このようなタンパク質の選別・分泌機構は多くの微生物から高等動物に至るまですべての生命体に保存されている.その中でもゴルジ体は,真核細胞の分泌経路上でタンパク質選別輸送機構とタンパク質の糖鎖修飾機構の中間地点(ハブ)として重要な役割を担っている12).ゴルジ体は扁平な袋状の膜構造がほぼ一定の間隔で槽を形成しており,各槽は粗面小胞体側からシス,メディアル,トランス槽板と呼ばれる.また,ゴルジ体の末端には選別輸送や輸送小胞の融合を行う非常に動的な区画であるゴルジネットワークが存在する.各槽板で機能が異なり,シス槽板では粗面小胞体から輸送されてきた輸送小胞の受け取りや粗面小胞体で機能するタンパク質の逆行輸送が行われる.また各種糖転移酵素により糖鎖修飾が始まり,メディアル槽板,トランス槽板と進むにつれさらなる糖鎖修飾が行われていく.そしてゴルジ体の最終区画であるトランスゴルジネットワークではエンドソームや細胞表層への選別輸送,細胞膜やエンドソームからの輸送小胞の受け取りを行っている.このように,ゴルジ体はタンパク質選別輸送機構や糖鎖修飾過程において重要な機能を果たしている(図1A).
ゴルジ体の形態は生物間で異なっており,一般的な哺乳類細胞のゴルジ層板はシスからトランス槽板が積み重なったものを一つの単位(本稿では,ミニゴルジ体として表記)として,それらが横方向に微小管やゴルジマトリックスタンパク質を介して連結融合し,核に隣接して局在するのに対し,ハエ・植物・菌類ではミニゴルジ体が細胞質に散在している12).このような,塊になっているか,散在しているかの違いは,ゴルジ体の細胞周期によるダイナミックな変化にも影響している.動物細胞ではゴルジ体が細胞周期によって解体と再構成の制御を受けており,ゴルジマトリックスタンパク質の制御だけでなく12, 13),ゴルジ体における小胞輸送の膜融合装置であるt-SNARE Syn5のユビキチン化の有無によっても,解体が促進するか否かが決まることがわかってきた14).
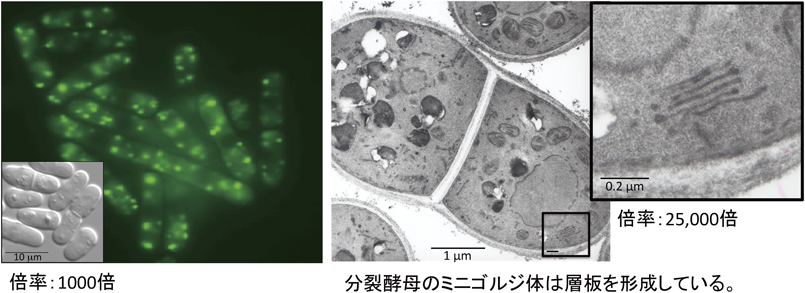
酵母におけるゴルジ体の形態は酵母種(属)間で異なっており,出芽酵母S. cerevisiaeの場合ではミニゴルジ体から,さらに各槽板が離れた状態で細胞質内に散在している15).この特徴と超解像共焦点レーザー顕微鏡を駆使して,各槽板間の逆行輸送と協調した槽成熟モデルが明快に示されている16, 17).一方で,分裂酵母S. pombeや出芽酵母であるメタノール資化性酵母Pichia pastorisでは多細胞生物にもみられるような,ミニゴルジ体が観察されている18)(図1B).分裂酵母のミニゴルジ体は一つの細胞に10から20個存在し,平均して3層以上(1から9層)の槽板が積み重なった状態として観察される(図2).このミニゴルジ体は微小管重合阻害剤(チアベンダゾール)を含む培地で培養すると,ミニゴルジ体の各槽板が解離するというよりは,各槽板の小胞化によって,1槽のみの層板構造が約2割から約7割に上昇する19).この層板構造の消失は,チアベンダゾールの除去により可逆的に回復する19)ことから,ミニゴルジ体の散在した各槽板を元の場所につなぎ戻す機構が存在することを示唆している.分裂酵母のゴルジ体膜表在性タンパク質としては,シス槽板の形成に関わるGRAB(GRIP-related Arf-binding)ドメイン20)を有するRud3,ゴルジ体膜の細胞質側への局在を可能にするGRIP(golgin-97, RanBP2α, Imh1p and p230/golgin-245)ドメイン21)を有するGrp1,槽板間の輸送に関わるヒトRab6ホモログであるRyh122),槽板間の繋留に関わるGRASPファミリーホモログであるGrh1などが保存されている.筆者らはこれらの欠損株の多くが,糖鎖修飾に障害がみられることから,ミニゴルジ体の機能に関わっていることを確認しているが,各槽板の積層化やミニゴルジ体の個数,細胞周期やストレス環境における散在場所の制御などへの関与は不明であり興味深い.
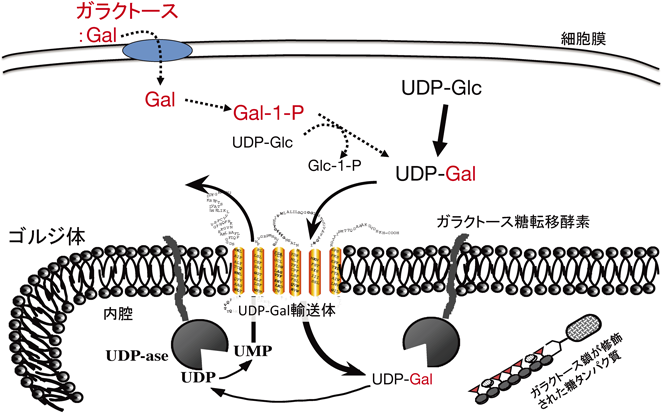
散在したミニゴルジ体を通過する糖タンパク質の多くは糖鎖が修飾されるが,酵母では唯一,分裂酵母はガラクトース鎖が修飾された糖タンパク質を分泌する23).一般的に酵母はマンノースのみから構成される糖鎖がゴルジ体で修飾され,細胞外へ分泌される糖タンパク質には50から100糖もの糖鎖が各種結合様式に対応した糖転移酵素によって修飾される.分裂酵母にはマンノース転移酵素だけでなく,10種類ものガラクトース鎖転移酵素が存在し24),基質となる糖ヌクレオチド(UDP-ガラクトース)の細胞質側からゴルジ体内腔への輸送体も動物細胞と同様に存在する(図3).UDP-ガラクトース生合成経路25)や輸送体が欠損した分裂酵母は,細胞表層のガラクトース鎖が欠損するため,桿状の細胞形態が保てず,丸みを帯びた形態になり,アミノ糖系抗生物質に感受性を示すようになる.細胞表層のガラクトース鎖は分裂酵母どうしを見分ける際の目印になっているため,ガラクトース鎖を認識するレクチン様タンパク質26)が結合できなくなることにより,窒素源飢餓のような貧栄養条件下ではガラクトース鎖を介した非性的凝集ができなくなり,胞子形成過程に進めず,結果的に致死となる.さらに,分裂酵母のガラクトース鎖は一部がピルビン酸化されており27),構造的にも機能的にも,ヒトの糖鎖の末端に修飾されるシアル酸と類似している点が興味深い28).これらのガラクトース鎖生合成に関わるタンパク質は,修飾される順序から,ミニゴルジ体のメディアルからトランス槽板で機能していると考えられるが,適した槽板への局在化機構や役割を終えた糖転移酵素の分解機構はほとんど明らかになっていない.
3. 分裂酵母のコイルドコイルタンパク質の過剰発現(機能破綻)からみえてくる現象
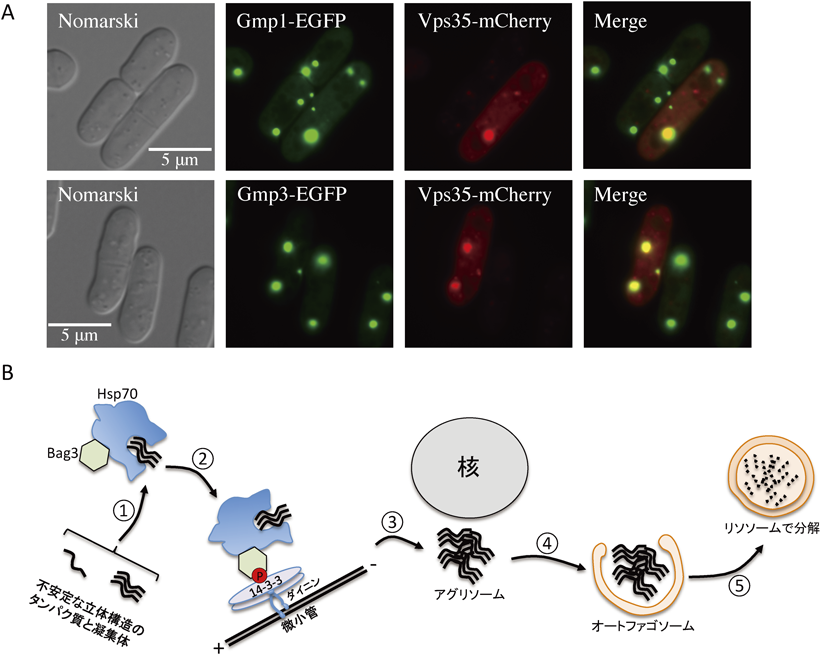
上記にあげたようなゴルジ体膜表在性のタンパク質にはコイルドコイルを有する特徴があり,ホモ・ヘテロな多量体化を促すことにより機能を発揮する.分裂酵母には1049個ものコイルドコイルタンパク質が存在し,機能不明なものが多い.筆者らがゴルジ体膜を含む画分からTOF-MS解析により同定した,ゴルジ体膜表層で機能していると考えられる機能未知なコイルドコイルタンパク質(Gmpファミリー)の中には,遺伝子破壊株の表現型は軽度な糖鎖修飾障害を示す程度だが,過剰発現させると,その構造的性質が増強される影響からか,細胞内に巨大な凝集塊を形成するものもある.さまざまなオルガネラマーカーとの共局在を調べたところ,この凝集塊は,エンドソームとゴルジ体間の逆行輸送に関わるレトロマー複合体のサブユニットVps35と一致した(図4A).このGmpタンパク質の過剰発現により出現した,いわば,人為的な巨大エンドソーム様オルガネラではあるが,エンドソーム画分のみを凝集化させる現象はまれであり興味深い表現型である.この凝集塊は微小管重合阻害剤により細胞質に拡散したことから,Gmp過剰発現による凝集体は微小管依存的に集約されていることが推察される.次に,培地の栄養条件による凝集体への影響を調べるため,窒素飢餓培地および低グルコース培地を用いた結果,凝集体の有意な減少が観察された.窒素飢餓状態でS. cerevisiaeは,TORC1を不活性化し細胞増殖機構などを制御することが知られている29, 30).また,オートファジーおよびプロテアソームを含むタンパク質分解経路が窒素再循環および細胞活性化の調節において主要な役割を果たしている31).一方,グルコース抑制時にS. cerevisiaeでは,プロテインキナーゼSnf1が,S. pombeではS. cerevisiaeのSnf1と相同性の高いプロテインキナーゼSsp2が活性化され,下流のストレス応答遺伝子の活性化やグルコース以外の炭素源代謝関連遺伝子の活性化などを行う29, 32, 33).これらのことよりGmp過剰発現による凝集体は,栄養飢餓時に抑制されるタンパク質によって維持されている,またはオートファジーによって分解されていることが示唆される.
この特徴と類似する生命現象は,動物細胞においてタンパク質凝集体を収集,分解するために形成されるアグリソームと呼ばれる構造物があげられる.細胞は,絶えず必要なタンパク質を新規に生産し続け,不要なタンパク質を分解し続ける必要がある.この際,常に完璧なタンパク質のみが生産されるわけではなく,30%程度のタンパク質が正しく生産されない34).そのため,ユビキチンプロテアソーム系(UPS)がタンパク質の品質管理を行っている.UPSは,細胞内でミスフォールドしたタンパク質にユビキチン鎖を付加し,このユビキチン鎖をプロテアソームが認識し分解するシステムである35).しかしながら,ハンチントン病やアルツハイマー病などヒトの神経変異疾患が生じている脳には,高度にユビキチン化された凝集タンパク質が蓄積しており,何らかの原因でUPSによる分解を逃れたものが蓄積していることが想定されている36).これらの異常に蓄積し,毒性化したタンパク質を処理する細胞内機能として,アグリソーム–オートファジー経路による分解メカニズムが報告されている.この経路では,細胞において毒性を示すタンパク質凝集体の隔離と分解のために,アグリソームと呼ばれる構造物を形成する37).近年,アグリソームの形成にはHDAC6–ユビキチン–プロテアソームを介した経路38, 39)やユビキチン化を必要としないBag3–オートファジー経路40),また,これらに依存しない経路41)が存在すると考えられている.液胞への輸送も関連してくるBag3–オートファジー経路では,①シャペロンであるHsp70, BAG3複合体がタンパク質凝集体と結合する,②14-3-3,ダイニン複合体とリン酸化されたBAG3が結合する,③形成された複合体が微小管依存的に核周辺区画に凝集したタンパク質を収集する,④アグリソームがオートファゴソーム膜に包まれる,⑤オートファゴソームとリソソームが融合し分解される,という一連の流れで毒性のタンパク質凝集体を分解している(図4B).これらのタンパク質のうち,Hsp70と14-3-3の分裂酵母ホモログはそれぞれ,Ssa1, Ssa12, Ssc1とRad24, Rad25としてデータベース上で表示されるが,BAG3に関しては該当するものがなかった.そこで,アグリソーム形成に必須であるとされる14-3-3の分裂酵母ホモログRad24の遺伝子破壊株rad24Δを用い解析を行ったところ,野生株にてGmp1を過剰発現した際と比べて有意に凝集率が減少した.この結果よりGmp過剰発現による凝集体はアグリソームと類似した機構で形成されることが推察される.
Gmpの本来の役割は分泌経路のゴルジ体膜などの表層で輸送小胞や槽板維持をサポートすることであると考えられるが,過剰発現によりみられる表現型は,分裂酵母がアグリソーム形成機構を解析するモデルとしても利用できることを示唆するものである.
4. 分裂酵母のゴルジ体を足場として機能するロンボイドプロテアーゼの謎
タンパク質分解酵素(プロテアーゼ)は,タンパク質のペプチド結合の加水分解を触媒する酵素の総称である.一般に,プロテアーゼによるペプチド結合の加水分解は,細胞質などの溶質(液体)中で起こると考えられていたが,疎水性の脂質二重層内においてペプチド結合を加水分解するプロテアーゼが存在することが明らかになってきた.この膜内の切断に関わるプロテアーゼは,膜内プロテアーゼ(intramembrane-cleaving proteases:I-CLiPs)と呼ばれ,基質となる膜タンパク質を膜の内部で切断し,細胞間のシグナル伝達や転写因子の遊離など,さまざまな生体内プロセスに関わることで注目されている42).現在,膜内プロテアーゼは,以下の四つのファミリーに分類されている.セクレターゼ[アルツハイマー病の原因タンパク質であるアミロイド前駆体タンパク質(APP)の膜貫通領域の近傍および膜内の切断を行う酵素群の総称]のうち,アスパラギン酸プロテアーゼであるγセクレターゼ,同じくアスパラギン酸プロテアーゼであるシグナルペプチドペプチダーゼ,亜鉛メタロプロテアーゼであるSite-2プロテアーゼ,セリンプロテアーゼであるロンボイドプロテアーゼ(以下,ロンボイド)である43).
これらの中で,ロンボイドはショウジョウバエのロンボイド(Rhomboid-1)の研究から見いだされた膜タンパク質ファミリーであり,原核生物からヒトを含む高等真核生物まで,ゲノムが解読されたほぼすべての生物に広く存在している.ロンボイド(rhomboid)は,偏菱形という意味で,ショウジョウバエのRhomboid-1に変異が生じた幼虫は,頭部骨格が偏菱形になるという表現型が確認されていた.その後,Rhomboid-1が新規の膜内プロテアーゼであることがわかり,その表現型が由来となって,ロンボイドプロテアーゼと名づけられた44).Rhomboid-1は,Spitz, Gurken, Kerenといった膜結合型EGF受容体リガンドの切断による活性化を通じて翅・眼・生殖細胞の発生・分化に関与している.たとえば,翅形成に関わるEGF受容体リガンドであるSpitzは,小胞体(ER)で膜結合型として合成されるが,エスコートタンパク質であるStarの働きでゴルジ体へ移行した後,そこに局在するRhomboid-1によって切断される.その結果,Spitzの内腔ドメインが膜から遊離し,細胞外へ分泌されることで,活性型のEGF受容体リガンドとして機能する.
ロンボイドの大きな特徴としては,複数回膜貫通タンパク質(6~7回膜貫通タンパク質)で,活性中心領域が脂質二重層の内部に埋め込まれた構造をとっており,基質となる膜タンパク質の膜貫通領域および近傍を切断するということがあげられる.ロンボイドの研究は活発に行われており,現在までに出芽酵母Saccharomyces cerevisiaeでのミトコンドリア融合の制御,Providencia stuartiiでのクオラムセンシングへの関与,寄生虫(マラリア原虫,トキソプラズマ原虫)の宿主細胞の侵入時に切断される接着因子の遊離など,機能や局在は生物種によって異なることがわかっている45, 46).
哺乳類でこれまでに明らかになっているロンボイドの種類や関連する疾病は,Freemanらによってまとめられている47).哺乳類では,ロンボイド様プロテアーゼも含めると14種類が存在するが,関与する機能がわかりつつあるのは3種類である.たとえば,パーキンソン病や糖尿病,創傷治癒,がん化など,ヒトの重篤な疾病と関連していることがわかってきているが,細胞内局在と機能の連関や活性化機構の詳しい解析は進んでいない.
2006年にI-CLiPsとして初めて大腸菌のロンボイドであるGlpGのX線結晶構造が報告されており,ロンボイドの触媒残基は,多くのセリンプロテアーゼが持つcatalytic triad(Ser–His–Asp)ではなく,catalytic dyad(Ser–His)からなることが明らかになった48).さらに,一般にロンボイドは,I型1回膜貫通タンパク質(Nout–Cin)を切断すると考えられてきたが,近年,II型1回膜貫通タンパク質(Nin–Cout)49)や,複数回膜貫通タンパク質50)を切断することも報告されており,ロンボイドの機能および基質は多岐にわたるということができる.
現在でもロンボイドの研究は活発に行われているが,その中でもロンボイドの研究における大きな進展は,2010年にロンボイドの基質となるタンパク質には,ロンボイドによって認識され,切断されるためのモチーフ(基質認識モチーフ)が存在することが明らかになったことである51).この認識モチーフが発見されるまでは,ロンボイドの基質にはαヘリックスを不安定化させる残基を有するという共通点はあったものの,ロンボイドによる基質切断機構についての統一的な見解は得られていなかった.認識モチーフが存在しても,その切断効率は38%ではあるが,現在もこのモチーフを有する基質が発見されており,今後もさらに多くの基質が発見され,新たな機能の解明につながることが期待される.また,ロンボイドと基質の親和性は低く,基質の切断はゆっくり起こることから,動力学的に制御されている反応であることが報告されている52).この性質は,他のプロテアーゼや膜タンパク質とは異なり,DNA修復酵素による反応に類似しており,β-ラクタムやβ-ラクトンがロンボイドを特異的に阻害するという報告もなされている53)ことから,新たな阻害剤の開発および,ロンボイドが関与している疾病の新薬の開発につながることも期待されている.
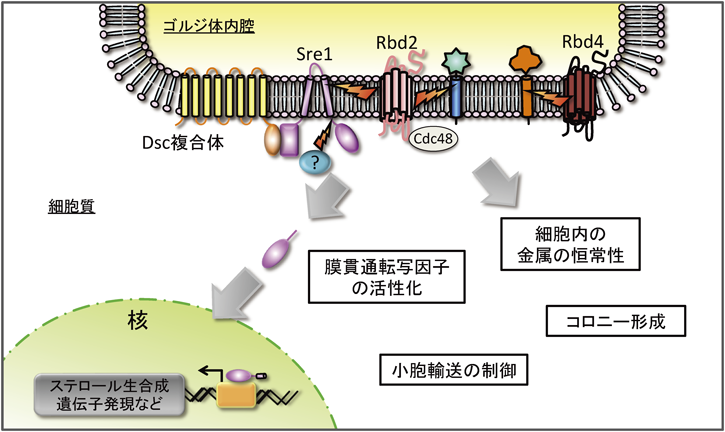
分裂酵母のゲノムデータベースには,ロンボイドと相同性の高い遺伝子が四つ(Rbd2, Rbd4, Rbd1, Rbd3)存在している.このうち,二つ(Rbd1, Rbd3)はミトコンドリア局在,二つ(Rbd2, Rbd4)がゴルジ体局在である.この二つのロンボイドは,一体何をゴルジ体膜で切断しているのだろうか?
最近,EspenshadeのグループがRbd2に関して一端を明らかにしており54),SREBP(sterol regulatory element binding protein)という膜結合型転写因子ファミリーに属するタンパク質の切断に関与していることがわかった(図5).SREBPは,出芽酵母S. cerevisiaeには存在しないが,多くの真菌類や哺乳類に存在し,主に脂質の恒常性をコントロールしている粗面小胞体を2回膜貫通しているタンパク質である.約30アミノ酸の内腔ループに分離されてN末端とC末端が細胞質に位置し,C末端領域に切断を活性化させるタンパク質(SREBP cleavage-activating protein:SCAP)と結合する領域があり,N末端領域には転写因子として機能する領域を有している.SCAPは,ER膜貫通タンパク質であり,ステロールを感知する領域(sterol sensing domain:SSD)を有している.
哺乳類におけるSREBP経路55)では,SCAPはmTORC1の下流に位置し,ステロールリッチの場合,SCAPがER常在性タンパク質であるINSIGとSREBPと三量体を形成することで,SREBP-SCAP複合体がERからゴルジ体へ移行せず,結果的にSREBPは不活性なままである.しかし,ステロールが不足すると,三量体形成が抑制され,SREBP–SCAP複合体はCOPII輸送小胞を通じてゴルジ体へ輸送される.そこで,ゴルジ体常在性の2種類の膜内プロテアーゼ(site-1 protease:S1P, site-2 protease:S2P)によって,ゴルジ体内腔側をS1P,細胞質側をS2Pに切断されることで,可溶性となったSREBPのN末端領域が核へ移行し,転写因子として機能することが知られている.また,このとき,INSIGの一つであるINSIG-1は,膜結合型E3ユビキチンリガーゼであるgp78と相互作用し,ユビキチン化され,プロテアソームによって分解される.
一方,分裂酵母のSre1は低酸素状態時にSre1–Scp1複合体がERからゴルジ体へ移行する.分裂酵母にはS1PとS2Pが存在しないため,その代わりに,ロンボイドプロテアーゼRbd2がゴルジ体内腔側のSre1を切断し,続いてSre1の細胞質側は,まだ未知のプロテアーゼによって切断されていると考えられている.切断されたSre1のN末端は核へ移行し,そこで転写因子としてステロール生合成遺伝子の発現制御を行うとともに,自身のプロモーターにも結合することで発現を上昇させる.
Rbd2はロンボイドに保存されているセリンの活性中心や,基質切断中の基質中間体であるオキシアニオン中間体の安定性に関わるHxxxxHxxxNモチーフ(x:任意のアミノ酸),膜貫通領域の二量体化に必要なGxxxGモチーフ,活性中心のセリンと水素結合をなしているヒスチジンについても機能に必須であることから,分裂酵母のロンボイドプロテアーゼとして間違いないと考えられる.しかしながら,大腸菌を宿主とした実験系で,最もよくロンボイドの基質として使われているTatA(細菌Providencia stuartii由来のType I膜タンパク質)とRbd2を発現させると切断が確認されるが,Sre1とRbd2を発現させたときには切断がみられない.また,TatAの切断箇所の解析から,これまでのロンボイドの基質認識配列と異なっていることが示唆されているが,この配列がSre1にないことから,ゴルジ体内腔で切断されている正確な箇所は不明である.Rbd2の活性制御に関しては不明な点が多いものの,ゴルジ体のE3ユビキチンリガーゼ複合体(Dsc1~5)がRbd2によって切断されなかった基質やDsc複合体そのもののユビキチン化に関与し,分解や局在化の決定56)にも関与していることが考えられている.また,AAAファミリーATPaseであるCDC48は,必須ではないもののRbd2とSre1のアダプターとして機能し54),CDC48の補因子Ufd1はDsc複合体の機能制御を介してRbd2の機能を制御している57)ことが考えられている.いずれにしても,分裂酵母のロンボイドは複雑な分子メカニズムによって制御されていることが示唆される.幅広い生物種に存在するにもかかわらず,その基質や機能解析が遅れているのは,各生物種で適した活性測定法を確立する必要があるためと考えられており58),基質の改変も含めて,分裂酵母内での切断箇所および活性測定が行える実験系の確立が必要である.
分裂酵母のゴルジ体にはRbd2以外にRbd4がある.筆者らは,遺伝子破壊株の表現型がRbd2やDsc複合体,CDC48等とは独立していることを確認しており,新たな基質と活性制御の理解が進むことを期待している(図5).
引用文献References
1) Barnett, J.A., Payne, R.W., & Yarrow, D. (1990) in Yeasts: Characteristics and Identification, 2nd Ed., pp. 1–1002, Cambridge University Press.
2) Barnett, J.A., Payne, R.W., & Yarrow, D. (2001) in Yeasts: Characteristics and Identification, 3rd Ed., pp. 1–1139, Cambridge University Press.
3) Helston, R.M., Box, J.A., Tang, W., & Baumann, P. (2010) FEMS Yeast Res., 10, 779–786.
4) Toyama, R. & Okayama, H. (1990) FEBS Lett., 268, 217–221.
5) Lee, M.G. & Nurse, P. (1987) Nature, 327, 31–35.
6) Kaufer, N.F., Simanis, V., & Nurse, P. (1985) Nature, 318, 78–80.
7) Hayles, J. & Nurse, P. (1992) Annu. Rev. Genet., 26, 373–402.
8) Giga-Hama, Y. & Kumagai, H. (1999) Biotechnol. Appl. Biochem., 30, 235–244.
9) Takegawa, K., Tohda, H., Sasaki, M., Idiris, A., Ohashi, T., Mukaiyama, H., Giga-Hama, Y., & Kumagai, H. (2009) Biotechnol. Appl. Biochem., 53, 227–235.
10) Wang, Z. (2017) Curr. Genet., 63, 427–433.
11) Wood, V., Harris, M.A., McDowall, M.D., Rutherford, K., Vaughan, B.W., Staines, D.M., Aslett, M., Lock, A., Bahler, J., Kersey, P.J., & Oliver, S.G. (2012) Nucleic Acids Res., 40(D1), D695–D699.
12) Wei, J.H. & Seemann, J. (2017) Curr. Opin. Cell Biol., 47, 43–51.
13) Xiang, Y. & Wang, Y. (2011) Cell Tissue Res., 344, 365–379.
14) Huang, S., Tang, D., & Wang, Y. (2016) Dev. Cell, 38, 73–85.
15) Preuss, D., Mulholland, J., Franzusoff, A., Segev, N., & Botstein, D. (1992) Mol. Biol. Cell, 3, 789–803.
16) Robinson, D.G., Brandizzi, F., Hawes, C., & Nakano, A. (2015) Plant Physiol., 168, 393–406.
17) Ishii, M., Suda, Y., Kurokawa, K., & Nakano, A. (2016) J. Cell Sci., 129, 3251–3261.
18) Suda, Y. & Nakano, A. (2012) Traffic, 13, 505–510.
19) Ayscough, K., Hajibagheri, N.M., Watson, R., & Warren, G. (1993) J. Cell Sci., 106, 1227–1237.
20) Sztul, E. & Lupashin, V. (2006) Am. J. Physiol. Cell Physiol., 290, C11–C26.
21) Munro, S. & Nichols, B.J. (1999) Curr. Biol., 9, 377–380.
22) Miyatake, M., Kuno, T., Kita, A., Katsura, K., Takegawa, K., Uno, S., Nabata, T., & Sugiura, R. (2007) Genetics, 175, 1695–1705.
23) Tanaka, N. & Takegawa, K. (2001) Trends Glycosci. Glycotechnol., 13, 519–532.
24) Ohashi, T., Fujiyama, K., & Takegawa, K. (2012) J. Biol. Chem., 287, 38866–38875.
25) Suzuki, S., Matsuzawa, T., Nukigi, Y., Takegawa, K., & Tanaka, N. (2010) Microbiology, 156, 708–718.
26) Matsuzawa, T., Morita, T., Tanaka, N., Tohda, H., & Takegawa, K. (2011) Mol. Microbiol., 82, 1531–1544.
27) Andreishcheva, E.N., Kunkel, J.P., Gemmill, T.R., & Trimble, R.B. (2004) J. Biol. Chem., 279, 35644–35655.
28) Higuchi, Y., Yoshinaga, S., Yoritsune, K., Tateno, H., Hirabayashi, J., Nakakita, S., Kanekiyo, M., Kakuta, Y., & Takegawa, K. (2016) Sci. Rep., 6, 26349.
29) Conrad, M., Schothorst, J., Kankipati, H.N., Van Zeebroeck, G., Rubio-Texeira, M., & Thevelein, J.M. (2014) FEMS Microbiol. Rev., 38, 254–299.
30) Milias-Argeitis, A., Oliveira, A.P., Gerosa, L., Falter, L., Sauer, U., & Lygeros, J. (2016) PLOS Comput. Biol., 12, e1004784.
31) Tesniere, C., Brice, C., & Blondin, B. (2015) Appl. Microbiol. Biotechnol., 99, 7025–7034.
32) Kayikci, O. & Nielsen, J. (2015) FEMS Yeast Res., 15, 1–8.
33) Matsuzawa, T., Fujita, Y., Tohda, H., & Takegawa, K. (2012) Eukaryot. Cell, 11, 159–167.
34) Schubert, U., Anton, L.C., Gibbs, J., Norbury, C.C., Yewdell, J.W., & Bennink, J.R. (2000) Nature, 404, 770–774.
35) Glickman, M.H. & Ciechanover, A. (2002) Physiol. Rev., 82, 373–428.
36) Gatchel, J.R. & Zoghbi, H.Y. (2005) Nat. Rev. Genet., 6, 743–755.
37) Johnston, J.A., Ward, C.L., & Kopito, R.R. (1998) J. Cell Biol., 143, 1883–1898.
38) Dompierre, J.P., Godin, J.D., Charrin, B.C., Cordelieres, F.P., King, S.J., Humbert, S., & Saudou, F. (2007) J. Neurosci., 27, 3571–3583.
39) Kawaguchi, Y., Kovacs, J.J., McLaurin, A., Vance, J.M., Ito, A., & Yao, T.P. (2003) Cell, 115, 727–738.
40) Jia, B., Wu, Y., & Zhou, Y. (2014) Prion, 8, 173–177.
41) Munoz-Moreno, R., Barrado-Gil, L., Galindo, I., & Alonso, C. (2015) Viruses, 7, 1823–1831.
42) Wolfe, M.S. & Kopan, R. (2004) Science, 305, 1119–1123.
43) Freeman, M. (2014) Annu. Rev. Cell Dev. Biol., 30, 235–254.
44) Mayer, U. & Nusslein-Volhard, C. (1988) Genes Dev., 2, 1496–1511.
45) Sibley, L.D. (2013) Biochim. Biophys. Acta, 1828, 2908–2915.
46) Adam, Z. (2013) Biochim. Biophys. Acta, 1828, 2933–2936.
47) Dusterhoft, S., Kunzel, U., & Freeman, M. (2017) Biochim. Biophys. Acta, 1864(11 Pt B), 2200–2209.
48) Maegawa, S., Ito, K., & Akiyama, Y. (2005) Biochemistry, 44, 13543–13552.
49) Herlan, M., Bornhovd, C., Hell, K., Neupert, W., & Reichert, A.S. (2004) J. Cell Biol., 165, 167–173.
50) Wan, C., Fu, J., Wang, Y., Miao, S., Song, W., & Wang, L. (2012) PLoS One, 7, e37452.
51) Strisovsky, K., Sharpe, H.J., & Freeman, M. (2009) Mol. Cell, 36, 1048–1059.
52) Dickey, S.W., Baker, R.P., Cho, S., & Urban, S. (2013) Cell, 155, 1270–1281.
53) Pierrat, O.A., Strisovsky, K., Christova, Y., Large, J., Ansell, K., Bouloc, N., Smiljanic, E., & Freeman, M. (2011) ACS Chem. Biol., 6, 325–335.
54) Hwang, J., Ribbens, D., Raychaudhuri, S., Cairns, L., Gu, H., Frost, A., Urban, S., & Espenshade, P.J. (2016) EMBO J., 35, 2332–2349.
55) Ricoult, S.J. & Manning, B.D. (2013) EMBO Rep., 14, 242–251.
56) Raychaudhuri, S. & Espenshade, P.J. (2015) J. Biol. Chem., 290, 14430–14440.
57) Burr, R., Ribbens, D., Raychaudhuri, S., Stewart, E.V., Ho, J., & Espenshade, P.J. (2017) J. Biol. Chem., 292, 16333–16350.
58) Arutyunova, E., Strisovsky, K., & Lemieux, M.J. (2017) Methods Enzymol., 584, 395–437.
59) Turi, T.G., Webster, P., & Rose, J.K. (1994) J. Biol. Chem., 269, 24229–24236.
60) Appenzeller-Herzog, C. & Hauri, H.P. (2006) J. Cell Sci., 119, 2173–2183.
61) Venkatagopalan, P., Daskalova, S.M., Lopez, L.A., Dolezal, K.A., & Hogue, B.G. (2015) Virology, 478, 75–85.