各々の生物種は固有の遺伝子を持ち,その遺伝子の情報に従って自らの身体を形作り,また,生命活動を営んでいる.この図式の中心には,いわゆるセントラルドグマと呼ばれる一連の過程があり,DNA−RNA−タンパク質という順番に情報が分子としてその形態を変えながら伝達される.端的に表現すれば,生命活動はタンパク質が触媒する多種多様な生化学的反応の集合体であり,個々に見れば個性豊かなタンパク質の活動が全体としては秩序をもって協調することで一つの生命を形作っている.生命活動の主要な担い手であるタンパク質は,DNA(直接的にはmRNA)の塩基配列によって指示された順番でアミノ酸が連結したひも状分子として誕生するが,合成が完了し,構造形成がなされた後に初めて生命活動に参加することができると考えられてきた.ところが,近年,合成の途上で働くユニークな因子がいくつか発見された.これらは,自身の翻訳を一時停止(アレスト)する性質を共有しつつ,多様な生理機能を発揮する.その性質から「翻訳アレストペプチド」や,単に「アレストペプチド」(英語では,regulatory arrest peptideやregulatory nascent chainなど)と呼ばれる1–3).本稿では,まず,代表的なアレストペプチドを紹介し,その後,一連の研究を通じてみえてきたアレストペプチドの共通性と多様性について論じる.また,アレストペプチド研究がもたらした生物学的な意義やインパクト,アレストペプチド研究の今後の展望とその潜在的可能性についても議論する.
1)SecMは制御された翻訳アレストを利用してタンパク質の分泌活性をモニターする
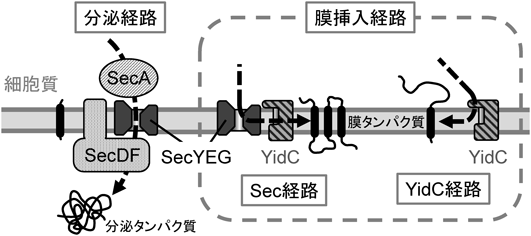
細胞質で合成されたタンパク質が細胞外へと輸送される経路,すなわち,タンパク質の分泌経路は,あらゆる生物にとって必須である.大腸菌で見いだされたアレストペプチドであるSecMは,細胞の持つタンパク質の分泌活性をモニターし,その活性が不足した状況において分泌駆動モーターであるSecA ATPaseの発現を誘導することで細胞の持つ分泌能を一定以上に維持する.SecMについて詳述する前に,真正細菌のタンパク質分泌経路について概説する.
分泌タンパク質は,N末端に疎水性アミノ酸残基に富んだ「シグナル配列」を持った前駆体として合成される.大腸菌における主要なタンパク質分泌経路はSecY, SecE, SecGの三つの膜タンパク質からなるSecトランスロコンを介したSec経路である(図1)4).Sec経路において,可溶性タンパク質であるSecA ATPaseは分泌タンパク質を細胞内から外へと押し出すモーターとして働く.一方,膜タンパク質であるSecDとSecFはヘテロ二量体を形成し,その大きな細胞外ドメインを介して基質分泌タンパク質を細胞外側から引っぱることで分泌を促進するモデルが提唱されている5, 6).
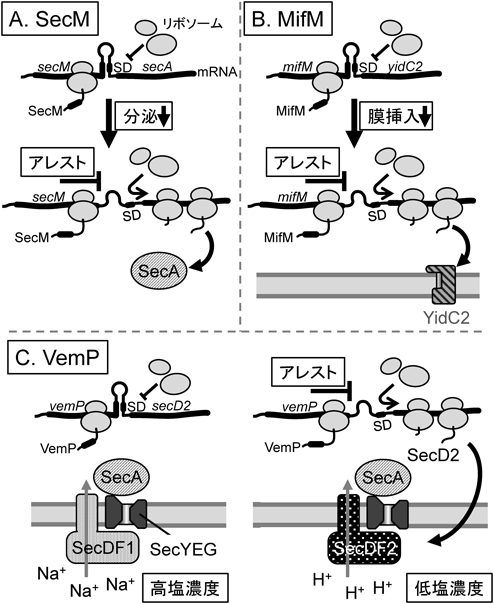
伊藤らは,彼らがsecMと命名したORFが,同じオペロンの下流にあるSecAのフィードバック制御に重要な役割を果たすことを示した(図2A)7).真正細菌における翻訳の開始には開始コドン上流のShine-Dalgarno(SD)配列をリボソームが認識することが必要であるが,secM-secAのmRNAは,secAのSD配列を含むかたちでステムループ構造を形成し,その結果,secAの翻訳開始は阻害される.一方,上流のSecMのコード領域がこのステムループを形成する配列の一部とオーバーラップしているため,secMを翻訳するリボソームがこの領域に存在している間はステムループが開裂する.さらにSecMは,自身の翻訳伸長を終止コドンの少し手前でアレストするというユニークな性質を持つ7).つまり,この翻訳伸長アレストは,リボソームがsecM mRNA上にとどまりステムループ構造の形成を阻害し,secAのSD配列を別のリボソームが認識できる状態に維持する働きを持つ.その結果,SecAの翻訳開始が支障なく起こることになる.
SecMはN末端にシグナル配列を持つ分泌タンパク質であり,Sec経路で分泌される.興味深いことに,SecM合成途上鎖の膜透過反応が引き金となり翻訳アレストが解除される.それにより翻訳を再開したリボソームはsecMの残りの数コドンを翻訳したのち終止コドンに到達し,翻訳終結因子の働きにより速やかにmRNAから解離する.つまり,細胞の分泌活性が高いときには,SecMの翻訳アレストに依存するSecAの合成はごく短時間しか起こらない.逆に,分泌活性が不足している状況下では,SecAの合成が持続する.特筆すべきは,SecMが,自身の翻訳伸長をアレストする能力を持つこと,そしてそのアレストの持続時間がSec分泌装置の活性に逆相関することである.このようにSecMは,翻訳途上鎖の状態で細胞の分泌活性をモニターするという点で,一般的なタンパク質と機能発現のあり方が大きく異なる.従来,翻訳途上の新生鎖は機能を持たない合成中間体とみなされ,機能研究の対象とはされてこなかったが,SecMによる翻訳アレストの発見は,細胞機能を調節する翻訳途上鎖(regulatory nascent chain)というユニークな生体機能分子のあり方を明らかにした.この発見は,やがて,翻訳途上鎖が主役を演じる生命現象を対象とした研究,いわば,nascent chain biology(新生鎖の生物学)ともいうべき新たな研究領域の創成へとつながる最初のきっかけであったように思われる.
2)新生ポリペプチド鎖はリボソーム成分との相互作用を介して自身の翻訳速度を調節する
SecMはどのような機構で自身の翻訳伸長をアレストするのであろうか.リボソームは大小二つのサブユニットからなる.翻訳が開始されるとリボソームはmRNA上を移動しながら,小サブユニットにある暗号解読センター(デコーディングセンター)でmRNAに転写された遺伝情報を解読する.大サブユニットには活性中心であるペプチジル転移酵素中心(PTC)があり,ここで遺伝情報に従ってアミノ酸が一つずつ連結されることでペプチド鎖が伸長する.具体的には,Aサイトに新たなアミノアシルtRNAが受容されると,PサイトのペプチジルtRNAから合成途上のペプチド鎖がAサイトのアミノアシルtRNAに転移するペプチジル転移反応が起こる.次いで,リボソームがmRNA上を1コドン分移動する転位反応(translocation)が起こる.この繰り返しによって伸長するポリペプチド鎖は,リボソームの大サブユニットにあるペプチド鎖排出トンネルを通ってリボソーム外へと排出される.このリボソームのトンネルは,およそ100 Åの長さであり,30から40アミノ酸残基の長さのポリペプチド鎖を収容できる8, 9).トンネルをPTCから出口方向に3分の1ほど進んだ領域には狭窄部位が存在するが,この部位は真正細菌ではリボソームタンパク質L4, L22とリボソームRNA成分によって形成されている.このトンネルの内部は,狭窄部位以外であれば,少なくともαヘリックス構造の形成に十分な幅があるが,大きなドメインへの高次構造の形成を許すほどのスペースは存在しない10–12).
リボソームのペプチド鎖排出トンネルは,新規合成されたポリペプチド鎖が初めて出会う環境である.リボソーム大サブユニットの高解像度での結晶構造を初めて解いたSteitzらは,このトンネル内壁を「テフロンのような」と形容し,トンネルは新生ポリペプチド鎖と相互作用をしないような環境になっていると論じた8).ところが,SecMの翻訳アレストは,SecMのC末端付近に存在する特定のアミノ酸配列が,自身を翻訳しているリボソームのペプチド鎖排出トンネルやPTC付近でリボソーム成分と相互作用することで引き起こされることが示された13).つまり,これらの結果は,リボソームのペプチド鎖排出トンネルが単なる物理的なトンネルであるだけでなく,新生ポリペプチド鎖と特異的に相互作用し,翻訳制御に積極的に関与しうることを意味している.また,タンパク質のアミノ酸配列が,そのタンパク質の立体構造や細胞内局在に関する情報以外に,自身の翻訳伸長速度を決定する情報を内包しうることを示している.後に,さまざまなアレストペプチドについてその分子機構の解析がなされ,PTCやトンネル成分が,さまざまな翻訳アレストにおいて普遍的に重要であることが明らかにされた1, 2).すなわち,新生鎖のアミノ酸配列が自身を合成する速度に関する情報を含むこと,また,リボソームトンネルやPTCがその情報を読み取り合成速度に反映するという図式は,SecMに限定されたものではなく,アレストペプチド全般に共通してみられるものであり,さらには,一般的なタンパク質合成の過程においても同様の制御が行われている可能性が考えられる.SecMの翻訳アレスト機構に関する研究から,新生ポリペプチド鎖による自律的な翻訳制御という新たな概念が生まれた.
1)MifMはYidC依存的なタンパク質膜組込み経路をモニターする
筆者らが枯草菌より見いだしたMifMは,YidC依存的なタンパク質の膜組込み経路をモニターする翻訳アレストペプチドである14, 15).YidC経路をモニターする翻訳アレストペプチドとしては初めての,かつ,現時点では唯一の報告例である.
細菌の膜タンパク質の膜への組込み経路は複数知られているが,主要な経路は分泌経路と同様にSecトランスロコンを介したものである.一方,Sec非依存的なタンパク質膜挿入経路として,YidC経路が知られている(図1)16).Sec経路では,SecYEG複合体が形成するチャネル構造を介してタンパク質が膜挿入されるが,YidCは,構造解析の結果などから,「非チャネル型」タンパク質膜組込み装置であり,Secチャネルとは大きく異なる機構でタンパク質を膜挿入していることが示唆された17–19).しかしながら,その具体的な分子メカニズムはいまだ明らかにされていない.
枯草菌にはYidCのホモログが二つ存在するが,第一のホモログであるSpoIIIJのみが恒常的に発現しており,第二のホモログYidC2(YqjG)は,SpoIIIJの活性が低下したときにのみ,その発現が誘導される20).このフィードバック機構の鍵を握る因子が,筆者らがMifMと命名したアレストペプチドである.その全体像は,大腸菌SecMによるSecAの発現制御と概念的に類似している(図2B).mifMとyidC2はオペロンを形成する.mifM-yidC2 mRNAは,yidC2のSD配列を内包する形でステムループ構造を形成するような配列を持つ.そのため,このステムループの形成はyidC2の翻訳開始を阻害する.一方,yidC2の上流にはmifMがあり,そのコード領域の3′末端付近はステムループ構造の内部にまで至る.この領域でmifMの翻訳がアレストすることでyidC2のSD配列の露出が維持されYidC2の合成が誘導される.一方,MifMはN末端に膜貫通領域を持ち,YidC依存的に膜挿入される.YidCによる膜挿入と共役してMifMの翻訳アレストは解除される.そのため,SpoIIIJの活性が十分な条件下ではMifMの翻訳アレストは速やかに解除され,YidC2の合成は抑制されるが,SpoIIIJの活性が不足している条件下では,MifMの翻訳アレストが長時間解除されず,その間YidC2の合成が持続する.枯草菌は,このような仕組みで,生育に必須なYidCの活性を常に一定以上に維持しているものと思われる14, 15).
2)MifMはアレストを小刻みに繰り返すユニークなアレストペプチドである
MifMの研究からは,翻訳アレスト様式の驚くべき多様性がみえてきた.新生ポリペプチド鎖はリボソームのトンネルを出口方向に向かって一方向に移動し続ける.そのため,トンネルやPTC成分とアレストモチーフとが特異的に相互作用する機会は1本のペプチド鎖合成につき一度きりしか訪れない.その制約を反映してか,アレストペプチドによる翻訳アレストは,多くの場合,翻訳伸長の途上で一度だけ,ある特定のコドンでのみ起こる.たとえば,SecMの場合,必ず165番目のグリシンコドンがPサイトに位置した状態で,また,後述する海洋性ビブリオ菌由来のVemPの場合は,156番目のグルタミンコドンがPサイトに来たときに,それぞれリボソームを停止させる21, 22).ところが,枯草菌mifMは,一度のMifM合成につき複数回アレストを起こすmultisite stallingという様式で翻訳伸長を遅延させる23).具体的には,主に89番目,90番目,91番目,92番目の四つのコドンがそれぞれリボソームのPサイトに位置するたびにアレストが起こる.下流遺伝子yidC2の発現量はリボソームのmRNA上での停滞時間の長さに相関するが,MifMは,1か所で強固な翻訳アレストを起こす代わりに,比較的弱い翻訳アレストを繰り返し起こすことで,下流遺伝子の発現に必要な長さのリボソーム停滞時間をトータルで稼いでいるものと考えられる.アレストペプチドの作用機序の進化の驚くべき柔軟性を示す例である.
では,なぜMifMはこのようなmultisite stallingを起こすことができるのであろうか.その分子機構の全貌はいまだ明らかにされていないが,変異解析や構造解析からその手がかりがいくつか得られている.MifMのmultisite stallingにおいては,MifMのアレストモチーフ内の一番C末端にある連続した負電荷アミノ酸残基(86DEED89)がPTC近傍で重要な役割を果たすことが示唆された23).このC末端の負電荷アミノ酸残基のクラスターは他のMifMホモログにも保存されている.さまざまな変異解析から,この領域にある酸性残基はいずれも翻訳アレストを起こすための相互作用に個別に寄与していることが示唆されており23),弱い相互作用が繰り返し出現することで,複数回翻訳アレストを実行することができるのであろう.
4. 海洋性ビブリオ菌VemPは,環境に応答して分泌装置をリモデリングする
最近,森らが見いだした海洋性ビブリオ菌由来の翻訳アレスト因子VemPは,タンパク質分泌をモニターする翻訳アレスト因子としてはSecMに次ぐ二番目の報告例である.SecMがSecAの発現を制御するのに対し,VemPは,もう一つの分泌駆動因子であるSecDF2複合体の発現を調節する22).また,後述のとおり,VemPは,SecDF2の発現調節を介して海洋性ビブリオ菌がさまざまな食塩濃度の環境に適応するために重要であることが示されている.
真正細菌のタンパク質分泌では,すでに述べたように,細胞質側にあるSecAが分泌駆動モーターとして働く一方,SecDF複合体は,大きな細胞外ドメインを介して分泌タンパク質を補足し,その構造変化を起こすことにより基質を細胞外へ引っぱり出すと考えられている(図1).その構造変化は大腸菌の場合,プロトンの細胞内への流入と共役して起こると提唱されている5, 6).
海洋性ビブリオ菌の一種であるVibrio alginolyticusは2組のSecDF複合体を持つ.興味深いことに,第一のSecDF複合体であるSecDF1は,プロトンではなくナトリウムイオンの流入を駆動力にする「ナトリウムイオン駆動型」SecDFであり,第二の複合体であるSecDF2は,大腸菌SecDFと同様に「プロトン駆動型」のSecDFである5, 22).高食塩濃度環境下では,SecDF1がナトリウムイオンを駆動力にしてタンパク質分泌を促進するが,環境中のナトリウムイオンが低下することでSecDF1が機能しなくなると,プロトン駆動型のSecDF2の発現が誘導され,プロトン駆動力を利用したタンパク質分泌が行われる.これは,おそらく,ナトリウムイオンに富んだ海水中と,比較的塩濃度の低い環境(たとえば,汽水域や感染した宿主細胞の体内など)との両方の環境に海洋性ビブリオ菌が適応するための巧妙な仕組みであると考えられる.
SecDF2の発現はVemPの翻訳アレストを介した機構で調節されている(図2C)22).その全体像はやはりSecMと概念的に類似している.vemP, secD2, secF2はオペロンを形成しており,そのmRNAはsecD2の手前でステムループ構造を形成し,その結果としてsecD2のSD配列がマスクされる.secD2とその下流のsecF2の翻訳はおそらく共役しており,secD2のSD配列が二次構造でマスクされると,secD2, secF2両者の翻訳が同時に抑制される.VemPが翻訳伸長アレストを起こすと,mRNA上でリボソームが停滞し,このmRNAの二次構造形成を妨げる.一方,VemPは,N末端にシグナル配列を持つ分泌タンパク質であり,Sec経路依存的なVemPの分泌と共役して翻訳アレストが解除される.したがって,Sec依存的なタンパク質分泌活性の活性が低下するとVemPの翻訳アレストが持続し,結果としてSecDF2が合成される.自然界では,このビブリオ菌が塩濃度の低い環境に移行したときにこの調節が起こると考えられる.すなわち,塩濃度の低下はナトリウムイオン駆動型であるSecDF1の機能低下をもたらし,その結果,分泌装置の活性が低下する.すると,VemPの翻訳アレストが持続することで,プロトン駆動型であるSecDF2の発現が誘導される.海洋性ビブリオ菌は,このような原理で,タンパク質分泌装置をその環境に最適化するようにカスタマイズしているものと思われる.同じSecモニター系であっても,SecMが単純に一つの分泌駆動因子の量を制御するのに対し,VemPは,二つの補助因子を入れ替えて分泌装置に質的変化をもたらす.
5. タンパク質の局在化装置を制御するアレストペプチドの共通性と多様性
1)多様な翻訳アレスト機構から類似の制御システムが生み出された
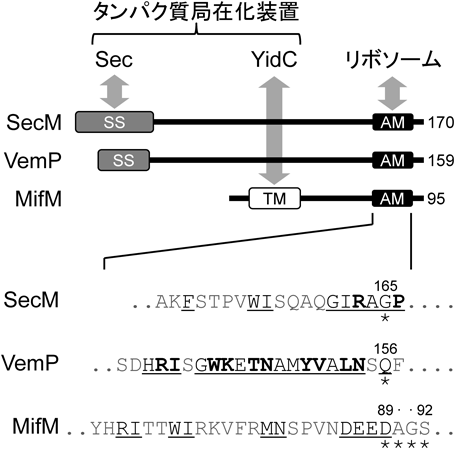
タンパク質の局在化装置をモニターするSecM, MifM, VemPは,いずれもN末端にタンパク質局在化シグナル(SecMとVemPは分泌シグナル,MifMは膜挿入シグナル)を持ち,Sec分泌経路やYidC膜挿入経路の基質としてそれぞれ認識される(図3).基質としてタンパク質の局在化装置の挙動を監視することから,モニター基質(monitoring substrate)という概念も提唱された22).ここでは,これら三者を比較し,タンパク質局在化装置をモニターするアレストペプチドの共通性と多様性,および,タンパク質局在化装置との結びつきについて考察したい.
SecM, MifM, VemPは,いずれもC末端付近にそれぞれに固有の翻訳アレストモチーフを持ち,翻訳アレストを介して,同じmRNA上の下流に存在する標的遺伝子(タンパク質局在化因子をコードする)の翻訳をシスに制御する.また,生化学および遺伝学的な解析から,三者はいずれもペプチジル転移反応を阻害することで翻訳アレストを引き起こすことが示唆された21–23).このような大まかな図式は互いに類似しているが,翻訳アレストを引き起こす分子メカニズムにはそれぞれ違いがあることも明らかになってきている.
三者の翻訳アレストモチーフのアミノ酸配列には明確な共通性がみられず(図3),また,クライオ電子顕微鏡を用いた構造解析からは,リボソームトンネル内で,これらの分子がそれぞれ異なるコンホメーションをとることが示唆されている.たとえば,SecMやMifMはトンネル内で二次構造を形成しないが24, 25),VemPは少なくとも二つのαヘリックス構造を含む非常にコンパクトな二次構造をトンネルの内部で形成する11).また,MifMやVemPは,トンネルやPTC付近のリボソーム成分との相互作用を介してリボソームのAサイトを不活性化し,アミノアシルtRNAの受容を阻害することが示唆されたが11, 25),SecMの場合,PサイトのペプチジルtRNAの配置に影響を与えることでペプチジル転移反応を阻害することが示唆された24).
SecMは,165番目のコドンがリボソームのPサイトに位置した状態で翻訳伸長を停止する.Aサイトに位置する166番目のプロリンコドンを他のアミノ酸のコドンや終止コドンに置換すると翻訳アレストが起こらなくなる21).Aサイトコドンの翻訳アレストへの寄与はアレスト研究の興味深いトピックの一つであるが26),SecMはとりわけAサイトコドンのアミノ酸の種類がプロリンであることがアレストに重要である点が特徴的である.SecMの翻訳アレストには,Aサイトのみならず,アレスト部位周辺のアミノ酸配列が重要であり,この領域はSecMホモログ間で配列がよく保存されている27, 28).一方,すでに述べたように,MifMのmultisite stallingは,Pサイトに89Asp,90Ala,91Gly,92Serが来たときにそれぞれ起こる23).つまり,SecMと異なり,PサイトやAサイトに特定種類のアミノ酸が位置することは,MifMの翻訳アレストに必須ではない.
MifMやVemPは,Aサイトに人工的に終止コドンを導入するとペプチジルtRNAの加水分解を伴う翻訳終結をも阻害するが22, 23),AサイトのプロリルtRNAが必須であるSecMの翻訳アレストは,このプロリンコドンの終止コドンへの置換により無効となり,速やかな翻訳終結が行われる21).したがって,MifMとVemPは,翻訳伸長のみならず翻訳終結をアレストする潜在的な性質を持つが,SecMはそのような性質を持たない.
2)物理的引っぱり力がアレスト解除の普遍的なトリガーとなる
翻訳アレストは異なる分子機構により引き起こされるが,翻訳アレストの解除は合成途上鎖が局在化(膜透過や膜組込み)の過程に参加することが引き金となっている点で共通である.おそらく,膜透過や膜組込の過程でポリペプチド鎖を引っぱる力が生じ,その引っぱり力がアレストモチーフとリボソーム成分との相互作用に打ち勝ち翻訳アレストが解除されると考えられている.たとえば,MifMのN末端の膜挿入シグナルをSec依存的な分泌シグナルに人工的に置き換えたキメラアレストペプチドは,Sec依存的な分泌によって翻訳アレストが解除される29).本来であればYidCによる膜挿入によって解除されるMifMの翻訳アレストを分泌に伴う引っぱり力が解除しうるというこの結果は,引っぱり力がアレスト解除の普遍的な引き金になりうることを示唆している.Bustamanteらは,1分子操作技術を駆使し,SecMの合成途上鎖をN末端側から光ピンセットで引っぱることでその翻訳アレストが解除されること,すなわち,物理力がアレスト解除を誘起できることを直接的に示した30).また,Bustamanteのグループとvon Heijneのグループは,SecMのアレストモチーフのN末端側のトンネル出口周辺にフォールディングするドメインを人工的に融合すると,そのドメインのフォールディングによって生じた引っぱり力がSecMの翻訳アレストを解除すること30),また,トンネルの内部でも小さなドメインのフォールディングが開始されることを見いだした12).この結果も,引っぱり力がアレスト解除の普遍的な引き金になりうることを支持している.アレストペプチドがタンパク質の局在化装置のモニタリングにしばしば利用されるのは,局在化が引っぱり力を伴う過程だからであると推察できる.逆に,引っぱり力を生み出す細胞内システムであれば,タンパク質の局在化経路以外でも,アレストペプチドがモニタリング機構として利用されるといった可能性もある.
一方で,ATPaseによって駆動されるSec依存的なタンパク質の分泌と,ATP非依存的な過程であるYidC依存的な膜挿入とでは,生じる引っぱり力の強さや性質が異なる可能性も考えられる.アレストモチーフがそれぞれの解除機構に対して最適化されたものであるのか,また,それがアレスト機構の多様性を生み出す要因になりえたかどうかといった問題も興味深い.なお,これらのアレストペプチドは,それぞれ非常に限られた生物種の範囲内でのみ保存されており,たとえば,SecMはγ-プロテオバクテリアのごく一部の種に,MifMはバシラス目の一部の種に,また,VemPはビブリオ属およびその近縁種のみに保存されている14, 22, 27).おそらく,これらのアレストペプチドは,進化の過程で比較的最近になってそれぞれの生物種の祖先が独立に獲得したものと考えられる.よって,アレストペプチドの多様な個性は進化の後づけであるのかもしれない.
6. 細胞内代謝産物・低分子化合物を感知する翻訳アレスト因子
SecM, MifM, VemPは,翻訳に必要な最小限の因子のみで翻訳アレストを引き起こすことができ,補助因子を必要としない.一方,ある種のアレストペプチドは,補助因子依存的に翻訳アレストを起こすことが知られている.その性質の違いから,前者を内因性の(intrinsic)翻訳アレスト,後者を誘導性の(inducible)翻訳アレストとして区別することができる1).内因性のアレストペプチドは,アレスト解除の機構が作動するかしないかによってアレスト状態が制御されるが,誘導性のアレストペプチドは,アレストに必要な補助因子の濃度によってアレストの制御がなされる.以下,細胞の代謝産物や抗生物質を感知し,代謝酵素や抗生物質耐性遺伝子の発現を調節する誘導性アレストペプチドについて紹介する.
1)細胞内トリプトファン濃度をモニターする大腸菌TnaC
大腸菌のトリプトファン分解酵素であるTnaAは細胞内に過剰なトリプトファンが存在するときにその遺伝子の転写が亢進することで合成が誘導される.上流遺伝子tnaCとtnaAとの間には,転写終結因子rho依存的なターミネーターがあり,細胞内トリプトファン濃度が低いときにはそこで転写が終結し,下流にあるtnaAは転写されない[転写減衰(transcriptional attenuation)]31).ところが,細胞内トリプトファン濃度が高いときには,この転写終結が起こらずに,RNAポリメラーゼは下流のtnaA遺伝子も併せて転写する.その切り替えのメカニズムは次のとおりである.tnaCを翻訳するリボソームは,トリプトファン高濃度条件下でtnaCの終止コドンの位置でmRNA上に停滞する.この位置にはRhoのmRNAへのエントリーに必要なrutと呼ばれるサイトが存在するが,mRNA上に停滞したリボソームはこのrutサイトを覆い隠す.そのため,RhoがmRNA上にロードされず,tnaC-tnaA間にあるターミネーターが働かなくなる32).
tnaCの終止コドン上でのリボソームの停滞はtnaCの翻訳終結が阻害されることにより起こる32, 33).tnaCのコードする24アミノ酸残基の短いポリペプチド鎖をすべて合成し終えたリボソームは,トリプトファン濃度が低いときには,翻訳終結因子RF2がtnaCの終止コドンUGAを認識することで翻訳を終結し,mRNAから解離する.ところが,トリプトファン濃度が高いときには,この翻訳終結がアレストし,リボソームはmRNA上に停滞することになる.この翻訳終結アレストにはTnaCの合成途上鎖上に存在する複数のアミノ酸残基からなるアレストモチーフと,リボソームに結合したトリプトファンが必要である32, 34–37).TnaCは,翻訳装置の挙動の変化が転写を調節するユニークな例であり,転写と翻訳が共役している細菌ならではの巧妙な機構といえる.
2)抗生物質を感知し薬剤耐性遺伝子を誘導するアレストペプチド
抗生物質耐性遺伝子の中には,ターゲットとなる抗生物質の存在を感知してその発現が誘導されるものがある.たとえば,グラム陽性菌で見いだされたクロラムフェニコールアセチル基転移酵素をコードするcat遺伝子や38–40),大腸菌で見いだされ多剤排出ポンプをコードするcmlAなどのクロラムフェニコール耐性遺伝子41),さらには,エリスロマイシン耐性遺伝子であるermA, ermB, ermC, ermDなどでそのような発現制御がみられる42, 43).共通しているのは,これらの遺伝子と同じオペロン内の上流に小さなORFが存在し,そのORFがコードする短いペプチド鎖が,それぞれの抗生物質依存的に翻訳伸長アレストを引き起こす点である.たとえば,クロラムフェニコール耐性遺伝子の上流にはクロラムフェニコール存在下で翻訳伸長アレストを起こすORFが存在し,エリスロマイシン耐性遺伝子の上流には,マクロライド系抗生物質に応答して翻訳伸長アレストを起こすORFが存在する.上流ORFの翻訳アレストは,mRNAの二次構造のリモデリングを介して薬剤耐性遺伝子の翻訳開始に必要なSD配列を露出状態に維持することで,その耐性遺伝子の合成を翻訳レベルで亢進する.
真正細菌のみならず,真核生物においても,誘導性翻訳アレストを利用したさまざまな機構が存在する.ここでは,真核生物で見いだされた誘導性翻訳アレストの代表例を紹介する.
1)アルギニンを感知しその合成にブレーキをかけるアレストペプチドAAP
アカパンカビのarg-2は,アルギニン生合成の初期に働くカルバモイルリン酸合成酵素の小サブユニットをコードしている.アルギニンが不足すると誘導され,アルギニンが過剰になるとその発現は抑制される.アルギニンに応答したarg-2の発現調節は,arg-2の上流ORF(uORF)であるAAP(arginine attenuator peptide)の働きによる.AAPは24アミノ酸残基からなる小さなポリペプチド鎖であるが,アルギニン存在下ではAAPを合成するリボソームは翻訳終結できずに終止コドンをAサイトに配置した状態でmRNA上に停滞する.その結果,下流のメインORFであるarg-2へのリボソームの供給が阻害され,arg-2の翻訳が抑制される44).出芽酵母でも同様の機構が保存されているが,この場合,uORFでのリボソームの停滞は,リボソームが下流ORFの開始コドンをスキャニングすることを妨げるのみならず,mRNAの分解(nonsense-mediated mRNA decay)をも引き起こすことが示唆された45).
真正細菌の場合,翻訳アレストペプチドが翻訳アレストを起こすと下流の遺伝子の発現が上昇するような仕組みが一般的である.ところが,真核生物のuORFがアレストペプチドである場合,その翻訳アレストは下流のメインORFの発現を負に制御する.真正細菌と真核生物の翻訳開始機構の違いを反映したものと思われる.
2)メチオニン生合成経路を調節するアレストペプチドCGS1
シロイヌナズナにおいてメチオニン生合成経路の初期のステップを担うシスタチオン-γ-シンターゼはCGS1遺伝子によってコードされ,その発現が,メチオニン誘導体であるS-adenosyl-L-methionine(SAMもしくはAdoMet)によって負のフィードバック制御を受ける.CGS1の第一のエクソンにはMTO1と呼ばれる領域が存在している.その領域の数アミノ酸残基下流において,CGS1の翻訳伸長は,SAM依存的にアレストする46).この翻訳アレストは,CGS1の翻訳自体を低下させるのみならず,mRNAの切断を誘起し,それらの結果としてシスタチオン-γ-シンターゼの合成量を低下させる47).前述のAAPがuORFで起こる翻訳アレストによるメインORFの発現制御という図式だったのに対し,CGS1の翻訳アレストは酵素本体のコード領域内で起こる.
CGS1の翻訳アレストは,94番目のセリンがペプチド鎖に取り込まれた後に起こるはずの転位(translocation)が阻害されることによって生じることが示唆されている46).現時点で,転位過程を阻害することが示唆されている翻訳アレストは,このCGS1と,後述するXBP1u48, 49)のみであり,真正細菌での報告例はない.アレストペプチドがリボソームの転位を阻害する機構はまったく不明であり,非常に興味深い問題である.
8. XBP1uによる翻訳の一時休止を利用したmRNA局在制御
アレストペプチドの多くは,特定の条件下で起こる,もしくは解除されるような条件的翻訳アレストを利用して細胞内外の環境を感知するセンサーとしての特性を発揮する.一方,ここで紹介するXBP1uはセンサーとして働くわけではなく,翻訳伸長の一時休止を巧妙に利用して自身のmRNAを小胞体膜に局在化させるユニークな因子である.
ヒトの小胞体ストレス応答における転写活性化因子XBP1をコードするmRNAは,小胞体ストレス依存的にスプライシングを受け,ORF内部にある26塩基からなるイントロンが除かれる.その結果,XBP1 mRNA内の3′側領域のコドンフレームがスイッチし,成熟型XBP1タンパク質をコードするXBP1s(spliced form)へと変換される50).XBP1の転写活性化ドメインはイントロンよりも下流にコードされており,このスプライシングを受けて初めてXBP1の開始コドンとインフレームになる.そのため,スプライシング前駆体であるXBP1u(unspliced form)mRNAがコードするタンパク質は転写活性化ドメインを持たず,転写活性化因子として機能することはできない.
XBP1u mRNAのスプライシング酵素IRE1αは小胞体タンパク質であり,小胞体ストレスに伴い活性化される.そのため,XBP1u mRNAが効率のよいスプライシングを受けるためには,XBP1u mRNAがIRE1αの存在する小胞体膜近傍に局在化される必要がある.XBP1uの小胞体膜へのターゲティングには,XBP1uの翻訳産物が重要な役割を果たす.XBP1uの翻訳産物は261アミノ酸残基からなる.N末端の165アミノ酸残基はXBP1sのものと同一であるが,C末端側には疎水性アミノ酸残基に富んだ領域(HR)があり,さらにC末端の終止コドン近傍には翻訳伸長を一時休止させるアミノ酸配列がある51).HRはSRP依存的にXBP1uの翻訳産物を小胞体膜へとターゲティングさせる役割を持つ.XBP1uの翻訳伸長の一時休止は,XBP1uの合成途上ポリペプチド鎖とリボソーム,mRNAを一つの複合体として長時間維持する効果がある.そのため,XBP1uが小胞体膜に局在化すれば,XBP1uのmRNAもまた小胞体膜近傍に配置されることとなる.XBP1は,翻訳伸長の一時休止を巧みに利用しmRNAの局在を制御するユニークな例であるが,同じmRNAが一つのフレームで転写活性化ドメインを,もう一つのフレームで疎水性領域と翻訳ポーズモチーフをコードする興味深い例でもある.
前項までで,代表的なアレストペプチドについて紹介してきたが,この節では,アレストペプチド研究がもたらした新概念や意義,他の研究分野への波及効果について論じてみたい.
1)アレストペプチド研究からみえてきた翻訳の実像
SecMの節でもふれたように,アレストペプチドの発見は,翻訳途上で働くタンパク質の存在を示した.また,その分子機構を解明する過程で,タンパク質のアミノ酸配列に自身の翻訳伸長速度を規定する情報が内包されうること,その情報は主にPTCやリボソームトンネル成分によってデコードされることなどが明らかになった.このような翻訳の速度制御が,より一般的なタンパク質の合成過程でも普遍的に行われているのかどうかについてはまだ明らかではないが,次世代シーケンサーを利用したリボソームフットプリンティング解析などからは,翻訳伸長が必ずしも一定速度で進行するわけではなく,頻繁にポーズすることが明らかにされつつある49, 52, 53).とりわけ,大腸菌のタンパク質合成の網羅的プロファイル解析からは,細胞内の8割以上のタンパク質が,翻訳伸長の途上で1回以上ポーズするという驚くべき結果が示された54).それらの解析からみえてきた翻訳の実像は,リボソームがmRNA上をスムーズに滑走するといったものではなく,mRNA上でリボソームがしばしば翻訳停止と再開を繰り返し,速度の緩急を伴って進行するといったものである.それぞれの翻訳のポージングの原因やその生物学的な意味合いについては今後の重要な研究課題であるが,一つの可能性としては,翻訳のポージングには,新生タンパク質のフォールディングや局在化を翻訳と共役させやすくする役割があることがあげられる.さらに,アレストペプチドが引き起こす翻訳アレストがタンパク質の局在化やフォールディングなどによって解除されるという研究結果を拡張して解釈すると,タンパク質が自身のフォールディングや局在化の進捗状況,さらには,さまざまな細胞内環境に応じて自身の翻訳伸長をフィードバック制御しながら自律的に自身の構造形成や局在化を促進するという魅力的な可能性もみえてくる.実際,真核生物において,強い熱ショックストレス条件下やシャペロンが不足した条件下などで翻訳伸長のポージングがグローバルに起こることも報告されている55, 56).翻訳は,自動的に進行する機械的な文字変換作業ではなく,リボソーム,新生鎖,細胞内因子の三者のネゴシエーションによって最適化されたかたちで進行する非常に有機的な過程であるという概念が生まれつつある.コドン使用や二次構造などmRNA側の要因による翻訳速度の調節に比べ,翻訳途上鎖による翻訳制御はより応答性が高く,個々の産物の状況をリアルタイムに反映した翻訳制御を可能にする.
2)リボソームトンネルの構造とタンパク質の分子進化
仮に,タンパク質の正しい構造形成がリボソームとの相互作用を介した翻訳速度調節に依存するとすれば,リボソームトンネルの構造がタンパク質の分子進化に影響を与えてもおかしくはない.アレストペプチドの場合であれば,たとえば,枯草菌MifMは,大腸菌のリボソームで合成すると,効率のよい翻訳アレストを起こすことができない29).変異解析から,大腸菌と枯草菌のリボソームタンパク質L22の構造の違いが種特異性の原因の一つであることが示唆されている25).つまり,MifMの翻訳アレストモチーフは枯草菌リボソームトンネルの構造に最適化されたかたちで進化してきたことを示している.このことは,アレストペプチドの多様性を生み出す一つの要因とも考えられる.種固有のリボソームの構造がアレストペプチドの分子進化に影響を与えたのと同様に,もしかしたら,一般的なタンパク質の分子進化にもリボソームの構造が影響を与える場合があるかもしれない.また,異種のリボソームを用いたリコンビナントタンパク質の合成がしばしば成功しない理由の一つとして,タンパク質のアミノ酸配列がネイティブのリボソームと正しく相互作用できるように(また,不必要な相互作用を避けるように)最適化されているためである可能性も考えられる.
3)アレストペプチドの研究ツールとしての応用例
アレストペプチドを研究ツールとして利用し,他の研究分野の発展に貢献した例もある.たとえば,翻訳伸長は非常に速く進行する過程であり,その途上で起こるイベントを観察し解析することは本来困難であるが,翻訳アレストモチーフを利用し,翻訳伸長を任意の場所で停止させると,翻訳伸長の途上で起こるイベントを観察することが可能になる.たとえば,翻訳と共役して起こるタンパク質のフォールディングや局在化の研究などにアレストモチーフを利用した研究例がいくつも報告されている12, 57–60)また,アレストペプチドはリボソームディスプレイにも利用されている61, 62).
多くの翻訳アレスト因子は,細胞内の特定の環境変化を感知するセンサーとしての特性を持つ.いずれも遺伝子発現を転写や翻訳レベルで制御する仕組みを併せ持っているため,lacZやGFPのようなレポーター遺伝子を利用すれば,細胞内の環境をβ-ガラクトシダーゼ活性や蛍光強度を指標に知ることができるはずである.たとえば,枯草菌MifMによって発現制御を受けるyidC2は,YidCの第一のホモログSpoIIIJの活性低下に呼応してその発現が誘導される.そのため,yidC2遺伝子とlacZをインフレームに融合したようなyidC2′-lacZ遺伝子を持つレポーター株は,SpoIIIJの活性低下に呼応してβ-ガラクトシダーゼ活性を上昇させる.この株を利用すれば,細胞の持つタンパク質の膜組込み能をβ-ガラクトシダーゼ活性を指標に測定することができる14).実際,このレポーター株のβ-ガラクトシダーゼ活性を上昇させるようなSpoIIIJ変異の解析から,タンパク質膜組込みに重要なYidC分子内のアミノ酸残基や領域,性質などがいくつか明らかにされた17, 19).同様のレポーター株は,他のアレストペプチドでも構築可能であり,タンパク質局在化装置の研究をはじめ,さまざまなかたちで応用することが可能であると思われる.
天然のアレストペプチドを改変することで,センサーとしての特性を変化させることもできる.たとえば,すでに述べたように,MifMのN末端にある膜貫通領域を分泌シグナルに置き換えると,このキメラタンパク質は,YidC依存的な膜組込み経路ではなく,Sec依存的な分泌経路をモニターするようになる29).このような互換性を利用すれば,細胞内で引っぱり力を生み出すイベントを感知するさまざまな力学センサーを人工的に生み出すことができるかもしれない.von Heijneらは,SecMやXBP1u由来の翻訳アレスト配列を膜タンパク質のC末端側に人工的に融合し,タンパク質の膜挿入に伴ってこれらの翻訳アレストが解除されることを示した.このアレスト解除は膜挿入に伴う引っぱり力によるものであるとの前提の下で,翻訳アレストの解除の効率を指標に,システマティックな変異解析から,Sec依存的な膜挿入時には少なくとも引っぱり力を生み出す過程が二つ存在することを提唱した59).翻訳アレストを力学センサーとして巧妙に利用した実例といえる.
4)創薬の新たなターゲットとしての翻訳アレスト
最近,翻訳アレストを引き起こす化合物が新たな創薬につながる可能性を示唆する報告がなされた.PCSK9(proprotein convertase subtilisin/kexin type 9)は血漿中のLDLコレステロール量を正に調節する働きを持つが,Lingnerらは,PF-06446846と呼ばれる化合物をラットに経口投与すると,血漿中のPCSK9の量が低下し,それに伴い,血中総コレステロール量が低下することを報告した63).そのため,この化合物は高コレステロール血症の治療薬としての利用が期待される.興味深いことに,PF-06446846は,PCSK9の翻訳伸長を途中でアレストさせることでPCSK9の特異的合成阻害剤として働くらしい63).化合物がある特定のタンパク質の翻訳アレストを誘導する現象は,TnaCやErmCL, CGS1などの低分子化合物依存的な誘導性翻訳アレストに類似している.PF-0644846の場合,完成したタンパク質の三次構造ではなく新生ポリペプチド鎖上の一次構造(アミノ酸配列)を標的とする点がユニークである.標的タンパク質の特定の立体構造をターゲットにした既存の創薬アプローチでは特異性の高い薬剤を生み出すことが難しいケースなどでは,このような薬剤が有望な選択肢になるかもしれない.現時点では,そのような薬剤スクリーニング法の確立など,克服すべき問題は多いと思われるが,誘導性翻訳アレストは,従来とはまったく異なるコンセプトに基づいた創薬へとつながる可能性を秘めているものと思われる.
5)nascent chain biology:翻訳途上鎖が主役を演じる生命現象を理解する
ごく最近,新生ポリペプチド鎖内のある特定のアミノ酸配列がリボソームに働きかけることにより,リボソームの大小サブユニット間の結合が不安定化し,翻訳伸長が途中で強制的に終了する現象が大腸菌において見いだされた64).IRD(intrinsic ribosome destabilization)と命名されたこの現象の発見は,翻訳途上のポリペプチドがリボソームと相互作用した結果,翻訳アレストのようにリボソーム機能に影響するというよりは,機能の前提となるリボソームの構造に影響を与える点で興味深い.さらに,IRDを引き起こすMgtLと呼ばれる短いポリペプチドが,細胞内マグネシウムイオンの濃度をモニターし,マグネシウム濃度を反映した頻度でIRDを誘導することでマグネシウムトランスポーターMgtAの発現調節に関与することも示唆された64).翻訳アレストはさまざまな形で細胞の機能調節に利用されているが,IRDもまた,それを巧みに利用した細胞機能制御が存在するようだ.なお,IRDは,終止コドンに依存しない翻訳終結シグナルとみなすこともできるため,DNAの塩基配列がそのままのかたちでタンパク質の一次構造を規定するとは限らないことを暗示している.
本稿で述べてきたように,アレストペプチド研究の進展は,翻訳途上鎖が主体となって細胞機能を調節する生命現象への関心を呼び起こし,一方では,翻訳やタンパク質のフォールディング,局在化などの関連分野と結びついて,nascent chain biologyともいうべき研究領域へと発展しつつある.セントラルドグマという生命の基本的な仕組みと密接に関連した研究領域であるだけに,今後とも,その進展が他のさまざまな研究分野へと波及する可能性を大いに秘めているように思う.完成されたタンパク質を対象とした研究だけでは理解できなかった生命現象に翻訳途上鎖の関与という新たな視点を加えることでその理解が深まるといったことは,今後ともさまざまな研究分野において起こりうるのではないかと考えられる.
あらゆるアミノ酸配列を組み合わせて多種多様なタンパク質を生み出すことがリボソームの最も重要な存在意義であるとすれば,翻訳アレストやIRDを起こすようなアミノ酸配列の存在は,ある意味,リボソームの翻訳装置としての万能性を揺るがす不都合な存在とみることもできる.ところが,リボソームの持つそのような不完全性ゆえに,実際の翻訳は,リボソームが単独で行う単純で円滑な文字変換作業ではなく,その翻訳産物やさまざまな細胞内因子がその制御にリアルタイムに関与することで,個々の翻訳産物に最適化されたかたちで進行するような可塑性を内包したものになりえたのかもしれない.さらに,そのようなリボソームであるからこそ,アレストペプチドやIRDを利用した多様な細胞制御機構の進化的な獲得を可能にしたのであろう.生物は,個々のシステムを完璧なものへと進化させるよりは,それらが持つ不完全さを許容したり,時にはそれらを逆手にとって多様な機能を生み出したりする方向に進化する性質があるのかもしれない.その結果生み出された可塑性を内包したシステムに,生物の一つの本質,「生物らしさ」があるように思える.
謝辞Acknowledgments
師として,また,共同研究者としてアレストペプチドに関する研究を長年支えてくださいました伊藤維昭博士に特別の感謝の意を表します.また,MifMの発見当初から研究を支えてくださったKit Pogliano博士,新生鎖やアレストペプチド研究をともに遂行し,また,さまざまな形で支援してくださいました,下川(千葉)直美博士,藤原圭吾博士,秋山芳展博士,森博幸博士,石井英治博士,中戸川仁博士に感謝します.伊藤維昭博士と下川(千葉)直美博士には,本稿の執筆に際して有益なアドバイスをいただきましたこともあわせて感謝します.
引用文献References
1) Ito, K., Chiba, S., & Pogliano, K. (2010) Biochem. Biophys. Res. Commun., 393, 1–5.
2) Ito, K. & Chiba, S. (2013) Annu. Rev. Biochem., 82, 171–202.
3) Tenson, T. & Ehrenberg, M. (2002) Cell, 108, 591–594.
4) Park, E. & Rapoport, T.A. (2012) Annu. Rev. Biophys., 41, 21–40.
5) Tsukazaki, T., Mori, H., Echizen, Y., Ishitani, R., Fukai, S., Tanaka, T., Perederina, A., Vassylyev, D.G., Kohno, T., Maturana, A.D., Ito, K., & Nureki, O. (2011) Nature, 474, 235–238.
6) Furukawa, A., Yoshikaie, K., Mori, T., Mori, H., Morimoto, Y.V., Sugano, Y., Iwaki, S., Minamino, T., Sugita, Y., Tanaka, Y., & Tsukazaki, T. (2017) Cell Reports, 19, 895–901.
7) Nakatogawa, H. & Ito, K. (2001) Mol. Cell, 7, 185–192.
8) Nissen, P., Hansen, J., Ban, N., Moore, P.B., & Steitz, T.A. (2000) Science, 289, 920–930.
9) Wilson, D.N. & Beckmann, R. (2011) Curr. Opin. Struct. Biol., 21, 274–282.
10) Tu, L.W. & Deutsch, C. (2010) J. Mol. Biol., 396, 1346–1360.
11) Su, T., Cheng, J., Sohmen, D., Hedman, R., Berninghausen, O., von Heijne, G., Wilson, D.N., & Beckmann, R. (2017) eLife, 6.
12) Nilsson, O.B., Hedman, R., Marino, J., Wickles, S., Bischoff, L., Johansson, M., Müller-Lucks, A., Trovato, F., Puglisi, J.D., O’Brien, E.P., Beckmann, R., & von Heijne, G. (2015) Cell Reports, 12, 1533–1540.
13) Nakatogawa, H. & Ito, K. (2002) Cell, 108, 629–636.
14) Chiba, S., Lamsa, A., & Pogliano, K. (2009) EMBO J., 28, 3461–3475.
15) Chiba, S. & Ito, K. (2015) J. Bacteriol., 197, 99–107.
16) Kuhn, A., Koch, H.G., & Dalbey, R.E. (2017) Ecosal Plus, 7.
17) Kumazaki, K., Chiba, S., Takemoto, M., Furukawa, A., Nishiyama, K., Sugano, Y., Mori, T., Dohmae, N., Hirata, K., Nakada-Nakura, Y., Maturana, A.D., Tanaka, Y., Mori, H., Sugita, Y., Arisaka, F., Ito, K., Ishitani, R., Tsukazaki, T., & Nureki, O. (2014) Nature, 509, 516–520.
18) Kumazaki, K., Kishimoto, T., Furukawa, A., Mori, H., Tanaka, Y., Dohmae, N., Ishitani, R., Tsukazaki, T., & Nureki, O. (2014) Sci. Rep., 4, 7299.
19) Shimokawa-Chiba, N., Kumazaki, K., Tsukazaki, T., Nureki, O., Ito, K., & Chiba, S. (2015) Proc. Natl. Acad. Sci. USA, 112, 5063–5068.
20) Rubio, A., Jiang, X., & Pogliano, K. (2005) J. Bacteriol., 187, 5000–5002.
21) Muto, H., Nakatogawa, H., & Ito, K. (2006) Mol. Cell, 22, 545–552.
22) Ishii, E., Chiba, S., Hashimoto, N., Kojima, S., Homma, M., Ito, K., Akiyama, Y., & Mori, H. (2015) Proc. Natl. Acad. Sci. USA, 112, E5513–E5522.
23) Chiba, S. & Ito, K. (2012) Mol. Cell, 47, 863–872.
24) Bhushan, S., Hoffmann, T., Seidelt, B., Frauenfeld, J., Mielke, T., Berninghausen, O., Wilson, D.N., & Beckmann, R. (2011) PLoS Biol., 9, e1000581.
25) Sohmen, D., Chiba, S., Shimokawa-Chiba, N., Innis, C.A., Berninghausen, O., Beckmann, R., Ito, K., & Wilson, D.N. (2015) Nat. Commun., 6, 6941.
26) Ramu, H., Vazquez-Laslop, N., Klepacki, D., Dai, Q., Piccirilli, J., Micura, R., & Mankin, A.S. (2011) Mol. Cell, 41, 321–330.
27) van der Sluis, E.O. & Driessen, A.J. (2006) Trends Microbiol., 14, 105–108.
28) Yap, M.N. & Bernstein, H.D. (2009) Mol. Cell, 34, 201–211.
29) Chiba, S., Kanamori, T., Ueda, T., Akiyama, Y., Pogliano, K., & Ito, K. (2011) Proc. Natl. Acad. Sci. USA, 108, 6073–6078.
30) Goldman, D.H., Kaiser, C.M., Milin, A., Righini, M., Tinoco, I. Jr., & Bustamante, C. (2015) Science, 348, 457–460.
31) Stewart, V., Landick, R., & Yanofsky, C. (1986) J. Bacteriol., 166, 217–223.
32) Gong, F., Ito, K., Nakamura, Y., & Yanofsky, C. (2001) Proc. Natl. Acad. Sci. USA, 98, 8997–9001.
33) Gong, F. & Yanofsky, C. (2001) J. Biol. Chem., 276, 1974–1983.
34) Gong, F. & Yanofsky, C. (2002) Science, 297, 1864–1867.
35) Bischoff, L., Berninghausen, O., & Beckmann, R. (2014) Cell Reports, 9, 469–475.
36) Seidelt, B., Innis, C.A., Wilson, D.N., Gartmann, M., Armache, J.P., Villa, E., Trabuco, L.G., Becker, T., Mielke, T., Schulten, K., Steitz, T.A., & Beckmann, R. (2009) Science, 326, 1412–1415.
37) Martinez, A.K., Gordon, E., Sengupta, A., Shirole, N., Klepacki, D., Martinez-Garriga, B., Brown, L.M., Benedik, M.J., Yanofsky, C., Mankin, A.S., Vazquez-Laslop, N., Sachs, M.S., & Cruz-Vera, L.R. (2014) Nucleic Acids Res., 42, 1245–1256.
38) Bruckner, R. & Matzura, H. (1985) EMBO J., 4, 2295–2300.
39) Alexieva, Z., Duvall, E.J., Ambulos, N.P. Jr., Kim, U.J., & Lovett, P.S. (1988) Proc. Natl. Acad. Sci. USA, 85, 3057–3061.
40) Rogers, E.J., Kim, U.J., Ambulos, N.P. Jr., & Lovett, P.S. (1990) J. Bacteriol., 172, 110–115.
41) Gu, Z., Harrod, R., Rogers, E.J., & Lovett, P.S. (1994) Proc. Natl. Acad. Sci. USA, 91, 5612–5616.
42) Vazquez-Laslop, N., Thum, C., & Mankin, A.S. (2008) Mol. Cell, 30, 190–202.
43) Ramu, H., Mankin, A., & Vazquez-Laslop, N. (2009) Mol. Microbiol., 71, 811–824.
44) Wang, Z. & Sachs, M.S. (1997) Mol. Cell. Biol., 17, 4904–4913.
45) Gaba, A., Jacobson, A., & Sachs, M.S. (2005) Mol. Cell, 20, 449–460.
46) Onouchi, H., Nagami, Y., Haraguchi, Y., Nakamoto, M., Nishimura, Y., Sakurai, R., Nagao, N., Kawasaki, D., Kadokura, Y., & Naito, S. (2005) Genes Dev., 19, 1799–1810.
47) Haraguchi, Y., Kadokura, Y., Nakamoto, M., Onouchi, H., & Naito, S. (2008) Plant Cell Physiol., 49, 314–323.
48) Yanagitani, K., Kimata, Y., Kadokura, H., & Kohno, K. (2011) Science, 331, 586–589.
49) Ingolia, N.T., Lareau, L.F., & Weissman, J.S. (2011) Cell, 147, 789–802.
50) Yoshida, H., Matsui, T., Yamamoto, A., Okada, T., & Mori, K. (2001) Cell, 107, 881–891.
51) Yanagitani, K., Imagawa, Y., Iwawaki, T., Hosoda, A., Saito, M., Kimata, Y., & Kohno, K. (2009) Mol. Cell, 34, 191–200.
52) Ingolia, N.T., Ghaemmaghami, S., Newman, J.R., & Weissman, J.S. (2009) Science, 324, 218–223.
53) Li, G.W., Oh, E., & Weissman, J.S. (2012) Nature, 484, 538–541.
54) Chadani, Y., Niwa, T., Chiba, S., Taguchi, H., & Ito, K. (2016) Proc. Natl. Acad. Sci. USA, 113, E829–E838.
55) Liu, B., Han, Y., & Qian, S.B. (2013) Mol. Cell, 49, 453–463.
56) Shalgi, R., Hurt, J.A., Krykbaeva, I., Taipale, M., Lindquist, S., & Burge, C.B. (2013) Mol. Cell, 49, 439–452.
57) Mitra, K., Schaffitzel, C., Shaikh, T., Tama, F., Jenni, S., Brooks, C.L. 3rd, Ban, N., & Frank, J. (2005) Nature, 438, 318–324.
58) Evans, M.S., Ugrinov, K.G., Frese, M.A., & Clark, P.L. (2005) Nat. Methods, 2, 757–762.
59) Ismail, N., Hedman, R., Schiller, N., & von Heijne, G. (2012) Nat. Struct. Mol. Biol., 19, 1018–1022.
60) Schaffitzel, C. & Ban, N. (2007) J. Struct. Biol., 158, 463–471.
61) Ohashi, H., Shimizu, Y., Ying, B.W., & Ueda, T. (2007) Biochem. Biophys. Res. Commun., 352, 270–276.
62) Contreras-Martinez, L.M. & DeLisa, M.P. (2007) J. Mol. Biol., 372, 513–524.
63) Lintner, N.G., McClure, K.F., Petersen, D., Londregan, A.T., Piotrowski, D.W., Wei, L., Xiao, J., Bolt, M., Loria, P.M., Maguire, B., Geoghegan, K.F., Huang, A., Rolph, T., Liras, S., Doudna, J.A., Dullea, R.G., & Cate, J.H. (2017) PLoS Biol., 15, e2001882.
64) Chadani, Y., Niwa, T., Izumi, T., Sugata, N., Nagao, A., Suzuki, T., Chiba, S., Ito, K., & Taguchi, H. (2017) Mol. Cell, 68, 528–539.e5.
著者紹介Author Profile
 千葉 志信(ちば しのぶ)
千葉 志信(ちば しのぶ)京都産業大学総合生命科学部准教授.理学(博士).
略歴1997年北海道大学薬学部卒.99年同大学大学院薬学研究科修了.2002年京都大学大学院理学研究科博士過程修了.京都大学ウイルス研究所,UCSD, 京都産業大学等で博士研究員,助教等を経て,2014年より現職.
研究テーマと抱負翻訳途上鎖が主役を演じる生命現象の探求ならびにタンパク質のバイオジェネシスの研究.
趣味撮影,釣り,自然散策.