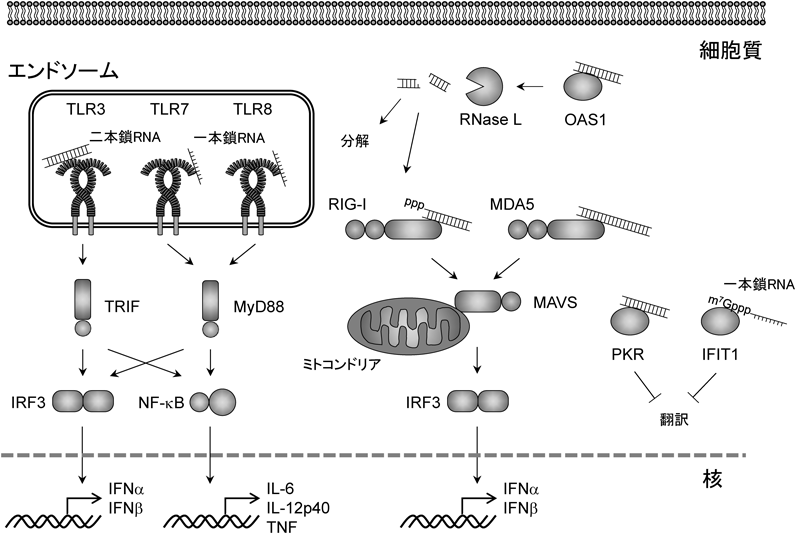
自然免疫は病原体感染を認識し迅速に免疫,炎症応答を誘導するシステムである.自然免疫細胞は,病原体の生存や感染に必要な共通の分子パターン(pathogen-associated molecular patterns:PAMPs)をパターン認識受容体(pattern recognition receptor:PRR)と呼ばれる生物種間で保存された病原体センサーにより認識する1).Toll様受容体(Toll-like receptor:TLR)は最もよく研究されているPRRであり,ヒトでは10種類,マウスでは12種類存在する.これらの受容体は細菌,真菌,ウイルスといった幅広い病原体の認識に関与する.さらにTLRの他にも,RIG-I様受容体(RIG-I-like receptor:RLR),NOD様受容体(NOD-like receptor:NLR)といった病原体センサーが特異的なリガンドを認識することで免疫応答を迅速かつ適切に引き起こし,病原体の侵入を防いでいる.PAMPsがPRRによって認識されると,PRRは特有のアダプター分子と会合し下流のシグナルを活性化することにより,NF-κBやAP-1といった転写因子がIL-6, IL-12p40などの炎症性サイトカインを誘導する(図1).病原体由来のRNA認識に関連するPRRとして,TLRファミリーではTLR3, TLR7, TLR8が知られており,そしてRLRファミリーのRIG-IとMDA5もまた,それぞれ特徴的なRNA配列を認識し,I型インターフェロン(type I IFN)の誘導に関与する2, 3).type I IFNは抗ウイルス応答において中心的な役割を有しており,さまざまなIFN誘導性遺伝子(interferon stimulating gene:ISG)の転写誘導に関わる.type I IFNは,ウイルス感染細胞のアポトーシスを誘導する一方,周囲の非感染細胞に対して感染抵抗性を付与する.また,樹状細胞の成熟やNK細胞の細胞傷害性を高める他,ウイルス特異的細胞傷害性T細胞の活性化に寄与する.一般に,自然免疫系におけるPRRの活性化により産生されたサイトカインやIFNが引き金となり,獲得免疫系が活性化される.T細胞は樹状細胞に提示された抗原を認識し,さらにサイトカインにより活性化する.B細胞はT細胞との相互作用により,クラススイッチ組換えと体細胞高頻度突然変異を起こし,抗原特異的な抗体産生細胞へと分化する.このように,自然免疫細胞は多種多様な病原体の侵入に対して迅速な炎症誘導を可能にしており,効率的に獲得免疫を活性化する橋渡し的な役割を果たしている.
免疫応答の過程で,RNAとその認識は,さまざまなステップにおいて重要な役割を果たしている.RNAは,RNAウイルスをはじめとした病原体のゲノムや複製中間体として機能するが,同時に,PAMPsとして宿主免疫系による病原体認識の標的となる.また,PRRシグナル経路の活性化によって誘導された免疫応答に関連するタンパク質をコードするmRNAも,厳密に制御されている.転写後制御はRNAの生成から分解に至るまでのあらゆるRNA代謝段階をつかさどる機構として定義され,炎症の誘導および収束においても大きく関与している.このようなRNA代謝の中心的役割を担っているのがRNA結合タンパク質(RNA binding protein:RBP)であり,自然免疫によるウイルスRNA認識や,炎症応答の転写後制御機構のあらゆる場面においてRNAとの相互作用を介して機能している4).プロテオームおよびin silicoの解析により,哺乳動物では約1500個のRBPが存在することが明らかとなっているが,これは転写因子の数とほぼ同程度である5).RNA代謝をつかさどる機能的ユニットとして,mRNAにRBPやnon-coding RNAが集合しリボヌクレオプロテイン(ribonucleoprotein:RNP)と呼ばれる複合体が形成される.この際,RNP複合体は細胞内局在を変えながらRBPの結合と解離によって絶えず変化しており,これにより時空間的なRNA代謝が可能となる.
本稿では,RNAを介した自然免疫応答の分子機構を,1)ウイルスなど外来RNAの認識機構とそれによる免疫応答,2)免疫応答がmRNAを介して調節される転写後制御機構に関して,最近我々が得た知見をふまえて概説したい.
1)ウイルスRNAセンサーの機能と自然免疫応答
感染したウイルスRNAを認識するPRRとしてTLRやRLRが知られている(図1).これらのPRRがRNAに結合すると細胞内シグナル伝達経路が活性化しNF-κBやIRF3といった転写因子を活性化することにより,type I IFNや炎症性サイトカインを誘導し,抗ウイルス応答を引き起こす.TLRファミリーの中で,ウイルスRNAセンサー受容体としてTLR3, TLR7が知られており,ヒトではTLR8もRNA認識に関与する1, 6–8).これらはいずれも,エンドソームにおいてRNAを認識する.一方,細胞質内におけるRNA認識機構にはRLRファミリーに属するRIG-I(別名DDX58)やMDA5(別名IFIH1)が知られている.両者はDExD/H box RNAヘリカーゼドメインとC末端のRNA結合領域を持ち,さらにN末端に細胞内シグナル活性化に必須のcaspase recruitment domains(CARD)ドメインを持つ9).LGP2(別名DHX58)はRIG-I様ヘリカーゼ領域を有しているがCARDは持たない.LGP2の機能はMDA5シグナルを促進することで,RNAウイルス認識に寄与するが10),LGP2によるRNA認識の意義に関しては十分明らかではない.
PRRシグナル伝達は,各々のTLR, RLRに特有のアダプター分子が会合することで,シグナル経路間で共通したモジュールを介して下流の転写因子を活性化する.TLR3以外のTLRはアダプター分子MyD88を介したシグナル伝達経路を活性化させ,下流のIKK複合体あるいはMAPキナーゼを介して,転写因子NF-κBやAP-1を活性化し,炎症性サイトカインを誘導する.TLR3にはTRIFと呼ばれる異なるアダプター分子が会合する.TRIFを介したシグナル伝達はNF-κBやAP-1だけでなく,キナーゼ群としてTBK1-IKKεを活性化し,IRF3をリン酸化する.リン酸化されたIRF3は二量体を形成し核内へ移行することにより,IFNβなどtype I IFN遺伝子発現を誘導する.一方,RIG-IやMDA5はミトコンドリアに局在するMAVS(別名IPS-1, CARDIF, VISA)を介して,TLR3のようにTBK1-IKKεを介してIRF3を活性化し,type I IFNを誘導する11).
2)パターン認識受容体により認識されるRNAの特徴
自然免疫は,自己と非自己の識別の観点から,宿主,および外来RNAを厳密に区別していることが明らかとなってきた.細胞質内における長い二本鎖RNA(double-stranded RNA:dsRNA)は,RNAウイルスに感染した細胞でRNAウイルスゲノム,もしくはウイルス複製の中間体として産生され,基本的に非感染細胞には存在しない.このため,dsRNAは宿主RNAセンサーのリガンドとして重要である.また,poly(I : C)は合成dsRNAアナログとして,TLR3だけでなく,RIG-IやMDA5によっても認識され,広く研究で使用されている.細胞内におけるdsRNAの認識にはその長さが重要であると考えられている.TLR3を介したNF-κBの活性化には35~39 bpの長さのdsRNAが必要である12).一方,MDA5は300 bp以上の長いdsRNAを認識,結合し13–15),MDA5がCARDを露出するようにdsRNA上でフィラメント状に整列し,これにより下流のMAVSを活性化すると考えられている(図1)16).MDA5はウイルス複製の際に生じるdsRNAを認識し,特に(+)鎖一本鎖RNA(ssRNA)ウイルスの中でもピコルナウイルス科に属するウイルスの認識に重要である14).一方で,長鎖dsRNAを生じない(−)鎖ssRNAウイルスによってもMDA5が活性化されることが報告されている17).これに対し,RIG-Iは5′-三リン酸基を持つ比較的短鎖のdsRNAを認識する18–21).RIG-Iを介した認識に必要なdsRNAの長さは最短18~19 bpである20, 22).多くのウイルス感染において,このような5′-三リン酸RNAが細胞質内に蓄積され,細胞質内のRNAセンサーによって認識される.一方,核内で生成された自己のmRNAは,細胞質に輸送される前に5′-三リン酸が5′-キャップ修飾を受けることでRNAセンサーによる認識を受けない.これに加えて,RIG-Iはレオウイルスなどによって生成される5′-二リン酸修飾されたdsRNAを認識することができる23).poly(I : C)もまた5′-二リン酸修飾されたdsRNAを含み,短いpoly(I : C)はRIG-Iのリガンドとしても機能する23).このようにRIG-Iは5′末端の二リン酸あるいは三リン酸を認識することにより,細胞質内に存在する非自己のdsRNAを識別する.
TLR7とTLR8はssRNAを認識するRNAセンサーとして機能することが知られている.これらTLRによって認識されるRNAには,その配列にいくつかの特徴があることが知られている.たとえば,ポリウリジン(polyU),グアノシンやウリジンリッチ(GU-rich)配列などはTLR7やTLR8を強く活性化する24).しかしながら,これらpolyUやGU-rich配列が微生物において頻度の高い配列ではないため,なぜこれらのリガンドが自己と非自己の識別に重要であるかは明らかになっていない.最近の研究により,TLR7はグアノシン誘導体を認識することや,TLR8がウリジンやssRNA由来の短い分解産物と結合することが報告されている25, 26).
3)抗ウイルス活性を有するRNA結合タンパク質
TLRやRLRシグナルにより産生されたtype I IFNは,さまざまなIFN誘導遺伝子発現を誘導するが,その中にはさらにウイルスRNAに結合しウイルスタンパク質発現を抑制するタンパク質をコードするものも多く存在する.中でも,IFN-inducible dsRNA activated protein kinase, PKR(遺伝子名eukaryotic translation initiation factor 2-alpha kinase 2:Eif2ak2)は,最もよく研究されている抗ウイルス活性を有するRNA結合タンパク質の一つである.非感染細胞においてPKRは不活性型として低発現しているが,ウイルスの侵入によりPKRがdsRNAと結合すると二量体化,さらに自己リン酸化を起こし,eukaryotic translation initiation factor 2A(eIF2A)のリン酸化を介してmRNA翻訳を抑制する27).さらに,活性化したPKRは翻訳抑制やIRF3の活性化を介してマクロファージにおいてアポトーシスを誘導する.またPKRはウイルス感染によって形成されるストレス顆粒に局在し,RIG-Iを介したウイルス応答の制御に寄与する28).IFN-induced protein with tetratricopeptide repeats 1(IFIT1)もまた,キャップ構造依存的翻訳を抑制する29).IFIT1は2′-O-メチル修飾を欠如したmRNAを認識することで,自己のRNAとの識別を可能にしている.もう一つの例として,2′-5′オリゴアデニル酸合成酵素(2′-5′-oligoadenylate synthetase 1:OAS1)によるRNA認識機構が知られている(図1).この場合,OAS1がdsRNAを認識すると2′-5′オリゴアデニル酸が合成され,これがセカンドメッセンジャーとしてRNase Lを活性化しウイルスRNAを分解する30).adenosine deaminase acting on RNA 1(ADAR1)もまた,抗ウイルス活性を有することが報告されている.ADAR1はdsRNAのアデノシンをイノシンに変換させるRNA編集(A-to-I editing)を行うことにより,コドンに変異を挿入しウイルスタンパク質の機能を喪失させる,あるいはウイルスRNAの二次構造を変化させることにより,抗ウイルス活性を示すと考えられている31).また最近,ADAR1は宿主RNAを編集することで,MDA5による内因性dsRNA認識を抑制していることが明らかとなった32, 33).ADAR1のRNA編集活性を欠失したマウスではtype I IFNが過剰に産生されており,ADAR1は内因性dsRNAを編集しそのRNA構造を変化させることで,自己のdsRNAをRNAセンサーに認識されないようにコントロールしていると考えられる.ADAR1は,ヒトにおいてIFN過剰亢進により発症するAicardi-Goutières症候群の原因遺伝子の一つであり,RNA代謝の障害は抗ウイルス応答のみならず,内因性RNAに対する異常な応答やtype I interferonopathyと総称される疾患の発症に関わることが明らかとなってきている34, 35).
遺伝子発現制御は基本的に大きく転写制御と転写後制御に分けることができる.これら二つの制御機構が両輪をなし,遺伝子発現を迅速に誘導し,かつ適切に収束させることが可能となる.転写後制御機構におけるRNA代謝制御はスプライシング,核–細胞質輸送,翻訳や分解など多岐にわたる.免疫系における転写後制御に関しては,サイトカインをコードする多くのmRNAの半減期が短いことが知られている36).このことはmRNA分解に関わる転写後制御機構が大きく関与していることを示唆している.
mRNA分解は,エキソヌクレアーゼ分解経路とエンドヌクレアーゼ分解経路の二つのメカニズムで説明される.エキソヌクレアーゼを介した分解は,まずmRNAのポリA鎖を脱アデニル化することにより始まる.このポリA短鎖化は,脱アデニル化酵素であるPARNやCCR4-NOT複合体,あるいはPAN2, PAN3などが担っており,どの脱アデニル化酵素が機能するかはRNPの構成タンパク質によって決まる.脱アデニル化されたmRNAは,DCP2によってキャップ除去された後,5′-to-3′エキソヌクレアーゼ(XRN1)あるいはエクソソームによって,それぞれ5′→3′分解,3′→5′分解を受ける.エンドヌクレアーゼを介した分解経路は,SMG6やRegnase-1(5-2)項を参照)によってmRNAが切断された後,前述のようにXRN1やエクソソームを介してさらにmRNA分解が促進される.
4. AUリッチエレメントを介したmRNA分解機構
AUリッチエレメント(AU-rich element:ARE)を介したmRNA分解機構(ARE-mediated decay:AMD)は最もよく研究されている転写後制御機構の一つである.AREはmRNAの3′側非翻訳領域(3′ UTR)に存在するAUUUA配列に特徴づけられるシスエレメントであり,このようなAREを有するmRNAは細胞内に約9%存在するとされている37).AREを含むmRNA分解は脱アデニル化によって始まる38).その結果,不安定化したmRNAはエキソヌクレアーゼによって5′あるいは3′より分解される39).
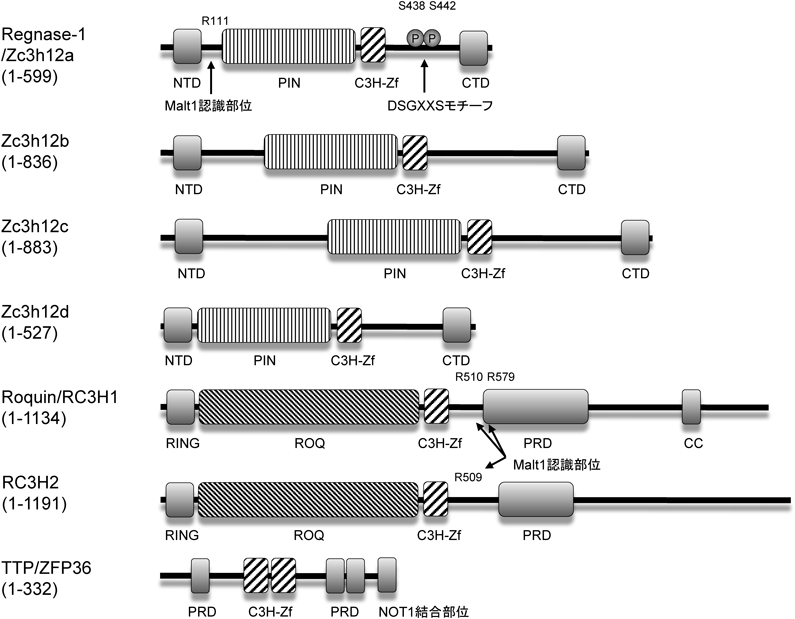
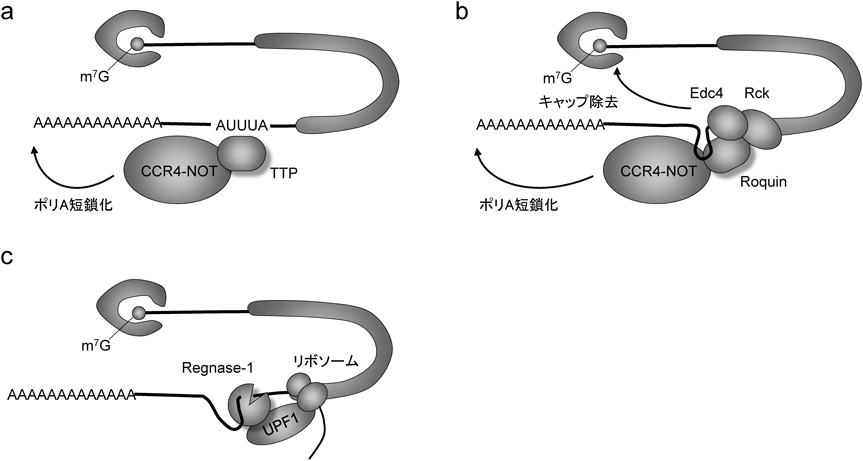
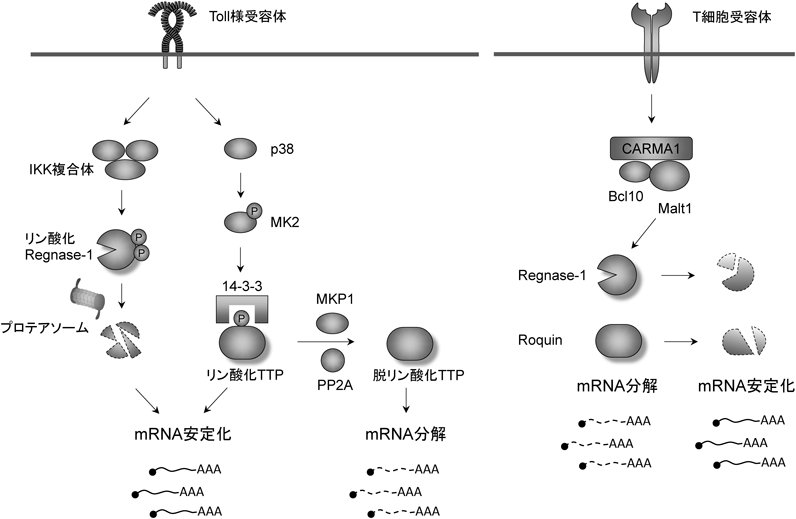
トリステトラプロリン(tristetraprolin:TTP,別名ZFP36)は最もよく研究されているARE結合タンパク質(ARE-BP)である40).TTPはCCCH型ジンクフィンガーを二つ有しており(図2),これによってTnfなどの標的mRNAの3′ UTRに存在するAREに結合する.TTP自体にmRNA分解能はなく,TTPはCCR4-CAF1-NOT脱アデニル化酵素複合体をリクルートし,標的mRNAの脱アデニル化を促進する(図3a)41, 42).これが引き金となり,標的mRNAはエクソソームによってさらに分解を受ける.TTPを遺伝的に欠損したマウスは関節炎を自然発症することが知られており,これにはTnf mRNAの安定化によるTNF過剰産生が一因であると考えられている43, 44).他にも,TTPはIl645–47),Csf248),Ifng49),Il1050)といった炎症に関わるmRNAを標的にすることが報告されている.TTPによるmRNA不安定化は,TTPタンパク質のリン酸化によって制御されることが知られている51, 52).すなわち,TLRシグナルが活性化すると,p38 mitogen-activated protein kinase(MAPK)によって活性化されたMAPK-activated protein kinase 2(MK2)によってTTPはリン酸化される.リン酸化されたTTPは14-3-3と結合することでCCR4-CAF1-NOT複合体との相互作用が阻害され,結果的にmRNAは安定化する.この過程は,MAPK phosphatase 1(MKP1)あるいはprotein phosphatase 2A(PP2A)によって脱リン酸化することで可逆的に制御されている.このように,TLRなどの炎症誘導シグナル経路による遺伝子発現は,RBPのタンパク質修飾を介して転写後制御と連動している.AUF1(別名hnRNPD)はmRNA不安定化を促進するARE-BPとして知られている.AUF1はp37, p40, p42, p45の四つのアイソフォームからなり,このうちp37が最もmRNA安定性に影響を与えるとされている53).AUF1欠損マウスは慢性的な炎症を引き起こし,またLPS(lipopolysaccharide)に対してより深刻なエンドトキシンショックを呈する54, 55).これは,TNFやIL-1βをコードするmRNA安定化によるサイトカインの過剰産生が原因と考えられている.AUF1同様,KH-type splicing regulatory protein(KSRP)もまたAMDに関連する因子であるが,詳細なメカニズムはよくわかっていない.プラズマ様樹状細胞においてKSRPはtype I IFNをコードするmRNAの制御に寄与することが示唆されている56).一方,HuR(別名ELAV1)のRNA分解に対する影響はより複雑である.マクロファージ特異的にHuRを欠損したマウスでは,LPS投与やデキストラン硫酸ナトリウム誘導腸炎においてより強い炎症応答を惹起する57).しかしながら,HuRは炎症性サイトカインをコードするmRNA分解を促進する一方で,そのような分解を抑制するという報告もあり,HuRの多様な機能性が示唆されている58–61).
1)Roquinを介したRNA認識と免疫制御
Roquinもまた,免疫機能に深く関与するCCCH型ジンクフィンガータンパク質である(図2).Roquinはジンクフィンガードメインの他に,RINGフィンガードメインとROQドメインを有している.RoquinはRc3h1とRc3h2で構成されるファミリーを形成しており,いずれも機能的ドメインが保存されている.結晶構造解析によりROQドメインがRNAとの結合に重要であることが示されている62, 63).RoquinによるRNA分解は,標的mRNAの3′ UTRに存在するconstitutive decay element(CDE)を介して起こる64).CDEは生物種間で保存された配列であり,構造的にステムループを形成している.Roquinを介したmRNA分解経路にはいくつかの報告があるが,現在のところ,TTPと同様,CCR4-CAF1-NOT複合体をリクルートし脱アデニル化を促進すると考えられている(図3b)64).また別の分解経路として,RoquinはEdc4およびRckと会合しキャップ除去を介してmRNA分解を促進するとの報告もある65).Roquinの1塩基変異マウスであるSanroqueマウスは,濾胞T細胞の増加を特徴とする自己免疫疾患を発症する66).RoquinとRoquin2をT細胞特異的に欠損したマウスはSanroqueマウスと同様の表現型を示す67, 68).このことから,これら二つのRoquinタンパク質は補完的に機能していると考えられている.T細胞では共刺激分子ICOSがRoquinの標的mRNAとして知られており,Sanroqueマウスにおける濾胞T細胞分化促進に寄与していると考えられている69).Roquin欠損マクロファージにおいても,TLR刺激に対してTnf mRNAの安定化が認められ,TNF産生が亢進する64).
2)Regnase-1を介したRNA認識と免疫制御
Regnase-1(別名Zc3h12a/MCPIP1)はCCCH型ジンクフィンガードメインとそのN末端側にRNA分解酵素として機能するPilT N-terminus like(PIN)ドメインを持つ65 kDaの細胞質タンパク質である(図2).Regnase-1はZc3h12b, Zc3h12c, Zc3h12dとファミリーを形成しており,いずれもジンクフィンガードメインおよびPINドメインを持つ70).注目すべきことに,ヒトゲノム上に存在するCCCH型ジンクフィンガータンパク質のうち,Regnase-1のようにヌクレアーゼ活性を有するものは,現時点においてZc3h12ファミリーに属する遺伝子のみである71).Regnase-1は,ジンクフィンガードメインとPINドメインを介して標的RNAと結合し分解することでRNA安定性を制御している.
Regnase-1によるRNA分解は3′ UTRに存在するステムループ構造を介して起こる(図3).このことは,Regnase-1はステムループ構造を分解コードとして認識していることを意味する.実際に,ゲノムワイドcross-linking immunoprecipitation(CLIP)シークエンス解析により,Regnase-1結合塩基配列は有意にステムループ構造を形成しうることが明らかとなった72).さらにこのステムループ構造の配列を詳細に検討した結果,Regnase-1結合ステムループ構造のループ部分にはピリミジン–プリン–ピリミジン配列を有する頻度が高いことが解明された.では,Regnase-1を介したmRNA分解はどのようにして起こるのか? Regnase-1は主に小胞体(endoplasmic reticulum:ER)あるいは細胞質内に局在し,リボソームタンパク質と相互作用している.これを反映して,Regnase-1は翻訳が起こっているmRNAを分解しており,さらに翻訳が終止コドンで終結することが3′ UTRに存在するステムループ構造の認識を可能にする.興味深いことに,翻訳終結の際,Regnase-1はATP依存的RNAヘリカーゼupframeshift 1(UPF1)と会合する72).さらに,UPF1のヘリカーゼ活性はRegnase-1を介したmRNA分解に必須であることから,RNAの二次構造の変化がRegnase-1によるRNA分解に何らかの影響を与えていることが示唆される.UPF1はナンセンス変異依存性mRNA分解機構(nonsense-mediated decay:NMD)に重要な因子であることが知られており,RNA品質管理機構の中心的役割を果たしている73).NMDは中途終止コドン(premature termination codon:PTC)を認識することで翻訳中のmRNAを分解するとされており,この過程にもUPF1ヘリカーゼ活性が必要であることがわかっている.Regnase-1を介したmRNA分解には,ステムループ構造が終止コドンから少なくとも20塩基下流に離れていることが必要であることから72),正常な翻訳終結とPTCで起こる異常な翻訳終結の間にUPF1を介した何らかの識別機構の存在が示唆される.
3)Regnase-1欠損マウスの表現型
Regnase-1はユビキタスに発現しているが,特に免疫細胞においてその発現が高い.Regnase-1を遺伝的に欠損したマウスはメンデルの法則に従い生まれるが,生後8週齢までに個体死に至る74, 75).脾腫やリンパ節腫大は著しく,特に肺や肝臓に炎症細胞浸潤が著しい.血液中にはさまざまなサブタイプの免疫グロブリンが上昇しており,興味深いことに抗核抗体や抗dsDNA抗体といった自己抗体の出現が認められる.その他,発育遅延や脂肪組織の萎縮,さらに十二指腸での鉄吸収障害による重度の貧血など多彩な表現型を呈する.Regnase-1欠損マクロファージはTLRリガンドに対してIL-6やIL-12p40といった炎症性サイトカインを多く産生する.しかしながら,TNFもまたRegnase-1の標的mRNAであるにもかかわらず,Regnase-1欠損マクロファージにおけるTNF産生には影響は認められない74).このことから,炎症性サイトカイン制御における何らかの特異的な認識機構が存在していることが示唆される.一方,過剰な抗体産生の結果と一致して,獲得免疫系の活性化も認められる.T細胞はほとんどがエフェクター・メモリー細胞に変化し,IFNγなどのサイトカインの産生増加を認める.B細胞もまた,その多くがクラススイッチ組換えを起こし抗体産生細胞である形質細胞への分化が認められる.これらの表現型を説明する上で,Il6やIl12bに加えて,Il1b, Il2などのサイトカインや,Icos, Tnfsf4といった共刺激分子,さらにRel, Cebpb, Nfkbiz, Nfkbidなどの転写因子および転写調節因子をコードするmRNAもRegnase-1によって分解されることが明らかとなっている76–78).以上のことから,Regnase-1はさまざまな細胞種において免疫応答を負に制御し,個体レベルにおいて自己免疫疾患の発症を抑制していることが示唆される.
4)Regnase-1の発現制御機構
Regnase-1はその発現自体も厳密にコントロールされている.マクロファージがLPSなどのTLRリガンドの刺激を受けると,Regnase-1タンパク質は速やかに分解される79).Regnase-1は,IκB kinase(IKK)によるリン酸化標的配列として知られているDSGXXSモチーフを持ち,TLRシグナルにより活性化したIKK複合体により435番と439番のセリン残基がリン酸化され,これによりβTrCPによるユビキチン化を促進しプロテアソーム分解系によって分解される(図4).このようなIKK複合体によるリン酸化を介したRegnase-1タンパク質の分解はTNF刺激では起こらないことから,シグナル依存的な要因が影響しているものと考えられる.実際に,DSGXXSモチーフのセリン残基をアミノ酸置換した変異Regnase-1タンパク質は分解を受けないことが確認されているが,このモチーフ以外にもRegnase-1タンパク質のリン酸化が確認されており,分解シグナルではないリン酸化の存在が示唆される.
Regnase-1タンパク質はリン酸化非依存的な分解も受ける.これにはシステインプロテアーゼであるMalt1が関与する80).この場合,T細胞受容体(TCR)の下流においてCARMA1, Bcl10, Malt1による複合体が形成され,Malt1が直接Regnase-1タンパク質の111番目のアルギニン残基を認識し切断する76).Malt1が関与するシグナルは他にも,ミエロイド系細胞に発現するDectin-1やNK細胞に発現するNKG2D,あるいはGタンパク質共役型受容体などがあり81),シグナル依存的にRegnase-1タンパク質の発現が制御されているものと考えられる.興味深いことに,RoquinもまたMalt1によって分解されることが報告されている(図4)77).Malt1はシグナル伝達分子として働きNF-κB転写活性化に重要であることが知られているが,同時に,プロテアーゼとして機能し,Regnase-1やRoquinといったRBPの発現を調節することで転写後制御にも関与すると考えられる.Malt1のプロテアーゼ活性を不活化したC472A変異マウスでは,実際にこれらRBPの分解が認められず,T細胞やその他免疫細胞の活性化が障害される.このようにMalt1プロテアーゼ活性は免疫細胞の活性化に必要であるにもかかわらず,このマウスは自己免疫性胃炎を発症する82–84).Malt1プロテアーゼ活性は制御性T細胞の分化にも影響を及ぼすことから,制御性T細胞の分化障害が免疫寛容破綻の原因であると考えられている.
一方,Regnase-1の発現はmRNAレベルでも厳密に調節されている79).Regnase-1 mRNAはTLRあるいはTCR刺激によって迅速に誘導される.興味深いことに,Regnase-1 mRNAの3′ UTRにはRegnase-1認識配列が存在する.つまり,Regnase-1は自身の3′ UTRを介して,発現を負に制御するフィードバック機構が存在する.これにより,過剰なRegnase-1タンパク質の発現を抑制している.以上のことから,免疫細胞における遺伝子発現制御にはRegnase-1によるmRNA安定性制御が密接に関与しており,その背景にはRegnase-1タンパク質およびmRNAのダイナミックな発現調節が存在する.
細胞内においてRNAは免疫機能にさまざまなステップで影響を与えている.TLRやRLRなどのRNAセンサーが抗ウイルス応答に重要であることは本文でも述べてきたが,自己のRNAに対するRNAセンサーの過剰応答が生体に与える悪影響に関しての認識も高まりつつある.一方で,RNA制御の根幹をなすRNA結合タンパクの機能制御に関する報告も以前より散見されてはいるが,いまだ全容解明には至っていない.TTP, RoquinやRegnase-1は免疫機能に影響を与える代表的なCCCH型ジンクフィンガータンパク質として認識されているが,ゲノム上には約60個のCCCH型ジンクフィンガータンパク質をコードする遺伝子が存在しており71),その多くはRNA制御機構および生物学的意義について明らかになっていない.さらに,プロテオーム解析によりRBPレパトアには,これまで知られているRNA結合モチーフ以外にも,in silicoでは同定できなかった非典型的RNA結合モチーフを持つものも少なからず存在していることが明らかになっている.今後,新しいRNA制御を介した免疫制御機構の解明が期待される.