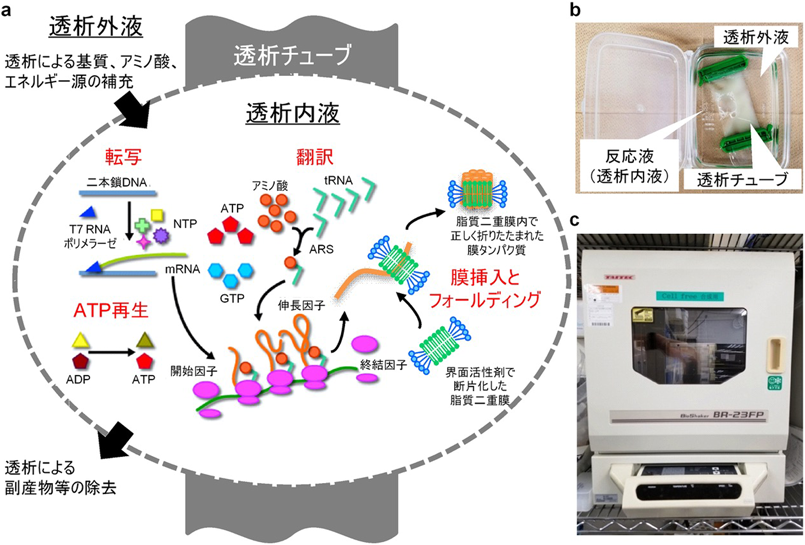
立体構造解析のための大腸菌無細胞タンパク質合成系を用いた哺乳類由来膜タンパク質調製手法Preparation techniques using an Escherichia coli cell-free system to produce membrane proteins for structural analysis
理化学研究所生命機能科学研究センタータンパク質機能・構造研究チームLaboratory for Protein Functional and Structural Biology, RIKEN Center for Biosystems Dynamics Research ◇ 〒230–0045 神奈川県横浜市鶴見区末広町1–7–22 ◇ 1–7–22 Suehiro-cho, Tsurumi-ku, Yokohama, Kanagawa 230–0045, Japan