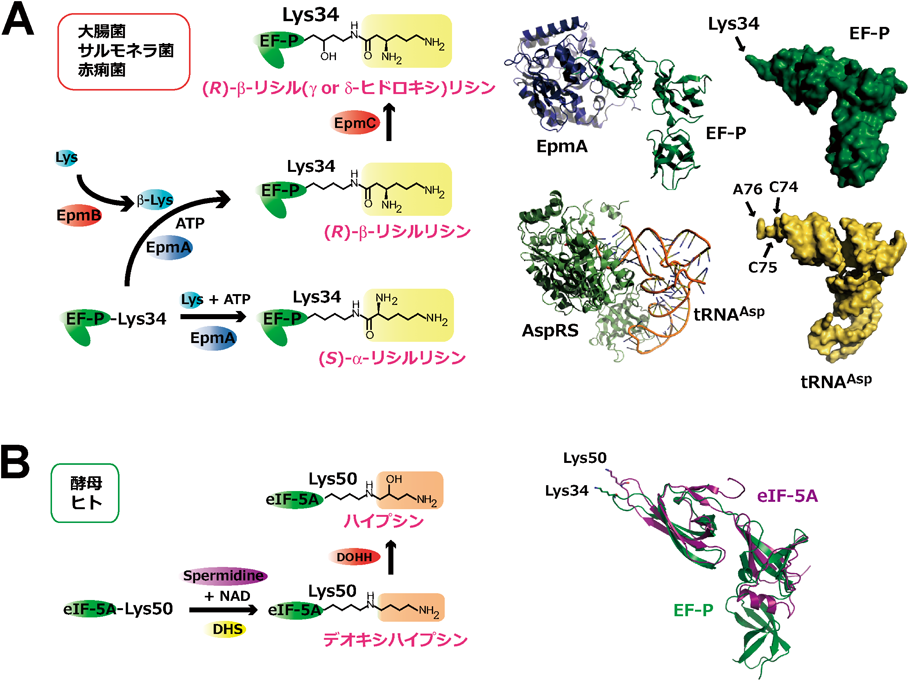
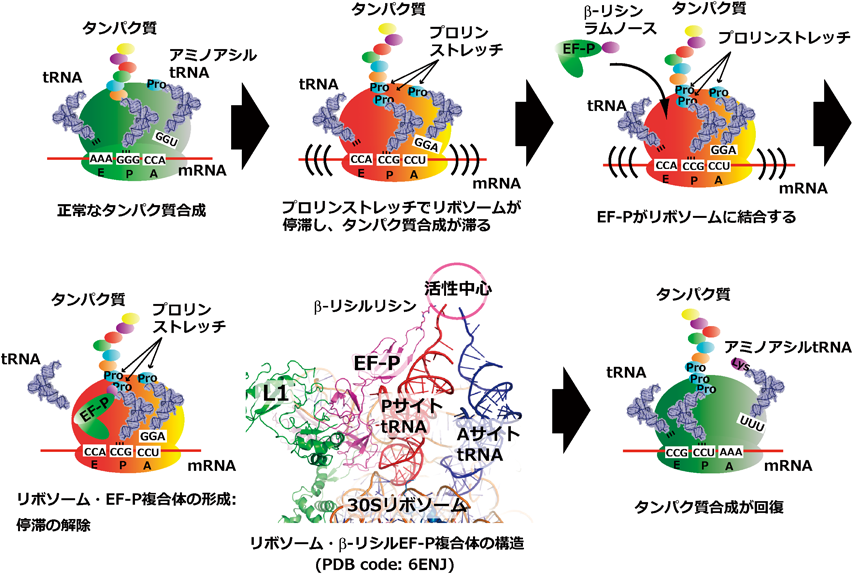
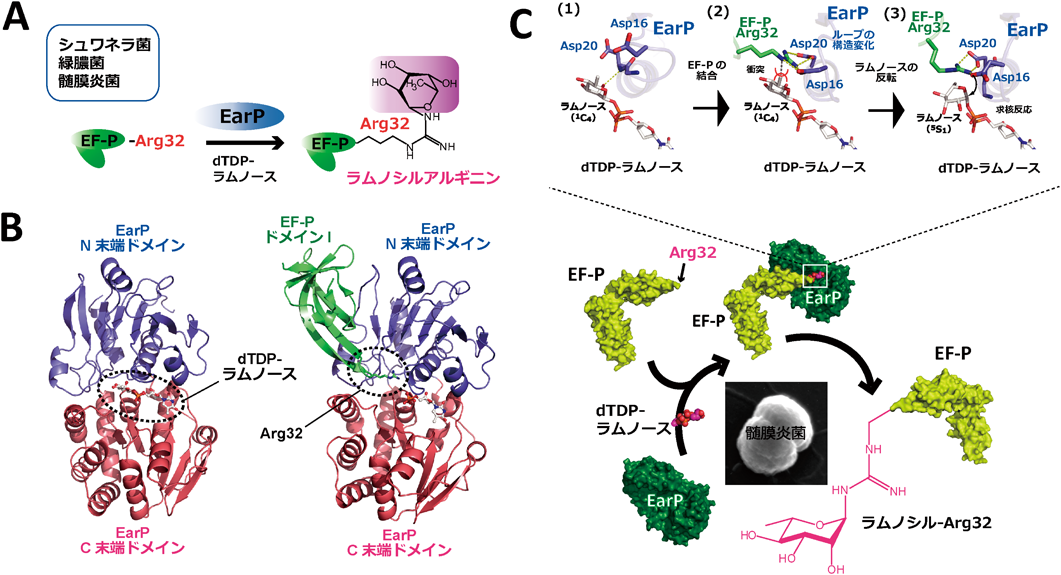
翻訳因子EF-Pのアミノアシル化とラムノシル化Post-translational modifications of translation factor EF-P
1 理化学研究所・横山構造生物学研究室RIKEN Structural Biology Laboratory ◇ 〒230–0045 神奈川県横浜市鶴見区末広町1–7–22 ◇ Suehiro-cho, 1–7–22 Tsurumi, Yokohama, Kanagawa 230–0045, Japan
2 理化学研究所・生命機能科学研究センター・非天然型アミノ酸技術研究チームRIKEN Center for Biosystems Dynamics Research ◇ 〒230–0045 神奈川県横浜市鶴見区末広町1–7–22 ◇ RIKEN Structural Biology Laboratory, Suehiro-cho, 1–7–22 Tsurumi, Yokohama, Kanagawa 230–0045, Japan