1940~50年代に,HodgkinとHuxleyがイカの巨大軸索を用い,細胞膜上には膜電位が変化すると開いてNa+やK+を透過させる“孔”が存在し,そのイオンの流出入が活動電位を生むことを示した1).以来,我々は電位依存性Na+チャネル(NaVチャネル)をNa+が通る孔の本体,すなわち脳機能を支える神経インパルスの核心分子であると理解するに至っている.Hartshorneらがラットの脳からNaVの精製に成功し2, 3),さらに野田らが電気ウナギの電気器官からNaVチャネルの孔の本体(αサブユニット)のcDNAをクローニングした4).ショウジョウバエでは,paraと呼ばれるNaVの突然変異体が分離され,NaVの遺伝子型と表現型の関係が明らかにされた5).ホヤにおいては,神経誘導シグナルに応じたNaV遺伝子の発現が,ニューロンにおける膜興奮性の出現に本質的に関わることが示された6).近年,海産の細菌が持つNaVチャネルについて,Na+選択性や膜電位依存的な開孔機構の理解が得られ7, 8),またついには,動物のNaVチャネルの立体構造が,ゴキブリ,電気ウナギ,ヒトのものについて報告された9–11).
これらの解析が多様な生物のNaVを用いてきたがゆえに,NaVのアミノ酸配列が歴史的に変化し,そしてその変化が生物の進化に歩調を合わせて起こってきたことも明らかになった.他の遺伝子ファミリーと同様に,NaVの遺伝子ファミリーも重複,変異,選択を経て成長してきた.これらによっていまや動物たちは,異なる発生段階,異なる細胞種,さらには異なる細胞内コンパートメントにおいて,似て非なるNaVを利用できる.本稿で我々は,哺乳類,脊椎動物,脊索動物,そして後生動物のNaVチャネルの進化と多様化の歴史を概説する.NaVの分子進化イベントのいくつかは,動物の生活様式の進化をよく反映していることがわかる12).
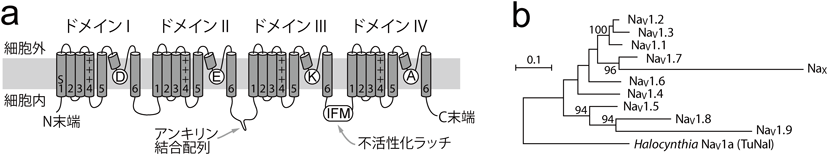
動物のNaVのαサブユニットは,四つの互いに相同なドメインI~IVが縦列した構造を持つ(図1).IからIVの各ドメインは,αヘリックスからなる膜貫通セグメントを六つ含んでいるので(S1~S6),結果,動物のNaVは約2000アミノ酸からなる全長の中に,計24回の膜貫通領域を持つ.各ドメインのS1~S6は,電位依存性K+チャネル(KVチャネル)と構造的によく対応しており,KVと同様にS1~S4は膜電位センサーとして,S5~S6はポア形成領域として機能する13).特に各S4セグメントには,3残基おきに正電荷を持ったArgないしLysが並び,これらが膜電位の感知に本質的な役割を担う.これらS4の塩基性アミノ酸は,膜内にありつつS1~S3の酸性アミノ酸と塩橋を形成しており,膜電位が脱分極するとS4は塩橋の相手を変えながら細胞外側に向けて動いていく.このS4とS1~S3との間の歯車のような動きにより,チャネルのゲートが開かれる9–11).各ドメインのS5~S6セグメントとそれらの間のP-loopと呼ばれる領域は,折りたたまれたチャネルタンパク質の中央部を占め,チャネルの内壁,ゲート,イオン選択性フィルターを形成する9–11, 13).
哺乳類は,NaV1.1から1.9まで九つのNaVアイソフォームを持つ.加えて,NaXという,NaV1.7に似るが電位依存性Na+チャネルというより,細胞外Na+濃度センサーとして働く因子も知られる14, 15).これらのcDNAとアミノ酸配列,遺伝子(SCN1A~11A)の染色体上の位置,発現パターン,テトロドトキシン(TTX)等の毒物に対する感受性,シングルチャネル・コンダクタンス,不活性化の特徴,生理機能に関して行われてきた包括的な解析結果の一部を表1にまとめる16).たとえば,哺乳類のNaV1.1~1.3の遺伝子は,染色体上でHoxD遺伝子クラスターの近くに縦列している(表1).これら三つの一次構造は互いによく似ており,これらが進化の過程で比較的最近,縦列重複(tandem duplication)により生じたことを示す(図1b).このクラスターはさらに,NaV1.7とNaXの遺伝子も含む.NaV1.7は末梢神経系(peripheral nervous system:PNS)の背根神経節(dorsal root ganglia:DRG)ニューロンに多く発現する16).NaV1.1~1.3, 1.7はみな,IC50=10 nMというきわめて高い感受性でTTXに反応する16, 17).中枢神経系(central nervous system:CNS)には,NaV1.1~1.3に加えてNaV1.6も発現するが,これは他のアイソフォームと異なり持続性電流を発する特徴的なチャネルである.他方,PNSの感覚ニューロンではNaV1.7に加えて,特に軸索が細く伝達速度が遅いもの(C繊維)においてNaV1.8や1.9が発現している16).筋肉組織のうち,心臓はNaV1.5を,骨格筋はNaV1.4を発現し,それぞれの組織で活動電位を発するのに活用している.心臓に発現するNaV1.5は,NaV1.8とNaV1.9の遺伝子とともに,HoxAクラスターの近傍で縦列している(表1).これらNaV1.5, 1.8, 1.9はみな相対的にTTXへの感受性が低く,IC50は1 μM以上である16, 17).対して骨格筋のNaV1.4は,CNSのNaV1.1~1.3, 1.6と同程度に,TTXに高い感受性を示す16, 17).NaV1.4とNaV1.6をコードする遺伝子は他のNaV遺伝子と縦列しておらず,それぞれ単独でHoxBおよびHoxCクラスターの近傍に位置する(表1).
表1 哺乳類の電位依存性Na+チャネルαサブユニット16)| チャネル名 | 遺伝子名 | 隣接Hoxクラスター | TTXのIC50 | 主な発現部位 |
|---|
| NaV1.1 | SCN1A | HoxD | 10 nM | CNS |
| NaV1.2 | SCN2A | HoxD | 10 nM | CNS |
| NaV1.3 | SCN3A | HoxD | 10 nM | CNS |
| NaV1.4 | SCN4A | HoxB | 10 nM | 骨格筋 |
| NaV1.5 | SCN5A | HoxA | 1~10 μM | 心臓 |
| NaV1.6 | SCN8A | HoxC | <10 nM | CNS |
| NaV1.7 | SCN9A | HoxD | 10 nM | PNS |
| NaV1.8 | SCN10A | HoxA | >10 μM | PNS |
| NaV1.9 | SCN11A | HoxA | 1 μM | PNS |
| NaX | SCN7A | HoxD | — | CNS他 |
| TTX:テトロドトキシン,CNS:中枢神経系,PNS:末梢神経系. |
これら九つすべてのNaVが,膜の脱分極に応じて開く活性化ゲートを持ち,Na+を選択的に透過する.一般に,生きた細胞の細胞質ではNa+濃度が細胞外液の1/10程度に抑えられている.NaVチャネルの開孔により,Na+は細胞内に流れ込み,この正電荷を持ったイオンの流入により,膜は脱分極する.多くのNaVチャネルは不活性化の機構を備えており,開孔後,Na+の透過は速やかに遮断される.この不活性化は,NaVのαサブユニット自体が持つ機能であり,これにより膜の再分極が早まり,活動電位はより鋭くなる1, 11, 18).哺乳類の九つのNaVはすべて,多かれ少なかれ不活性化能を示し,それには“不活性化ボール”ないし“不活性化ラッチ”と呼ばれるドメインIII~IV間のリンカー配列を必要とする11, 18)(図1a).この不活性化ラッチは,開いたゲートにフィットする構造を持ち,ゲートが開くとこれがすばやく潜り込んでNa+の透過をブロックすると考えられている(ball-and-chain model)11, 19)(図1a).このリンカー配列には,Ile-Phe-Met(I-F-M)という三つの連続する疎水性アミノ酸がよく保存され,実際に,特に中心のPhe残基は不活性化に必須のサイトである19).
IからIVの各ドメインに存在するP-loopは,イオンの流路に突出し,このループの内側の頂点部分がイオン選択性フィルターを構成する9–11, 20).このNa+選択性に関わるアミノ酸残基は,ドメインIのAsp(D),ドメインIIのGlu(E),ドメインIIIのLys(K),そしてドメインIVのAla(A)からなる(D/E/K/A型ポアシグニチャー)(図1a).対照的に,多くの電位依存性Ca2+チャネル(CaVチャネル)は,NaVに相同な24回膜貫通構造を備えるが,E/E/E/Eというポアシグニチャーを持ち,これがCa2+選択性を与える.またこのNa+選択性フィルターのあたり,特にドメインIのシグニチャー残基の周辺が,TTXの結合ドメインだと考えられている21).このイオン選択性フィルターは流路の中央部に位置し,この細胞質側寄りに,電荷を持ったアミノ酸に囲まれた空所(central cavity)がある.流路の壁は主に各ドメイン由来のS6が作り,これが活性化ゲート自体を構成している9–11, 13).
以上の一般的特徴,すなわち6セグメント×4ドメインの構成を持つ24回膜貫通型の構造,S4を中心とした膜電位センサー,D/E/K/A型の選択性フィルター,活性化ゲート,不活性化ラッチ,それらは哺乳類のすべてのNaVチャネルのαサブユニットに共通して備わっている11, 13, 16).
細菌もNaVチャネルを持ち,それらはBacNaVと呼ばれる7, 8, 22).BacNaVの発見は,膜電位の変化に応じて開孔するNa+チャネルが,原核生物においてすでに進化していたことを確証した.しかしこれらBacNaVは,非常にコンパクトなS1~S6セグメントを持つ6回膜貫通型のサブユニットが,ホモ四量体を形成してできており,動物の24回膜貫通型のNaVチャネルとは大きく構造が異なる.あるBacNaVに単純な変異を導入すると,カルシウム選択的なチャネルに変わるので,原核生物は電位依存性Ca2+チャネルも持っている可能性が高い23).加えて,結晶構造が最初に解かれた電位依存性イオンチャネルであるKVAPは,古細菌のK+チャネルである24).したがって原核生物は,膜電位で開孔するイオン種特異的な6回膜貫通型チャネルをさまざまに,真核生物の出現のはるか以前から利用してきたといえる.
この6回膜貫通型チャネルは,機能的に膜電位センサーを構成するS1~S4ユニットと,チャネルの孔を構成するS5~S6ユニットに分けられる.原核生物のKcsAや真核生物のKirファミリーの因子はどちらもK+チャネルだが,2回膜貫通型のサブユニットがホモ四量体を形成してできる.これらはいわば,6回膜貫通型チャネルの膜電位センサーユニットがなく,S5~S6の“ポア形成領域”だけが四つ会合する形で成り立っているものである25).K2Pチャネルファミリーのタンパク質は,二つのポアドメインが縦列重複した形態を示し,これが二量体化することで偽四放射対称のチャネルポアを形成する26).最近の研究は,膜電位センサー領域もまた,ポアドメインとは独立に機能しうることを明らかにしている.たとえば,電位依存性脱リン酸化酵素(voltage-sensing phosphatase:VSP)はS1~S4に似た膜電位センサードメインがホスファターゼドメインと連結しており,これが膜電位変化に応じて膜のリン脂質の脱リン酸化を触媒する27).さらにはHV1/voltage-sensor-domain-only-proteinと呼ばれる電位依存性H+チャネルは,S1~S4の膜電位センサー領域に対応する配列のみからなる28, 29).
原核生物でこれまで同定された電位依存性チャネルはすべて,6回膜貫通型サブユニットのホモ四量体により形成される.真核生物のKVチャネルやCa2+透過性を持つCatSperチャネルも,6回膜貫通型サブユニットがそれぞれホモあるいはヘテロ四量体を形成してできる30).これらに加えて真核生物には,12回あるいは24回膜貫通型の,より大きな電位依存性チャネルタンパク質が存在している.12回膜貫通型のサブユニットからなるチャネルにはtwo-poreチャネル(TPCs)が知られ,これらはエンドソームやリソソームといった細胞内膜系で機能する陽イオンチャネルとされる31).24回膜貫通型のイオンチャネルは,NaVばかりでなく,L型,N/P/Q/R型,T型のCaVチャネル(それぞれCaV1, CaV2, CaV3と呼ばれる),さらに陽イオン・リークチャネル(NALCN)を含む大きなタンパク質ファミリーを形成している.真核生物の24回膜貫通型チャネルはみな,相同なドメインが4回縦列した構成を持ち,これはもともと6回膜貫通型の因子の遺伝子が,2回の縦列重複を行って生じたと考えられる.実際に,NaVやCaVのドメインIのアミノ酸配列は,ドメインIIやIVよりIIIに類似し,ドメインIIの配列はIやIIIよりIVに似る30, 32).また,これらのドメインI~IVの配列は,BacNaVやKVのものよりもCatSperの配列に,さらにいえば,CatSperの配列よりも互いの間でより高い類似性を示す.すなわち,真核生物の24回膜貫通型チャネルは,BacNaVの重複に由来するのではなく,CatSper様の6回膜貫通型タンパク質が2回,縦列重複して生じたと推定される30).この縦列重複は,ドメインIからIVの中にそれぞれ独立の“回転非対称な”変異の蓄積を可能にした点で,大きな意味があったと考えられる.すなわち後に,イオン選択性フィルターにおけるD/E/K/Aという回転非対称なアミノ酸使用により,Na+選択性が達成された.また以下でみていくように,速い不活性化やアンカーリング機能などNaVに新たに導入されたイノベーションも,回転非対称な変異にもとづいて生まれたものである.
上記に加えてもう1種類,NaV2と呼ばれる24回膜貫通型チャネルが多くの無脊椎動物で同定されている.この因子は哺乳類のNaV1に似たアミノ酸配列を持つが,ポアシグニチャーがD/E/K/AではなくD/E/E/Aである33–40).近年になって,このサブファミリーのチャネルは,Na+よりもCa2+を透過することが明らかになり,CaV4と呼ぶのがよいと提案されている36, 40).これら,通常のNaV1との配列類似性と,Ca2+を通すという特徴の両方を考え,本稿ではこのサブファミリーをNaV2(CaV4)と呼ぶことにする.
24回膜貫通型のNaV,CaV,NALCNファミリーの進化的な分岐は,多細胞動物(後生動物)の起源に先立つものである(図2a).NaV1/NaV2(CaV4)のクレードとCaV1/2やCaV3のクレードとの分岐は,動物の系統と,後生動物に最も近縁な原生生物とされる襟鞭毛虫の系統との分岐に先立つ37, 39, 41).NALCN様の配列は菌類にも見いだされるので,NALCNファミリーの起源は,動物と襟鞭毛虫と菌類を含むグループであるオピストコンタ(Opisthokonta)の祖先までさかのぼることになる41).図2aは実際に,互いに系統的に遠く隔たった単細胞の真核生物である酵母[Saccharomyces(菌)],ゾウリムシ[Paramecium(繊毛虫)],クラミドモナス[Chlamydomonas(緑藻)]がともに,異なるタイプの24回膜貫通型チャネルを持つことを示している.この系統樹は,NALCN, NaV, CaVファミリーを分ける遺伝子重複は,ユニコンタ[Unikonta(動物,菌,アメーボゾアを含む)]とバイコンタ[Bikonta(陸上植物,藻類,繊毛虫などを含む)]の分岐以前,すなわち真核生物の起源近く,あるいはそれ以前にさかのぼることを示唆する42, 43).
他方で,この系統樹は,多くの真核生物のグループが特定の因子を持っていないことも示している.それらはおそらく二次的な喪失によるもので,BrunetとArendtはこのNaV/CaVチャネルの喪失が,それらの生物における鞭毛・繊毛の非存在と関連していることを指摘している44).実際,ゾウリムシとクラミドモナスのCaVチャネルは繊毛/鞭毛の膜に局在し,活動電位を生み出して繊毛打/鞭毛打の波形を変化させる45, 46).24回膜貫通型CaV/NaVチャネルの繊毛・鞭毛への局在は,後のニューロンの出現への前適応になったと考えられる44).
哺乳類でみられるような各サブファミリーに特異的なポアシグニチャー,すなわちNaV1のD/E/K/A, CaV1/2のE/E/E/E, CaV3のE/E/D/D, NALCNのE/E/K/Eというモチーフは,比較的最近確立した41, 47)(図2b).たとえば,襟鞭毛虫類で同定されたNaV1/NaV2(CaV4)チャネルはD/E/E/A型のポアシグニチャーを持つ.また後生動物のうち左右相称動物の成立以前に分岐したクシクラゲ(有櫛動物)やセンモウヒラムシ(板形動物)のNaV1/NaV2(CaV4)もD/E/E/A型であったので,NaV2(CaV4)が持つD/E/E/A型が祖先型で,NaV1のD/E/K/Aというモチーフは,左右相称動物の祖先でできたものと考えられる37–39, 47).左右相称動物の成立以前に分岐したもう一つのグループである刺胞動物(クラゲ,イソギンチャク)からは,NaVアイソフォームが五つ見つかっており,興味深いことに,このうち三つはD/E/E/A,一つはD/E/E/T,もう一つはD/K/E/Aというポアシグニチャーを持っていた38, 39, 48).後者二つはD/E/E/A型から派生したもので(図3),特に刺胞動物のD/K/E/A型のチャネル(NaV2.5)はおそらくNa+透過型であり興味深い.これは左右相称動物におけるD/E/K/A型のNaV1チャネルの起源とは独立に,Na+選択性を平行進化させたものと考えられる38, 39, 48).対して,刺胞動物のNALCN様の遺伝子は共通してE/E/E/Eというポアシグニチャーをコードし,これはCa2+選択性を示すと思われる.左右相称動物のNALCNは,おしなべてE/E/K/E型のポアシグニチャーを持つので,この変化によりこのチャネルは陽イオンを非選択的に通すようになったと考えられる41).
以上から,多細胞動物はその起源において,二ないし三つのCaVサブファミリーの遺伝子と,一つのNaV1/NaV2(CaV4)サブファミリーの遺伝子(ただしD/E/E/A型),そして一つのNALCN様の遺伝子を持っていたと予想できる39, 49).興味深いことに,これらの中に実はNa+を選択的に透過するものはなく,むしろこれらはCa2+を優先して透過するチャネルだった.動物は,電位依存性Na+チャネルを持たずに世に生まれた.そして真のNaVチャネル,すなわちD/E/K/A型のNaV1チャネルが進化したのは,左右相称動物の祖先においてであった.
左右相称動物の系統は,左右対称なボディプランを備えた動物から構成される.この動物グループは,三つのCaV(CaV1~3),二つのNaV1/NaV2(CaV4),そして一つのNALCNという計6種類の24回膜貫通型チャネルを基本レパートリーとして備えていた.このうち,NaV1/NaV2(CaV4)のサブファミリーの二つの因子こそNaV1とNaV2(CaV4)であり,NaV1チャネルはまさに,左右相称動物の共通祖先に生まれた39, 47)(図3).このNaV1チャネルはD/E/K/A型のポアシグニチャーを備え,これにより左右相称動物は,適応してきた環境に最も豊富に存在する陽イオンであるNa+を,膜興奮性の制御に特異的に利用できるようになった.NaV1は,Na+ポンプ(Na,K-ATPase)が作った細胞膜内外の大きな濃度差を利用した“速い反応性”(自身が出現する以前には達成しえなかったほどに速い)を興奮性細胞に付与することに成功しただろう.NaV1はまた,Ca2+が誘起する多様な細胞内プロセスを直接刺激することなく,電気シグナルを生み出すことができる点でも重要だっただろう.
D/E/K/A型のポアシグニチャーに加えてこのとき,ドメインIIIとIVの間のループに不活性化ラッチが導入された可能性がある(図3b).Na+の選択的利用により高速の脱分極が可能になった代わりに,不活性化ラッチがチャネルの開孔をごく一過的にすることで,細胞内イオン環境の変化を最小にし,膜電位を静止状態にいち早く引き戻させる.また,膜が再分極して不活性化ラッチが孔から外れない限り再びチャネルは開孔しないという状態,すなわち膜興奮の“不応期”が出現し,これが神経シグナルを一方向に流す仕組みの基礎となった.そしてこの不応期の出現こそが,現在,多くの動物が採用するニューロンの連続的な発火,すなわち活動電位の頻度に神経シグナルの強度を符号化する原理をもたらした.
左右相称動物の進化的起源と多様化は,いわゆる「カンブリア爆発」と呼ばれる地史イベントと対応している.つまりNaV1の起源は,この動物の爆発的多様化と時を同じくしているのである.古生物学者は,いわゆる“食う−食われる”の関係がカンブリア紀に確立したことを示唆している.この関係が体のサイズと複雑さの増加をもたらし,感覚器を洗練させ,運動を加速させ,CNSの高度化を導いた50–52).Parkerによる仮説は,特に眼の発達がこの爆発のカギとなったとしている52).NaV1により可能になった「新型」の神経インパルス,すなわち“Ca2+シグナルとは独立に働き,速く,一方向的で,頻度にコード化できる”新しいインパルスが,それが出現する以前よりも大きな体の出現を可能にし,感覚システムの運動システムからの分離,ひいては中枢神経系の発達を促したと考えられる.捕食者が被食者を,被食者が捕食者を見逃さないために,互いの感覚処理システムと運動制御システムが競争的に拡大し,精緻化していった.それはまさに,NaV1の起源と進化に支えられたものだったと想像できる.
NaV1の出現は,NaV2(CaV4)をともに生み出した遺伝子重複によっている(図3a).結果,大部分の左右相称動物は,NaV1とNaV2(CaV4)という二つのNaVサブタイプを保持している.たとえばショウジョウバエのゲノムには,NaV1をコードするparaと,NaV2(CaV4)をコードするDSC1がある5, 33, 53–55).同様に,軟体動物,環形動物,脊索動物といった主要な左右相称動物の系統はすべて,NaV1に加えてNaV2(CaV4)を持つ(図3).NaV2(CaV4)チャネルは,塩基性アミノ酸が等間隔に並ぶS4セグメントを持ち,ポアシグニチャーは一般にD/E/E/Aである.上述したように,Na+よりもCa2+やBa2+といった2価イオンに高い透過性を示す36, 40).ショウジョウバエのDSC1の変異体の解析からは,嗅覚刺激に応じた行動発現への関与,ストレス存在下における神経回路のパフォーマンスの安定化への関与が示唆されている54, 55).
脊索動物の姉妹群を構成する棘皮動物(ウニやヒトデの仲間)と半索動物(ギボシムシの仲間)は,D/E/E/A型のポアシグニチャーを持つNaV2(CaV4)を保持しているが,NaV1を失っている56)(図3).NaV1チャネルを捨てれば,速い神経インパルスを高頻度に送る仕組みを失うことになると思われるが,実際,これらの生物は速い捕食者ではなく,防御的な特徴を発達させ,体もそれほど大きくはない.たとえば,棘皮動物の体は炭酸カルシウムの骨格で覆われ,時にトゲを発達させる.半索動物は普通,海底の泥や砂の中,あるいは棲管に潜んで生活する.これらの動物では体全体を覆う表皮散在神経網が発達し,CNSは相対的に発達度が低い57).しばしばこの表皮散在神経網が神経管に集中化したことで脊椎動物のCNSが成立したとの説が考察されるが57),棘皮・半索動物におけるNaV1の喪失は,この方向性の説を支持しない.むしろNaV1の喪失の結果として,左右相称動物の起源以前の“遅い”動物の状態にこれらの動物は“先祖返り”したようにも解釈しうる.この点からも,NaV1の進化が左右相称動物の体のサイズや生活様式の限界を引き上げた貢献を推し量ることができる.
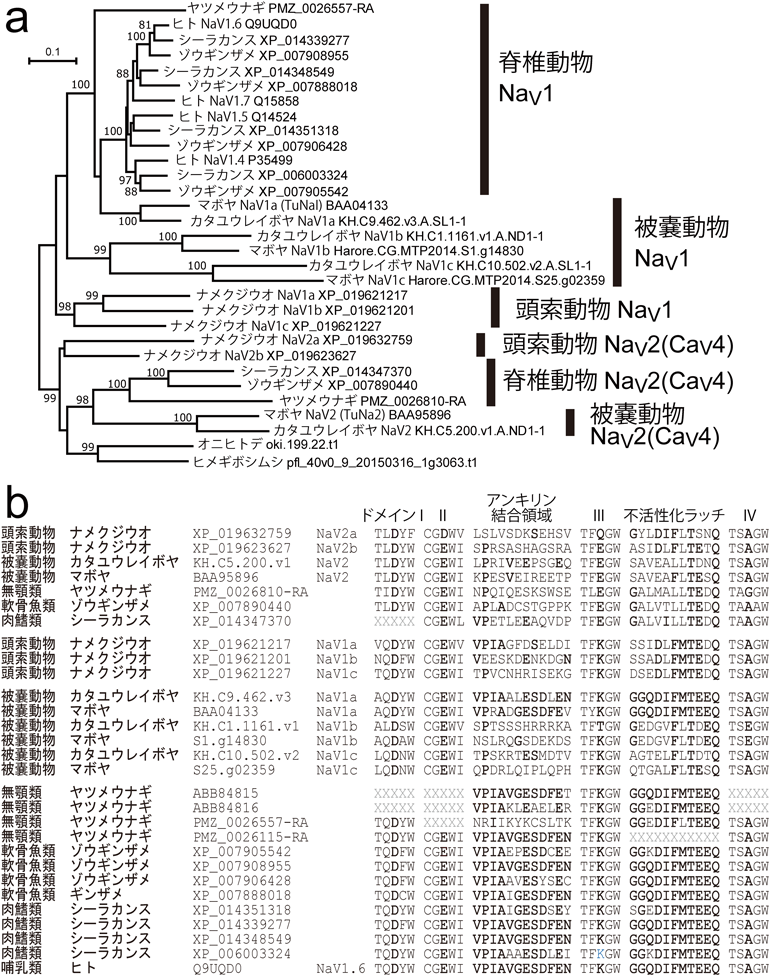
左右相称動物が放散したとき,脊索動物の系統では,脊椎動物とともに頭索動物(ナメクジウオ)と被嚢動物(尾索動物,ホヤの仲間)が派生した.近年の比較ゲノム解析により,ナメクジウオがこの三つのグループの中で最初に分岐し,被嚢動物と脊椎動物が姉妹群を構成することがわかっている58, 59).頭索動物と被嚢動物は,脊椎動物の体が確立する前の状況を知る“観察窓”となる動物である.
ナメクジウオは,海の砂地に濾過摂食者として生息している.発達した眼や脳を持たず,活発な捕食者でもないが,砂から泳ぎ出たり,またすばやく砂に戻って潜り込んだりする.ナメクジウオはNaVチャネルの遺伝子を五つ持つようだ.そのうちの三つはD/E/K/A型のポアシグニチャーを持つNaV1チャネルである.残り二つはNaV2(CaV4)タイプのパラログで,そのうちの一つ(NaV2a,図4)は,D/E/E/AではなくD/D/Q/Aというシグニチャーを持つ.このNaV2aのイオン透過性は未知だが,この因子がこの動物の興奮性細胞に新たな調節オプションを付与している可能性が考えられる.
似たような分子進化の軌跡が被嚢動物でも見いだされる.数種の被嚢動物のゲノムが解読され,ホヤには四つのNaV1/NaV2(CaV4)の遺伝子があることが判明している6, 35, 60, 61).これらのうち,NaV1a(これまでTuNaIと呼んでいたもの)は典型的なD/E/K/A型のポアシグニチャーを持ち,ほぼすべてのニューロンタイプに発現している6, 62).これはまた,ドメインIII~IV間のループに脊椎動物のものに近いI-F-Mを含む不活性化ラッチ,そしてドメインII~III間のループにアンキリン結合モチーフ(以下を参照)と非常に似た配列を備える(図4).ホヤはまた,D/E/E/A型の典型的なNaV2(CaV4)チャネルも保持している35)(図4;これまでTuNa2と呼んでいたもの).このNaV2(CaV4)も,ホヤの一部の(すべてではない)ニューロンが発現しており,NaV1とNaV2(CaV4)が入れ子状に発現するパターンは,ショウジョウバエのparaとDSC1でみられている関係に類似する35, 53).
ホヤにおける残り二つのチャネル(NaV1bとNaV1cと呼ぶ;以前はそれぞれNaV3と4と呼んでいたもの60))は,分子系統樹の上ではNaV1ファミリーに分類できるもので,被嚢動物のNaV1b, cのクレードは脊椎動物のNaV1のクレードからは独立している(図4a).このうちNaV1cのポアシグニチャーはD/E/K/Aだが,NaV1bのそれはD/E/(KまたはTまたはM)/Eになっている56)(図4b).またNaV1b, cはともに,I-F-Mモチーフを持った不活性化ラッチ構造と,明確なアンキリン結合モチーフ(後述)を欠く.
被嚢動物の中で,これらのパラログはどのように使い分けられているのだろうか.被嚢動物を代表するグループであるホヤは,成体の時期は固着性だが,幼生の時期はオタマジャクシの形をして海の中を泳ぐ.ホヤは,脊椎動物との類縁性の近さや,成熟した配偶子が豊富に得られること,「モザイク発生」をすることなどから,発生生物学研究のモデルとして長く研究に利用されてきた.特に,ホヤはニューロンや筋肉の分化に伴うイオンチャネルの発現に関する研究に,多大な貢献をしてきた63, 64).たとえば,細胞質分裂の阻害剤で処理して卵割を停止させた胚でも,神経細胞系譜の割球でNa+スパイクが発生することを示した歴史的な研究がある65, 66).この神経細胞系譜の割球におけるNa+電流の発現は,隣接する内中胚葉系譜の割球からの線維芽細胞成長因子(FGF)様の誘導シグナル(まさに神経誘導シグナル)に依存し,かつNaV1aチャネル(TuNaI)の遺伝子発現と固く相関する6, 66, 67).この系において,受精からニューロンの成熟段階に至るまでの間に,3タイプの電位依存性Na+電流が見いだされている68, 69).AタイプのNa+電流は単一の種類のNaVチャネルが担っており,受精卵から初期胚のすべての割球でみられる.このタイプのNa+電流は,卵が受精した際に発する活動電位(“受精電位”)を起こす電流と同じものと考えられる70).残りの2タイプのうち,Cタイプは分化した神経細胞割球で現れるNa+電流で,上記のように神経誘導に依存して発現するNaV1a(TuNaI)に担われていると考えられる6).もう一つのBタイプは,神経が分化していく過程でAタイプが消えてCタイプの電流が現れるまでの短い期間に現れる非典型的なNa+電流で68),短時間の連続した開孔(バースト活動)が持続するという特徴を持つ.現在のところ,これら3タイプの電流とNaVのアイソフォームの間の対応は完全には解明されていないが,これらの研究からは,受精や分化の過程にあるニューロンと分化したニューロンとでは,それぞれ異なる膜興奮性の制御がなされており,それぞれに適したNaVアイソフォームの利用が行われている可能性が示唆される.
被嚢動物が持つNaV1アイソフォームの中で,NaV1aは脊椎動物のNaV1パラログとの間で,「I-F-Mを含む不活性化ラッチ」と「アンキリン結合モチーフ」を共有している71)(図1, 3b, 4b).ここで成立したI-F-Mを含む不活性化ラッチのモチーフ配列(GGQDIFMTEEQ)は他の無脊椎動物には明確にみられず,また脊椎動物で同定されたすべてのNaV1チャネルでほぼ完全に保存されていることから,これが被嚢動物と脊椎動物の祖先で“新モデルの”不活性化ラッチとしてアップグレードされ,その後も利用されてきたと想像される.対してアンキリン結合モチーフは,ドメインII~III間のループに存在する約12個のアミノ酸(典型的にはVPIAVGESDFEN)からなる領域を指し,脊椎動物のNaV1チャネルの特にCNSで機能するアイソフォームによく保存されている(図4b, 5b参照).これに似た配列は別に,脊椎動物のKCNQ2/3(KV7.2/7.3とも呼ばれる)というK+チャネルにも見いだされ,これらはアンキリンGタンパク質との結合に必須で,さらには興味深いことにNaVチャネルとKCNQ2/3チャネルを,ミエリン化された軸索の起始部(axon initial segment:AIS)やランビエの絞輪に局在化させる71, 72).ホヤのNaV1aは,この脊椎動物のアンキリン結合モチーフに約70%一致した配列を持ち,ナメクジウオのNaV1aでも40%程度が一致している71)(図4b).軸索のミエリン鞘と,その結果生じる跳躍伝導は,顎のある脊椎動物(顎口類)の特徴である73).つまり頭索動物と被嚢動物にミエリンはないのに,これらの祖先のNaV1で,アンキリン結合モチーフに近いものが生まれたと考えられる.脊椎動物の系統で最初に分岐した無顎類のヤツメウナギもミエリンを持たないが,この動物のNaV1はアンキリン結合モチーフを明確に持ち,またこの配列はNaV1チャネルがAISに集積されるのに重要であると考えられている71).被嚢動物と脊椎動物の祖先ですでに,NaV1の軸索での局在がアンキリンとの相互作用によって可能になり,活動電位の発火効率の向上が果たされたのかもしれない.
頭索動物と被嚢動物をみてわかるのは,これらの中では,遺伝子重複によって生み出されたアイソフォームが互いに異なるように進化し,被嚢動物のNaV1bやナメクジウオのNaV2bのように,ポアシグニチャーにも及ぶ変化を受け入れてきたことである.遺伝子重複は,これまでも指摘されてきたように,生じたアイソフォームに,他とは異なる特別な調節オプションを与えうる74).現在のナメクジウオやホヤでみられるもの以上に多くのパラログが,脊椎動物の成立以前に生まれ,淘汰されたのだろう.そのようなものの中で,アップグレードされた不活性化ラッチと新たに導入されたアンキリン結合配列を備えた因子(ホヤのNaV1aのような)が,我々のすべてのNaV1チャネルを生み出す祖となった(図4a).
脊椎動物は単系統群であり,無顎類(ヌタウナギとヤツメウナギ),軟骨魚類(サメ,エイ),条鰭類(ポリプテルス,チョウザメ,ガー,アミア,真骨魚),肉鰭類(シーラカンスとハイギョ),四肢類(両生類,爬虫類,鳥類,哺乳類)を含む.この放散の過程の間に,脊椎動物は海水,淡水,湿地,乾地といったさまざまな環境に進出し,適応してきた.特に捕食者としての能力がよく発達しており,この能力は,神経軸索に沿ったミエリン鞘の形成と跳躍伝導,神経堤とプラコードに由来する“new head”と称される新しい感覚システムの導入により高められたと考えられている73, 75).活発な捕食者の存在は,被食者側の能力の強化をも刺激する.複雑な感覚入力を高速で情報処理し,効率的な運動調節につなげる能力は,水中と陸上の脊椎動物の放散を強くサポートしただろう.脊椎動物の祖先に備わっていたNaV1の機能は,これらのステップに本質的な役割を果たしたと考えられる.
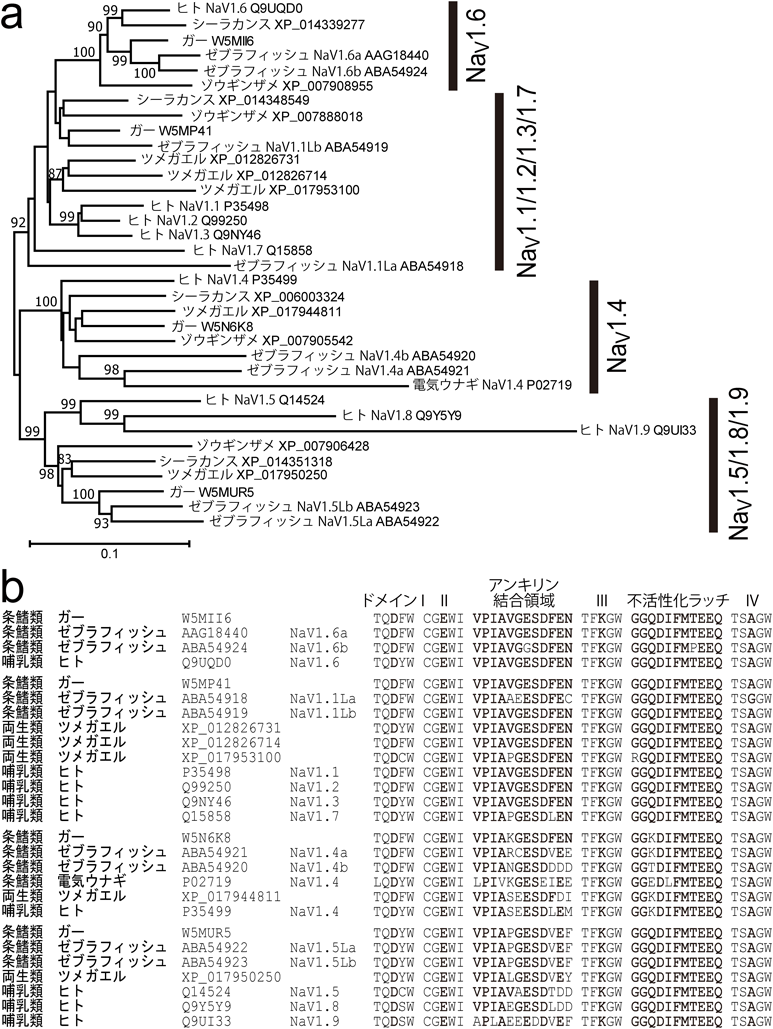
無顎類のヤツメウナギでは,複数種のNaV1チャネルの存在が予想される.そのうち二つは以前に報告されたものだが37, 71),少なくともあと2種類は存在するようだ(図4b).ギンザメなどの軟骨魚類も,4~5種類以上のNaV1アイソフォームを持つ(図4;いくつかの配列は短すぎ,ここでは省いた).我々の分子系統解析では,軟骨魚のNaV1アイソフォームは,脊椎動物の原始的な状態,すなわち羊膜類と真骨魚で起こった激しい重複(後述)以前の(NaV1.1/1.2/1.3/1.7),(NaV1.4),(NaV1.5/1.8/1.9),(NaV1.6)の各グループの因子を一つずつもつ状態を反映していることがみてとれた56)(図4,図5).長い配列情報がデータベースで得られた三つのヤツメウナギのNaV1は,おそらくすべてD/E/K/A型のポアシグニチャーを持ち,不活性化ラッチもホヤとの祖先から引き継ぐモチーフ配列を高い割合で保存していた.ただ,アンキリン結合モチーフの保存性はさまざま(50~100%)だった(図4b).
上述したように,真のミエリン鞘はヤツメウナギの軸索にはみられないが,軸索の起始部であるAISは他の軸索領域より細くなっており,そこにNaV1が特に集積されている71).軸索の細さは,局所の電気容量を下げ,電気抵抗を上げるので,鋭い活動電位の生成に資する構造となる.NaV1を細いAISに高濃度で局在させることは,効果的に活動電位を誘導する上で理に適う71, 76).このAISのマシナリは,顎口類におけるランビエの絞輪および跳躍伝導の進化への前適応になったと考えられる.
興味深いことに,ヤツメウナギ,軟骨魚類,シーラカンスはNaV2(CaV4)チャネルの遺伝子を持っている(図4).ポアシグニチャーはヤツメウナギではD/E/E/G,軟骨魚類とシーラカンスでは典型的なD/E/E/A型だった(図4b).NaV2(CaV4)は無脊椎動物にのみ存在すると考えられてきたが,今やこのチャネルは脊椎動物の系統にも残っていると認識された12).他方で,我々はNaV2(CaV4)を条鰭類や両生類のゲノムには見つけられなかったので,このチャネルは条鰭類と四肢類の系統で独立に失われたと考えられる.この喪失イベントは,これらの動物が海を離れて内陸の環境に進出するのと時期を同じくするようにみえるので,体を取り巻くイオン環境の変化が,NaV2(CaV4)を維持する選択圧を下げた可能性が想定される.しかしこのタイプのチャネルの動作原理がいまだはっきりしない以上,この問題は今後の課題となる.
7. 真骨魚と羊膜類におけるNaV1の独立な遺伝子重複
条鰭類,肉鰭類,四肢類の共通祖先は,HoxA~Dクラスターにそれぞれリンクする少なくとも4種類のNaV1を持っていたと考えられる38, 56).羊膜類のNaV1.1~1.3, 1.7をコードする遺伝子は,哺乳類ではさらにNaXの遺伝子も,HoxDクラスターにリンクした祖先遺伝子から連続的な遺伝子重複によって,おそらく[(1.3(1.2, 1.1))(1.7, X)]の順に,羊膜類になってから生じた56, 77).他方で,HoxBとHoxCにそれぞれリンクしたNaV1.4とNaV1.6をコードする遺伝子は,縦列重複を行っていない(図5).このうちNaV1.4は無羊膜類でも羊膜類でも骨格筋で,NaV1.6は神経系で働いてきたと考えられる.HoxAとリンクしたNaV1.5, 1.8, 1.9は,おそらく[1.5(1.8, 1.9)]の順に遺伝子重複を繰り返すことでもたらされた56, 77)(図5).このグループのNaV1は心臓における機能が古いようで,実際,哺乳類ばかりでなく,サメやハイギョにおいて,NaV1.5(より正確には,NaV1.5と1.8/1.9の祖先遺伝子のオーソログ)は心臓に発現しているようだ77).アノールトカゲやニワトリのゲノムにはNaV1.8や1.9の遺伝子があるが,ツメガエルのゲノムにはないので,NaV1.8と1.9をもたらした縦列重複は,NaV1.1/1.2/1.3/1.7の多様化と同様,羊膜類の進化に際して起こったと考えられる77)(図5).
哺乳類において,NaV1.1~1.3と1.6は主にCNSのニューロンに発現し,速い活動電位の生成に貢献している.PNSでは,NaV1.1や1.6もある程度発現しているが,その他に多くの感覚ニューロンでNaV1.7, 1.8, 1.9が,また幼若期にはNaV1.5も発現している16, 78)(表1).羊膜類の祖先はNaV1.6を主にCNSで,NaV1.7(より正確には1.7と1.1/1.2/1.3の祖先遺伝子)を主にPNSで,ニューロンの興奮性をコントロールするのに利用してきたのかもしれない.後に,NaV1.1~1.3がNaV1.7様の祖先タンパク質からもたらされ,羊膜類でさらに高度化したCNSの動作にリクルートされていったと考えられる.CNSで働くNaV1.1~1.3, 1.6は,発現する組合わせが細胞種によって異なるばかりでなく,ニューロンでの細胞内局在が異なることも知られている77, 79, 80).すなわち羊膜類におけるCNSサブタイプの多様化は,ニューロンが持つ多様な形態に適合した膜興奮性のfine-tuningを可能にしたかもしれない77).
NaV1.5は心臓で働くのに対し,羊膜類でこの因子から派生したNaV1.8と1.9は,背根神経節(DRG)の痛覚(侵害受容)ニューロンで主に機能する78)(表1).したがってNaV1.8と1.9を生み出した遺伝子重複は,痛覚の起源を考える上でも興味深い.NaV1.8と1.9は他のNaV1とは異なり,比較的ゆっくりした活性化と不活性化を示すチャネルである78)(図5b).このことは速い活性化と不活性化が追求されてきたチャネルのパラログの中に,新たに“遅さ”を特徴としたチャネルが生まれたことを示す.このNaV1.8/1.9の遅い動作は,侵害刺激の入力量に応じて,長く加算的な神経シグナルを生み出すことに関係しているようである.実際,これらは(特にNaV1.9は),小さなミエリン化されてないDRGニューロンに発現している78).すなわちニューロンの形態自体も,“速さ”を追求してない点で符合する.また哺乳類のNaV1.9のアンキリン結合部位は,配列の保存性がかなり低い(図5b).これらのことは,痛覚受容のいくつかの特性が羊膜類における発明であったことを示唆する.少なくとも,感覚ニューロンにおいて遅いNaV1.8と1.9を,速いNaV1.6や1.7と使い分けることで,異なる感覚情報を異なる様式で伝達できるようになっていることがわかる78).
羊膜類におけるNaV1遺伝子ファミリーの拡大はこのように,縦列重複によるものだった.羊膜類の進化に際して,類似の縦列重複がNaV1遺伝子の周囲の遺伝子で起こった事実はなく,またCaV1~3の遺伝子でも起きていない.したがって,このような縦列重複による進化は,羊膜類のNaV1ファミリーにかなり選択的に起こったと考えられる77).この羊膜類における多様化のパターンは,真骨魚類の系統で起きたものと対照的である.真骨魚類でもNaV1チャネル遺伝子の増加がみられるが,これは真骨魚の祖先で一度,ゲノム全体の重複(3Rと呼ばれる)が起こったことによる56, 77, 81, 82).つまり,真骨魚で起こった遺伝子重複は,NaV1に選択的なものではなく,グローバルなものだった.
ゼブラフィッシュにおける解析から,CNS, PNS,心臓,骨格筋の中で,発現しているNaV1アイソフォームに大きな多様性があることが示されている81, 82).たとえば,ゼブラフィッシュの心臓は,3Rで生じたNaV1.5パラログの両方を発現するが,DRGは片方だけを発現する81, 82).ゼブラフィッシュのDRGニューロンの電気生理学解析によると,2種類のNa+電流が確認され,第一のタイプは大きなニューロンから記録される速い不活性化を示すもの,第二のタイプは小さいニューロンから記録されるゆっくりと不活性化するものだった82).このうち前者はNaV1.6と(あるいは)NaV1.7によるもので,後者はNaV1.5のパラログの一つによるものと考えられているが82),このNaV1.5パラログは3Rで生じたペアのもう一方(DRGに発現しない方)に比べて不活性化が遅い83).真骨魚類と羊膜類のNaV1.5パラログ群の由来の違いを考えれば,DRG中の大きな,あるいは小さな感覚ニューロンに,それぞれ速いNaV1と遅いNaV1を発現させ,異なる感覚情報の伝達を担わせる様式は,真骨魚と羊膜類で独立に確立されたと推定される.また,これらの“遅い”NaV1サブタイプの分子進化が,ともに不活性化ラッチの配列変化を伴わずに起きている点も興味深い(図5).
羊膜類は,陸上環境への完全な適応を果たした動物群で,ついには巨大な恐竜,空飛ぶ鳥,そして哺乳類を生じた系統である.羊膜類の体の最大の特徴は,発達した前脳である.上でみてきた羊膜類におけるNaV1アイソフォームの拡張,すなわちNaV1.1~1.3と1.8/1.9の進化は,さらなる体サイズの増加,CNSでの情報処理の高速化,PNSに生まれた新たなイノベーションと結びついている77).これこそが,ついにはヒトの進化を導くに至った前適応だったと考えられる.
NaVチャネルの進化の歴史をみると,各ステップでさまざまな機能単位が繰り返し重複し,多様化していったさまをみてとれる.24回膜貫通型チャネルの成立には,CatSper様の6回膜貫通型のユニットが,2回縦列重複することが必要だった.その後,24回膜貫通型のポリペプチドの中に,回転非対称な変異を蓄積できるようになった.そしてこのチャネルは一つのユニットとして,さらなる重複と多様化の“タネ”となった.このようなトレンドの下で,NaV1ならD/E/K/A, NaV2(CaV4)ならD/E/E/Aという回転非対称なポアシグニチャーが成立し,NaV1にはさらに不活性化ラッチやアンキリン結合モチーフといった回転非対称に配置された機能領域が導入されていった.重複によって生み出された遺伝子のいくつかは進化のデッドエンドとなり,他のものは新たな重複と多様化を導く“タネ”となった.NaVチャネルのレパートリーは,ある動物系統では縮小され,他の系統では拡張された.NaV1チャネルが棘皮・半索動物で失われたのは前者の例であり,羊膜類で特に多様化したのは後者の例であろう.そしてそれぞれの例が,各動物群の適応現象によく対応している.NaVの存在/非存在やアイソフォームの多様化は,膜興奮性の制御に根本的なモーダルシフトを導ける.この根幹機構の変化は,動物の体の生理的な,ひいては形態的な可能性を拡張したり,あるいは制限したりしてきただろう.すなわち,体サイズの限界を押し広げたり,被食者を見定めて捕獲したり,捕食者を見定めて逃避したり,痛みを受けた記憶を思い起こして標的を変えたり,気流を感じつつ飛翔したり,魅力的な配偶相手を選んだり…….
未解明の問題も数多く残されている.たとえば陸上脊椎動物に至る道筋の中で,Na+選択的なNaV1が生まれ,Ca2+も通すNaV2(CaV4)が失われた.興味深いことに,NaV1/NaV2(CaV4)チャネルに限らず,脊椎動物の進化の流れにおいては,さまざまなイオンチャネルグループで平行的にCa2+透過性が失われてきた84–86).イオンチャネルの分子進化に関するさらなる研究が,さまざまな動物を取り巻く環境への未知の適応現象のディテールを,今後明らかにするだろう.
謝辞Acknowledgments
マボヤ(Halocynthia roretzi)のゲノム情報を未公開の段階から利用させていただいたPatrick Lemaire博士(IBDM, フランス),澤田均博士(名古屋大学),西田宏記博士(大阪大学)に感謝いたします.カタユウレイボヤを用いた研究はナショナルバイオリソースプロジェクトにサポートしていただいています.本稿の精読と細部のチェックを西野純子氏にしていただきました.
引用文献References
1) Hodgkin, A.L. & Huxley, A.F. (1952) Currents carried by sodium and potassium ions through the membrane of the giant axon of Loligo. J. Physiol., 116, 449–472.
2) Hartshorne, R.P. & Catterall, W.A. (1981) Purification of the saxitoxin receptor of the sodium channel from rat brain. Proc. Natl. Acad. Sci. USA, 78, 4620–4624.
3) Hartshorne, R.P., Keller, B.U., Talvenheimo, J.A., Catterall, W.A., & Montal, M. (1985) Functional reconstitution of the purified brain sodium channel in planar lipid bilayers. Proc. Natl. Acad. Sci. USA, 82, 240–244.
4) Noda, M., Shimizu, S., Tanabe, T., Takai, T., Kayano, T., Ikeda, T., Takahashi, H., Nakayama, H., Kanaoka, Y., Minamino, N., et al. (1984) Primary structure of Electrophorus electricus sodium channel deduced from cDNA sequence. Nature, 312, 121–127.
5) Loughney, K., Kreber, R., & Ganetzky, B. (1989) Molecular analysis of the para locus, a sodium channel gene in Drosophila. Cell, 58, 1143–1154.
6) Okamura, Y., Ono, F., Okagaki, R., Chong, J.A., & Mandel, G. (1994) Neural expression of a sodium channel gene requires cell-specific interactions. Neuron, 13, 937–948.
7) Payandeh, J. & Minor, D.L. Jr. (2015) Bacterial voltage-gated sodium channels (BacNaVs) from the soil, sea, and salt lakes enlighten molecular mechanisms of electrical signaling and pharmacology in the brain and heart. J. Mol. Biol., 427, 3–30.
8) Catterall, W.A. & Zheng, N. (2015) Deciphering voltage-gated Na+ and Ca2+ channels by studying prokaryotic ancestors. Trends Biochem. Sci., 40, 526–534.
9) Shen, H., Zhou, Q., Pan, X., Li, Z., Wu, J., & Yan, N. (2017) Structure of a eukaryotic voltage-gated sodium channel at near-atomic resolution. Science, 355, eaaul4326.
10) Yan, Z., Zhou, Q., Wang, L., Wu, J., Zhao, Y., Huang, G., Peng, W., Shen, H., Lei, J., & Yan, N. (2017) Structure of the NaV1.4-β1 complex from electric eel. Cell, 170, 470–482.e11.
11) Pan, X., Li, Z., Zhou, Q., Shen, H., Wu, K., Huang, X., Chen, J., Zhang, J., Zhu, X., Lei, J., et al. (2018) Structure of the human voltage-gated sodium channel NaV1.4 in complex with β1. Science, 362, eaau2486.
12) Nishino, A. & Okamura, Y. (2018) Evolutionary history of voltage-gated sodium channels. In Voltage-gated Sodium Channels: Structure, Function and Channelopathies. (Chahine, M. ed.). Handb. Exp. Pharmacol., 246, 3–32.
13) Catterall, W.A. (2000) From ionic currents to molecular mochanisms: The structure and function of voltage-gated sodium channels. Neuron, 26, 13–25.
14) Matsumoto, M., Hiyama, T.Y., Kuboyama, K., Suzuki, R., Fujikawa, A., & Noda, M. (2015) Channel properties of NaX expressed in neurons. PLoS One, 10, e0126109.
15) Hiyama, T.Y. & Noda, M. (2016) Sodium sensing in the subfornical organ and body-fluid homeostasis. Neurosci. Res., 113, 1–11.
16) Goldin, A.L. (2001) Resurgence of sodium channel research. Annu. Rev. Physiol., 63, 871–894.
17) Catterall, W.A., Goldin, A.L., & Waxman, S.G. (2005) International union of pharmacology. XLVII. Nomenclature and structure-function relationships of voltage-gated sodium channels. Pharmacol. Rev., 57, 397–409.
18) Stühmer, W., Conti, F., Suzuki, H., Wang, X., Noda, M., Yahagi, N., Kubo, H., & Numa, S. (1989) Structure parts involved in activation and inactivation of the sodium channel. Nature, 339, 597–603.
19) West, J.W., Patton, D.E., Scheuer, T., Wang, Y., Goldin, A.L., & Catterall, W.A. (1992) A cluster of hydrophobic amino acid residues required for fast Na+-channel inactivation. Proc. Natl. Acad. Sci. USA, 89, 10910–10914.
20) Heinemann, S.H., Terlau, H., Stühmer, W., Imoto, K., & Numa, S. (1992) Calcium channel characteristics conferred on the sodium channel by single mutations. Nature, 356, 441–443.
21) Noda, M., Suzuki, H., Numa, S., & Stühmer, W. (1989) A single point mutation confers tetrodotoxin and saxitoxin insensitivity on the sodium channel II. FEBS Lett., 259, 213–216.
22) Ren, D., Navarro, B., Xu, H., Yue, L., Shi, Q., & Clapham, D.E. (2001) A prokaryotic voltage-gated sodium channel. Science, 294, 2372–2375.
23) Yue, L., Navarro, B., Ren, D., Ramos, A., & Clapham, D.E. (2002) The cation selectivity filter of the bacterial sodium channel, NaChBac. J. Gen. Physiol., 120, 845–853.
24) Jiang, Y., Lee, A., Chen, J., Ruta, V., Cadene, M., Chait, B.T., & MacKinnon, R. (2003) X-ray structure of a voltage-dependent K+ channel. Nature, 423, 33–41.
25) Bichet, D., Haass, F.A., & Jan, L.Y. (2003) Merging functional studies with structures of inward-rectifier K+ channels. Nat. Rev. Neurosci., 4, 957–967.
26) Honoré, E. (2007) The neuronal background K2P channels: Focus on TREK1. Nat. Rev. Neurosci., 8, 251–261.
27) Murata, Y., Iwasaki, H., Sasaki, M., Inaba, K., & Okamura, Y. (2005) Phosphoinositide phosphatase activity coupled to an intrinsic voltage sensor. Nature, 435, 1239–1243.
28) Ramsey, I.S., Moran, M.M., Chong, J.A., & Clapham, D.E. (2006) A voltage-gated proton-selective channel lacking the pore domain. Nature, 440, 1213–1216.
29) Sasaki, M., Takagi, M., & Okamura, Y. (2006) A voltage sensor-domain protein is a voltage-gated proton channel. Science, 312, 589–592.
30) Liebeskind, B.J., Hillis, D.M., & Zakon, H.H. (2013) Independent acquisition of sodium selectivity in bacterial and animal sodium channels. Curr. Biol., 23, R948–R949.
31) Calcraft, P.J., Ruas, M., Pan, Z., Cheng, X., Arredouani, A., Hao, X., Tang, J., Rietdorf, K., Teboul, L., Chuang, K.-T., et al. (2009) NAADP mobilizes calcium from acidic organelles through two-pore channels. Nature, 459, 596–600.
32) Strong, M., Chandy, K.G., & Gutman, G.A. (1993) Molecular evolution of voltage-sensitive ion channel genes: On the origins of electrical excitability. Mol. Biol. Evol., 10, 221–242.
33) Salkoff, L., Butler, A., Wei, A., Scavarda, N., Giffen, K., Ifune, C., Goodman, R., & Mandel, G. (1987) Genomic organization and deduced amino acid sequence of a putative sodium channel gene in Drosophila. Science, 237, 744–749.
34) Sato, C. & Matsumoto, G. (1992) Primary structure of squid sodium channel deduced from the complementary DNA sequence. Biochem. Biophys. Res. Commun., 186, 61–68.
35) Nagahora, H., Okada, T., Yahagi, N., Chong, J.A., Mandel, G., & Okamura, Y. (2000) Diversity of voltage-gated sodium channels in the ascidian larval nervous system. Biochem. Biophys. Res. Commun., 275, 558–564.
36) Zhou, W., Chung, I., Liu, Z., Goldin, A., & Dong, K. (2004) A voltage-gated calcium-selective channel encoded by a sodium channel-like gene. Neuron, 42, 101–112.
37) Zakon, H.H. (2012) Adaptive evolution of voltage-gated sodium channels: The first 800 million years. Proc. Natl. Acad. Sci. USA, 109(Suppl 1), 10619–10625.
38) Gur Barzilai, M., Reitzel, A.M., Kraus, J.E., Gordon, D., Technau, U., Gurevitz, M., & Moran, Y. (2012) Convergent evolution of sodium ion selectivity in metazoan neuronal signaling. Cell Rep., 2, 242–248.
39) Moran, Y., Liebeskind, B.J., & Zakon, H.H. (2015) Evolution of voltage-gated ion channels at the emergence of Metazoa. J. Exp. Biol., 218, 515–525.
40) Gosselin-Badaroudine, P., Moreau, A., Simard, L., Cens, T., Rousset, M., Collet, C., Charnet, P., & Chahine, M. (2016) Biophysical characterization of the honeybee DSC1 orthologue reveals a novel voltage-dependent Ca2+ channel subfamily: CaV4. J. Gen. Physiol., 148, 133–145.
41) Liebeskind, B.J., Hillis, D.M., & Zakon, H.H. (2012) Phylogeny unites animal sodium leak channels with fungal calcium channels in an ancient, voltage-insensitive clade. Mol. Biol. Evol., 29, 3613–3616.
42) Roger, A.J. & Simpson, A.G.B. (2008) Evoluton: Revisiting the root of the eukaryote tree. Curr. Biol., 19, R165–R167.
43) Cavalier-Smith, T. (2010) Kingdoms Protozoa and Chromista and the eozoan root of the eukaryotic tree. Biol. Lett., 6, 342–345.
44) Brunet, T. & Arendt, D. (2015) From damage response to action potentials: Early evolution of neural and contractile modules in stem eukaryotes. Philos. Trans. R. Soc. Lond. B Biol. Sci., 371, 20150043.
45) Machemer, H. & Ogura, A. (1979) Ionic conductances of membranes in ciliated and deciliated Paramecium. J. Physiol., 296, 49–60.
46) Fujiu, K., Nakayama, Y., Yanagisawa, A., Sokabe, M., & Yoshimura, K. (2009) Chlamydomonas CAV2 encodes a voltage-dependent calcium channel required for the flagellar waveform conversion. Curr. Biol., 19, 133–139.
47) Liebeskind, B.J., Hillis, D.M., & Zakon, H.H. (2011) Evolution of sodium channels predates the origin of nervous systems in animals. Proc. Natl. Acad. Sci. USA, 108, 9154–9159.
48) Anderson, P.A.V., Holman, M.A., & Greenberg, R.M. (1993) Deduced amino acid sequence of a putative sodium channel from the scyphozoan jellyfish Cyanea capillata. Proc. Natl. Acad. Sci. USA, 90, 7419–7423.
49) Moran, Y. & Zakon, H.H. (2014) The evolution of the four subunits of voltage-gated calcium channels: Ancient roots, increasing complexity, and multiple losses. Genome Biol. Evol., 6, 2210–2217.
50) Conway-Morris, S. (1986) The community structure of the Middle Cambrian phyllopod bed (Burgess Shale). Palaeontology, 29, 423–467.
51) Gould, S.J.(1990) Wonderful life: The Burgess Shale and the nature of history. WW Norton & Co.
52) Parker, A.(2003) In the blink of an eye. Basic Books.
53) Hong, C.S. & Ganetzky, B. (1994) Spatial and temporal expression patterns of two sodium channel genes in Drosophila. J. Neurosci., 14, 5160–5169.
54) Kulkarni, N.H., Yamamoto, A.H., Robinson, K.O., Mackay, T.F.C., & Anholt, R.R. (2002) The DSC1 channel, encoded by the smi60E locus, contributes to odor-guided behavior in Drosophila melanogaster. Genetics, 161, 1507–1516.
55) Zhang, T., Wang, Z., Wang, L., Luo, N., Jiang, L., Liu, Z., Wu, C.-F., & Dong, K. (2013) Role of the DSC1 channel in regulating neuronal excitability in Drosophila melanogaster: Extending nervous system stability under stress. PLoS Genet., 9, e1003327.
56) Widmark, J., Sundström, G., Ocampo Daza, D., & Larhammar, D. (2011) Differential evolution of voltage-gated sodium channels in tetrapods and teleost fishes. Mol. Biol. Evol., 28, 859–871.
57) Holland, N.D. (2003) Early central nervous system evolution: An era of skin brains? Nat. Rev. Neurosci., 4, 617–627.
58) Delsuc, F., Brinkmann, H., Chourrout, D., & Philippe, H. (2006) Tunicates and not cephalochordates are the closest living relatives of vertebrates. Nature, 439, 965–968.
59) Putnam, N.H., Butts, T., Ferrier, D.E., Furlong, R.F., Hellsten, U., Kawashima, T., Robinson-Rechavi, M., Shoguchi, E., Terry, A., Yu, J.K., et al. (2008) The amphioxus genome and the evolution of the chordate karyotype. Nature, 453, 1064–1071.
60) Okamura, Y., Nishino, A., Murata, Y., Nakajo, K., Iwasaki, H., Ohtsuka, Y., Tanaka-Kunishima, M., Takahashi, N., Hara, Y., Yoshida, T., et al. (2005) Comprehensive analysis of the ascidian genome reveals novel insights into the molecular evolution of ion channel genes. Physiol. Genomics, 22, 269–282.
61) Brozovic, M., Martin, C., Dantec, C., Dauga, D., Mendez, M., Simion, P., Percher, M., Laporte, B., Scornavacca, C., Di Gregorio, A., et al. (2016) ANISEED 2015: A digital framework for the comparative developmental biology of ascidians. Nucleic Acids Res., 44(D1), D808–D818.
62) Okada, T., Hirano, H., Takahashi, K., & Okamura, Y. (1997) Distinct neuronal lineages of the ascidian embryo revealed by expression of a sodium channel gene. Dev. Biol., 190, 257–272.
63) Takahashi, K., Miyazaki, S., & Kidokoro, Y. (1971) Development of excitability in embryonic muscle cell membranes in certain tunicates. Science, 171, 415–417.
64) Simoncini, L., Block, M.L., & Moody, W.J. (1988) Lineage-specific development of calcium currents during embryogenesis. Science, 242, 1572–1575.
65) Takahashi, K. & Yoshii, M. (1981) Development of sodium, calcium and potassium channels in the cleavage-arrested embryo of an ascidian. J. Physiol., 315, 515–529.
66) Takahashi, K. & Okamura, Y. (1998) Ion channels and early development of neural cells. Physiol. Rev., 78, 307–337.
67) Okado, H. & Takahashi, K. (1988) A simple “neural induction” model with two interacting cleavage-arrested ascidian blastomeres. Proc. Natl. Acad. Sci. USA, 85, 6197–6201.
68) Okamura, Y. & Shidara, M. (1990) Changes in sodium channels during neural differentiation in the isolated blastomere of the ascidian embryo. J. Physiol., 431, 39–74.
69) Okamura, Y. & Shidara, M. (1990) Inactivation kinetics of the sodium channel in the egg and the isolated, neutrally differentiated blastomere of the ascidian. J. Physiol., 431, 75–102.
70) Fukushima, Y. (1981) Identification and kinetic properties of the current through a single Na+ channel. Proc. Natl. Acad. Sci. USA, 78, 1274–1277.
71) Hill, A.S., Nishino, A., Nakajo, K., Zhang, G., Fineman, J.R., Selzer, M.E., Okamura, Y., & Cooper, E.C. (2008) Ion channel clustering at the axon initial segment and node of Ranvier evolved sequentially in early chordates. PLoS Genet., 4, e1000317.
72) Pan, Z., Kao, T., Horvath, Z., Lemos, J., Sul, J.Y., Cranstoun, S.D., Bennett, V., Scherer, S.S., & Cooper, E.C. (2006) A common ankyrin-G-based mechanism retains KCNQ and NaV channels at electrically active domains of the axon. J. Neurosci., 26, 2599–2613.
73) Zalc, B. (2016) The acquisition of myelin: An evolutionary perspective. Brain Res., 1641(Pt A), 4–10.
74) Ohno, S.(1970) Evolution by gene duplication. Springer-Verlag.
75) Gans, C. & Northcutt, R.G. (1983) Neural crest and the origin of vertebrates: A new head. Science, 220, 268–273.
76) Kole, M.H. & Stuart, G.J. (2012) Signal processing in the axon initial segment. Neuron, 73, 235–247.
77) Zakon, H.H., Jost, M.C., & Lu, Y. (2011) Expansion of voltage-dependent Na+ channel gene family in early tetrapods coincided with the emergence of terrestriality and increased brain complexity. Mol. Biol. Evol., 28, 1415–1424.
78) Lai, J., Porreca, F., Hunter, J.C., & Gold, M.S. (2004) Voltage-gated sodium channels and hyperalgesia. Annu. Rev. Pharmacol. Toxicol., 44, 371–397.
79) Hu, W., Tian, C., Li, T., Yang, M., Hou, H., & Shu, Y. (2009) Distinct contributions of NaV1.6 and NaV1.2 in action potential initiation and backpropagation. Nat. Neurosci., 12, 996–1002.
80) Lorincz, A. & Nusser, Z. (2010) Molecular identity of dendritic voltage-gated sodium channels. Science, 328, 906–909.
81) Novak, A.E., Taylor, A.D., Pineda, R.H., Lasda, E.L., Wright, M.A., & Ribera, A.B. (2006) Embryonic and larval expression of zebrafish voltage-gated sodium channel alpha-subunit genes. Dev. Dyn., 235, 1962–1973.
82) Won, Y.-J., Ono, F., & Ikeda, S.R. (2012) Characterization of Na+ and Ca2+ channels in zebrafish dorsal root ganglion neurons. PLoS One, 7, e42602.
83) Chopra, S.S., Stroud, D.M., Watanabe, H., Bennett, J.S., Burns, C.G., Wells, K.S., Yang, T., Zhong, T.P., & Roden, D.M. (2010) Voltage-gated sodium channels are required for heart development in zebrafish. Circ. Res., 106, 1342–1350.
84) Schredelseker, J., Shrivastav, M., Dayal, A., & Grabner, M. (2010) Non-Ca2+-conducting Ca2+ channels in fish skeletal muscle excitation-contraction coupling. Proc. Natl. Acad. Sci. USA, 107, 5658–5663.
85) Nishino, A., Baba, S.A., & Okamura, Y. (2011) A mechanism for graded motor control encoded in the channel properties of the muscle ACh receptor. Proc. Natl. Acad. Sci. USA, 108, 2599–2604.
86) Hirai, S., Hotta, K., Kubo, Y., Nishino, A., Okabe, S., Okamura, Y., & Okado, H. (2017) AMPA glutamate receptors are required for sensory-organ formation and morphogenesis in the basal chordate. Proc. Natl. Acad. Sci. USA, 114, 3939–3944.
著者紹介Author Profile
 西野 敦雄(にしの あつお)
西野 敦雄(にしの あつお)弘前大学農学生命科学部准教授.博士(理学)(2002年3月,京都大学).
略歴1973年神奈川県生まれ.97年東京大学理学部卒業.2001年京都大学大学院理学研究科中退.同年東京大学新領域創成科学研究科助手.04年自然科学研究機構研究員.07年大阪大学理学研究科助教.12年より現職.
研究テーマと抱負脊索動物の系統で起こったさまざまな筋肉タイプの進化を,“イオンチャネルや収縮タンパク質のモデルチェンジ”という視点で読み解きたいと思っています.自分のNaV1をいかし,何事も素早くするのが目標.
ウェブサイトhttp://nature.cc.hirosaki-u.ac.jp/staff/atsuo-nishino/
趣味パズルを解く.読書.家族と会話.
 岡村 康司(おかむら やすし)
岡村 康司(おかむら やすし)大阪大学大学院医学系研究科教授.博士(医学)(1989年3月,東京大学).
略歴1960年東京都生まれ.85年東京大学医学部卒業.89年同大学院医学研究科修了.東京大学助手,工業技術院主任研究官,自然科学研究機構教授などを経て2008年より現職.
研究テーマと抱負膜電位信号タンパク質についてwhat?(特性)how?(分子動作原理)why?(生理学,進化上の意義)を理解することを目指しています.Qを解くための研究だけでなく新たなQを生み出す研究を心がけたいです.
ウェブサイトhttp://www.med.osaka-u.ac.jp/pub/phys2/okamura/index.html
趣味地方の美術館など.