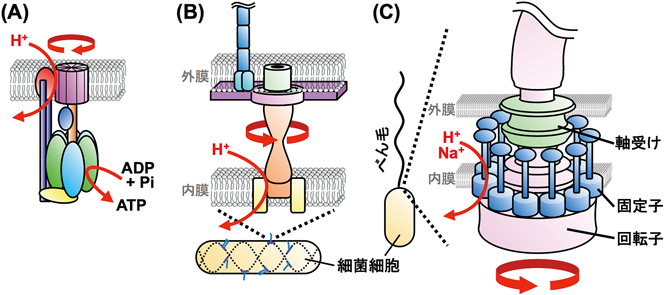
自然界には,イオン駆動力をエネルギー源として回転する分子モーターが3種類存在する.一つはF型ATPaseに代表されるATP合成/分解酵素であり,真核生物の細胞小器官(ミトコンドリア,葉緑体,液胞など)の膜上や原核生物の細胞膜上に存在し,イオンの流入と共役してATPを合成し,あるいは逆にATPを分解するエネルギーを使ってイオンの濃度勾配を作る(図1A)1).二つ目が,ある種の滑走細菌が持つ運動器官(滑走装置)で,近年,プロトン駆動力を用いて回転することが発見された(図1B)2).三つ目がべん毛モーターであり,真正細菌が持つ運動器官であるべん毛の基部において膜に埋め込まれる形で存在し,イオン流によりべん毛を回転させることで細菌の運動を可能にする(図1C)3).ATPaseの回転運動は酵素反応過程において副次的に生じるのに対して,べん毛モーターの回転運動はイオン駆動力を純粋に回転力に変換するために行われる現象であり,べん毛モーターは世界最小の回転原動機であるといえる.
べん毛は細菌細胞の表面から生えたらせん繊維状のタンパク質複合体で,細菌はこれをスクリューのように回転させることで,自身の生存に有利な環境を求めて遊泳する.べん毛モーターは,人類が発明した電気機械モーターと同様に,固定子・回転子・軸受けという三つの部品から構成される.しかし機械モーターと異なり,べん毛モーターは直径約50ナノメートルと非常に小さく,またすべてタンパク質を材料として作られる.回転子が10種類以上・200分子以上のタンパク質からなるのに対して,固定子は2種類のタンパク質からなり,回転子の周囲へと最大で1ダース前後の固定子が集合する4, 5).固定子はイオンチャネルとして働き,固定子を介したイオンの流入と共役して固定子の細胞質側領域が回転子と相互作用し,回転力が生み出される.このエネルギー源となる共役イオン[プロトン(H+),ナトリウムイオン(Na+)など]の種類は,細菌の種類によってさまざまであり,たとえば大腸菌,サルモネラなどではH+が,ビブリオ属細菌や枯草菌などではNa+も使用される3, 6).近年,ある種の細菌ではカリウムイオン(K+)やカルシウムイオン(Ca2+)で駆動されるべん毛モーターも見つかった7).
べん毛モーターはその小ささでありながら秒速200~1000回転以上という速さで回転し,またトップスピードから瞬時に回転方向を切り替えることができる.それでいて,ほぼ100%に近いエネルギー変換効率を持つというきわめて優れたモーターである.これまで,べん毛モーターの研究は遺伝学的・生化学的・物理学的・構造学的研究などさまざまな角度から行われ,回転特性や回転方向制御機構,形成機構などが明らかにされた.しかし,最も重要なモーターの回転力発生機構そのものに関しては明らかになっていることは多くない.どうやって固定子は回転子の周りに正しく集合し,どうやって固定子内をイオンが流入し,そしてどのような仕組みで固定子と回転子が相互作用するのか,未解明な部分が多い.本稿では,著者らが発見して研究を進めた海洋性細菌(Vibrio alginolyticus)や超好熱性細菌(Aquifex aeolicus)のNa+駆動型べん毛モーターの研究の進展を紹介しつつ,最新のべん毛モーター構造とそのエネルギー変換機構についての知見を概説したい.
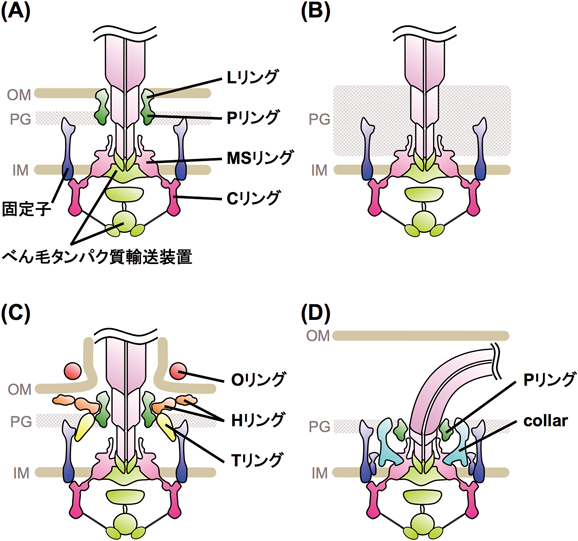
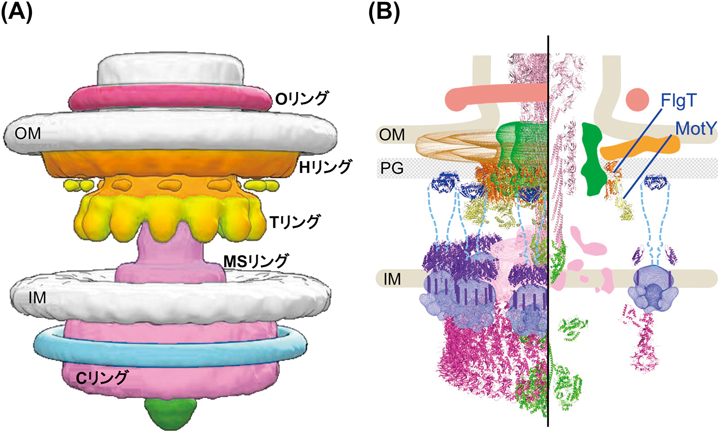
べん毛基部体は,回転子と軸受けを内包したモーターの大部分を占める部分であり,多くのタンパク質が中心軸の周りにリング状に集合して作られる(図2).べん毛基部体を形作るリング構造は,菌体外側から順にそれぞれLリング,Pリング,MSリング,Cリングと呼ばれる(図2A)8–11).グラム陽性菌ではLリングとPリングがなく(図2B),海洋性ビブリオ菌V. alginolyticusの極べん毛モーターはこれらに加え,HリングおよびTリングを持つ(図2C)12, 13).スピロヘータの一種Borrelia burgdorferiは“collar(襟)”と呼ばれる特徴的なリング構造を持つ(図2D)14).これらのリング構造の中で回転力発生のために最も重要なのが,細胞質に存在するCリングである.Cリングは三つのタンパク質FliG, FliM, FliNからなる15).一つのモーターに対して,FliGとFliMがそれぞれ約30分子,FliNが100分子以上集合する9, 16, 17).このCリングの周りへと十数個の固定子が集合する(図1C)4, 5).
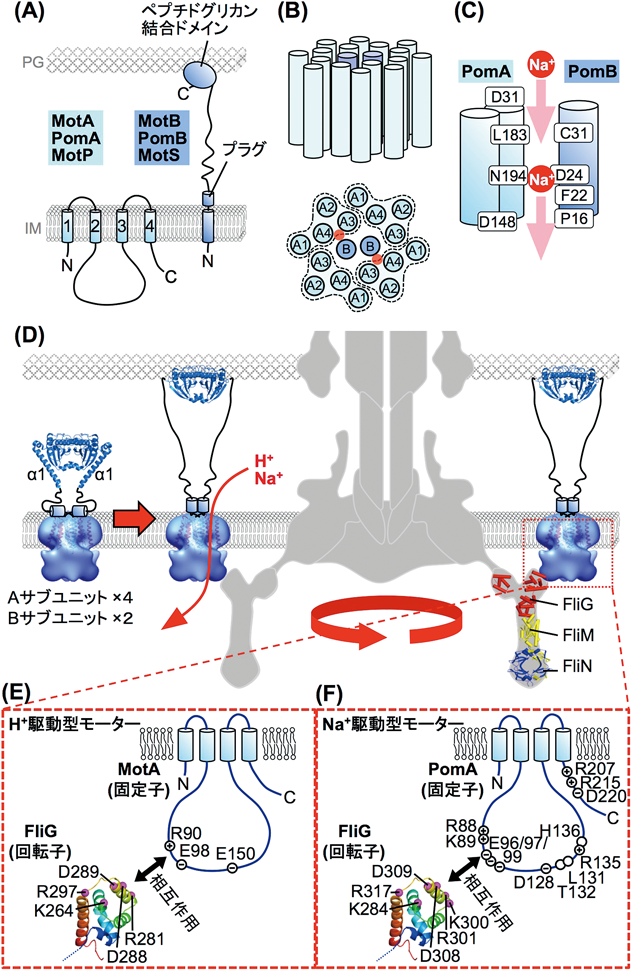
固定子は,モーターが回転力を発生する際,エネルギー変換ユニットとして働く膜タンパク質複合体であり,2種類のタンパク質MotAとMotB(あるいはPomAとPomB,あるいはMotPとMotS)からなる(図3A).PomAとPomBはH+共役型以外の固定子タンパク質として著者らによって同定されたタンパク質である.以下,MotA/PomA/MotPをAサブユニット,MotB/PomB/MotSをBサブユニットと呼称する.AサブユニットとBサブユニットは4:2のヘテロ六量体を形成し,一つの固定子を形成する18–20).Aサブユニットは4回膜貫通型のタンパク質で,2番目と3番目の膜貫通領域の間に細胞質側ループを持つ(図3A)21, 22).この細胞質側ループ上の保存された荷電残基が回転力発生のためのFliGとの相互作用において重要であると考えられている23, 24).Bサブユニットは1回膜貫通型のタンパク質で,カルボキシ末端(C末端)側に大きなペリプラズム側領域を持つ21)(図3A).このペリプラズム側領域には,ペプチドグリカン結合モチーフが存在し,固定子をペプチドグリカン層に固定する働きを担う.
固定子はイオンチャネルとして働き,Bサブユニットの膜貫通領域とAサブユニットの3番目と4番目の膜貫通領域でイオンチャネル孔を形成する(図3B, C)25–28).Bサブユニットの膜貫通領域には,共役イオンが結合する保存されたアスパラギン酸(Asp, D)残基が存在する23, 29, 30).このAsp残基をアスパラギン(Asn, N)残基に置き換えた変異体ではイオンの流入が妨げられ,べん毛モーター機能が完全に阻害される31).V. alginolyticusのNa+駆動型モーターにおいて,PomB上の保存されたAsp残基(D24)から2アミノ酸離れたフェニルアラニン残基(F22)はイオン結合ポケットからのNa+の効率的な放出に重要であると考えられている32).Bサブユニットはペリプラズム側領域にプラグ領域と呼ばれる部位を持ち(図3D),固定子が回転子の周りに集合していないときにイオン流入を防ぐ働きを担っている33–36).固定子PomA/PomBが回転子の周りへと集合するためには環境中の共役イオン(Na+)が必要であり37),またFliGのC末端ドメイン内のいくつかの変異により固定子の回転子周囲への集合能が低下することから,PomAの細胞質側領域とFliGのC末端ドメインとの間の相互作用が,回転力発生のみならず固定子の集合にも重要であり,著者らはこの相互作用がPomBのプラグの開閉やペリプラズム側領域の構造変化を誘発するというモデルを提唱した38, 39).これらの構造変化により共役イオンが固定子内へと流入してイオン結合ポケットへと結合できるようになり,その結果として固定子が回転子の周りへと集合固定できるようになると推測された.実際,Aサブユニットの細胞質側ループ上の保存された荷電残基(アルギニン残基(Arg, R)およびグルタミン酸残基(Glu, E)は回転力産生においてのみならず,固定子の回転子周囲への集合にも重要である24, 40).また,プラグ領域を欠失した固定子を細菌内で大量発現すると細菌の生育が阻害されることが知られている.この生育阻害は,プラグを失った固定子を介して細胞内へと過剰なイオンが流入することによって生じる35, 36).
このプラグ欠失固定子を用いることで,非常に簡便に固定子のイオン透過活性を測定することができる.著者らはプラグ欠失固定子(PomA/PomBΔL)を大量発現した際の細胞内Na+濃度を2種類の方法(原子吸光およびナトリウム蛍光指示薬を用いた顕微鏡イメージング)によって測定し,細胞内Na+濃度が3倍程度上昇することを発見した.この上昇はPomB上の保存されたD24の変異により完全に抑制され,また細胞内Na+濃度上昇量は生育阻害と高い相関を持つことが示された31).つまり細菌の生育を観察するだけで固定子のイオン透過活性を定量することができ,これを用いることでイオン透過活性を減弱させる変異や,影響しない変異,または上昇させる変異を容易に見分けられる.これを応用した一例として,回転力発生に直接関係すると考えられる固定子細胞質側領域の変異によってチャネル活性が影響されることから,イオン透過と固定子構造変化の強い相互関係が見いだされた.最近,著者らは固定子複合体を精製し,固定子のNa+イオンの結合を分光学的手法によって検出することに成功した41).さらに,PomAの膜貫通領域に存在するThr残基の重要性を明らかにしている.べん毛固定子複合体のイオン流入機構解明も間近であると思う.
前述のとおり,べん毛モーターの回転力発生のためには,固定子–回転子間の相互作用が重要である.固定子内をイオンが流入することによって,固定子の細胞質側領域の構造が変化し,それにより固定子–回転子間相互作用が変化して回転力が生み出される,というモデルが現在広く信じられている(パワーストロークモデル)42).大腸菌のH+駆動型モーターの研究から,MotAの細胞質側ループおよびFliGのC末端側のドメイン(Cドメイン)上の保存された荷電残基間の静電的相互作用(MotA-R90 vs. FliG-D289間の相互作用,MotA-E98 vs. FliG-R281間の相互作用)が回転力発生に重要であると考えられている(図3E)43).一方,V. alginolyticusのNa+駆動型モーターでは,それらの保存された荷電残基[PomAのR88, K89, E96, E97, E99およびFliGのリシン(K)284, R301, D308, D309, R317]を無電荷残基や逆電荷残基に置き換えてもモーターの機能が失われない44, 45).しかしNa+駆動型キメラ固定子PomA/PotB(V. alginolyticusのPomBのペリプラズム側領域だけを大腸菌のMotBに置き換えたキメラタンパク質)による大腸菌べん毛モーター回転能を解析したところ,モーター機能への保存荷電残基の重要性が確認されたことから46),V. alginolyticusのNa+駆動型モーターにおいては,PomA-FliG間の保存荷電残基間の相互作用は回転力産生に関係するものの,他の重要な残基が存在する可能性が考えられた.Na+駆動型モーターはH+駆動型モーターより高速回転するため,速い回転を発揮するために他の多数の荷電残基が必要と思われた(図3F).
V. alginolyticusのNa+駆動型モーターにおいて,PomA細胞質側ループ上の131, 132, 135, 136番目のロイシン,トレオニン,アルギニン,ヒスチジンの変異により,モーターは温度感受性の運動能や運動能の低下を示す47).同じくPomA細胞質側にあたるC末端領域上の変異(R207E, R215E, D220K)によっても,モーター機能が阻害される48).FliGのCドメイン上の保存荷電残基の変異を含めたいくつかの変異によっても,固定子の集合能およびべん毛モーター機能が低下する39).したがって,これらの変異部位がPomA-FliG間相互作用に関与しており,Na+駆動型モーターの回転力発生におけるPomAの細胞質側領域とFliGのCドメインとの間の相互作用の重要性が考えられる.V. alginolyticusのPomAやFliGは大腸菌などのMotAやFliGよりも多数の荷電残基をそれらの領域に内包する.その荷電残基に変異を導入することで,PomAにおいては七つの,FliGにおいては六つの荷電残基がモーターの回転力発生に関与することがわかり,特にPomA-E97とFliG-K284間の相互作用がNa+駆動型モーター特異的に重要であることが判明した49).またNa+駆動型モーターではH+駆動型モーターに比べ多くの荷電残基が固定子–回転子間相互作用に重要であり,固定子–回転子間の相互作用界面はこれまで提案されてきたものと比べ,非常に複雑であることがわかった.モーターの二つの回転方向(反時計回りと時計回り)に対してこれらの荷電残基の寄与を調べたところ,野生型やPomA上の変異体では両方向の回転速度は同等なのに対して,FliG上のK284やR317の変異体では反時計回りのみ著しく回転速度が低下するものがみられ,これらの残基は反時計回りの回転方向の構造をサポートするのに関わっている50).
これまで固定子はその分子構造がほとんど明らかになっていなかった.さまざまな細菌に由来するAサブユニットタンパク質を作製しその性質を比較することで,超好熱性細菌Aquifex aeolicus由来のMotAが安定かつ多量に精製できることが明らかとなり,化学的架橋実験などからMotAは単独で安定な四量体を形成することがわかった.この四量体MotAについて電子顕微鏡単粒子解析により構造決定を行い,界面活性剤の皮膜をまとった膜貫通領域と,トゲ状の突起を持つアーチ状の細胞質領域という二つの構造を明確に区別して観察することに成功した(図3D)51).
固定子が機能するためには,モーターの回転力に負けないようにしっかりと回転子の周囲に固定される必要がある.固定子はモーターに組み込まれるとペプチドグリカン層に結合することで固定される.最大で1ダース前後集合する固定子は,モーターに組み込まれたり外れたりして動的に入れ替わり,モーターから外れた固定子はイオンを流さなくなる.サルモネラのMotBの119番目のロイシンをプロリンに置換した変異(L119P)は固定子をモーターへと組み込まれやすくし,またモーターに組み込まれていなくても固定子のイオン透過活性を高いまま維持する36).このL119Pを導入した固定子は野生型の固定子と比べはるかに強くペプチドグリカンに結合する.X線結晶構造解析と核磁気共鳴(NMR)法を用いることで,MotBペリプラズム側領域の長いαヘリックス(α1)の構造変化を発見し,二重システイン変異を導入したジスルフィド架橋実験と組み合わせることで,α1が伸びた構造に変化することで固定子の一部が伸び上がり,プラグが開くと同時に隠れていたペプチドグリカン結合部位が露出し,それによって固定子が細胞壁にしっかり固定されることを明らかにした(図3D)52).
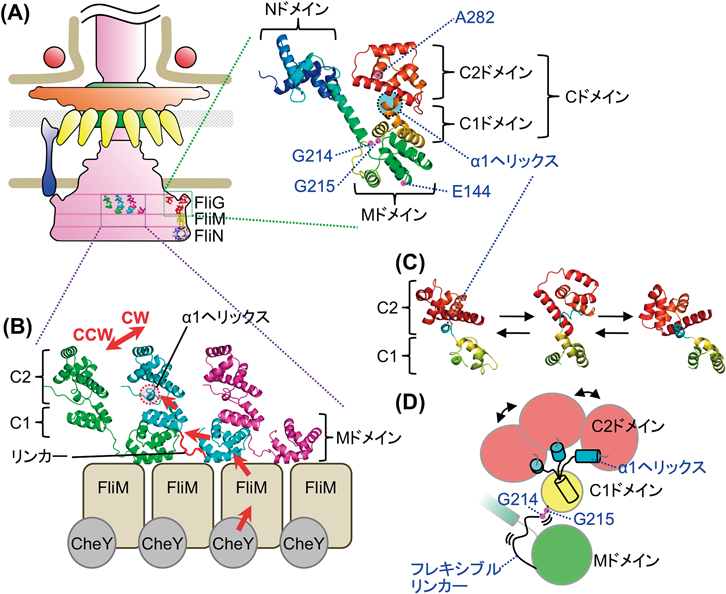
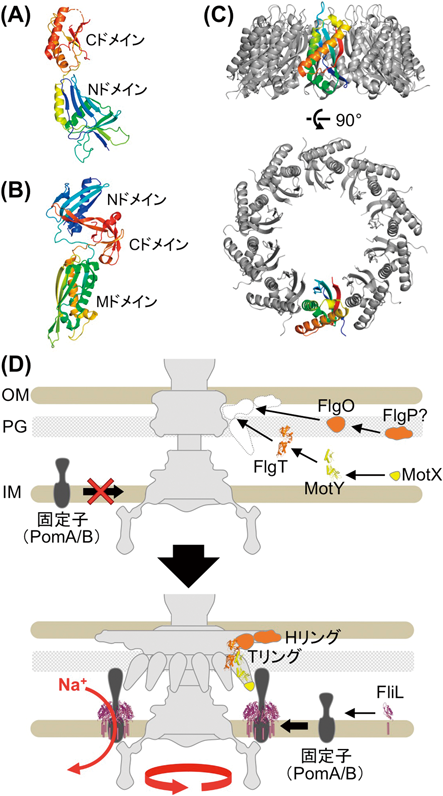
Cリングを構成するタンパク質FliG, FliM, FliNは互いに相互作用することで,大きなリング状構造を形成する.このリング構造が,回転力の発生あるいは回転方向を高速で切り替えるスイッチ機構を制御する.FliGは,アミノ末端(N末端)側ドメイン(Nドメイン),中間ドメイン(Mドメイン),C末端側ドメイン(Cドメイン)から構成される(図4A, B).V. alginolyticusのFliGのCドメインについて,NMR法と分子動力学(MD)解析によって構造を調べたところ,FliGのCドメインは一つに固定された構造にとどまるのではなく,主に3種類の構造を形成し,それらの構造間を動的に行き来することがわかった(図4C)53).一方,モーターの回転方向が時計回りに固定されるFliGの282番目のアラニン(A)をTに置換した変異を導入すると,そのような複数の構造はみられなかった.FliGのCドメインは三つのαヘリックスを有するC1ドメインと,六つのαヘリックスを有するC2ドメインから構成され,C2ドメインの1番目のαヘリックスがC1-C2ドメイン間をつなぐ柔軟性に富んだ“蝶番”として働くことで,Cドメイン全体の動的な構造変化を生み出し,モーターの回転方向の変換制御を担っていることが明らかとなった(図4D).他にも,モーターの回転方向に影響するFliGの変異を探索したところ,214番目グリシン(G)をセリンに置換した変異(G214S)はモーターの回転の反時計回りへの偏り,215番目のGをAに置換した変異(G215A)は時計回りへの固定,さらには144番目のEをDに置換した変異は頻繁な回転方向の切り替えを,それぞれ引き起こすことがわかった54, 55).MドメインとCドメインの間にはグリシン残基が並んだ柔軟性に富んだ部分(フレキシブルリンカー)が存在し,G214とG215がこの部分に相当する.FliGのMドメインとCドメインについてNMR法とMD解析によりこれら3種類の変異の影響を調べたところ,G214SおよびG215A変異により構造の柔軟性が制限されており,フレキシブルリンカーがFliG構造の柔軟性のために決定的な役割を担っていることがわかった(図4D).G214SおよびG215A変異体のモーター構造をクライオ電子顕微鏡を用いて解析したところ,Cリング構造に違いがあることが明らかになった(投稿準備中).一方,E144D変異体では野生型と類似の構造特性を持つが,外部環境からのシグナルへの応答に対して異常を示す.E144はFliMとの結合部位に存在し(図4A),FliMを介した走化性シグナルが正しく伝わらないため回転方向の切り替え頻度に影響することがわかった.以上のように,FliGの内在的な動的構造特性およびFliG–FliM間の綿密な相互作用が,モーター回転方向の瞬時かつ精密な切り替えを可能にしていることが明らかになった.
7. V. alginolyticusのNa+駆動型べん毛に特徴的な構造
前述のとおりV. alginolyticusのNa+駆動型べん毛は,大腸菌などのH+駆動型べん毛にはない特徴的な構造を内包する(図2A, C).特にLリングとPリングの外側や下側に存在するHリングとTリングはNa+駆動型モーター特有の高速回転に寄与し,HリングやTリングが形成されない変異体ではモーターは回転しない12, 56).Tリングは著者らがMotX, MotYと名づけた2種類のタンパク質からなり,固定子をモーターに安定に組み込ませるために寄与する.MotYはべん毛基部体に直接,MotXはMotYを介して基部体に結合する.X線結晶構造解析からMotYは,基部体およびMotXと相互作用するNドメインと,ペプチドグリカン結合ドメインであるCドメインからなることがわかった(図5A)12).一方HリングはFlgTやFlgOなどのタンパク質から構成される56).FlgTのX線結晶構造はNドメイン,Mドメイン,Cドメインの三つのドメインから構成される(図5B).Mドメインは基部体と結合しMotXとMotYの集合のための足場として,NドメインはHリングの外膜部分の形成のために,CドメインはFlgTが作るリング構造の安定化に,それぞれ寄与する(図5D).
著者らは,V. alginolyticusの多べん毛変異体の細胞をクライオ電子線トモグラフィーで観察することで,“sheath(鞘)”と呼ばれるべん毛を覆う膜構造の外側に存在する新たな構造を見いだし,Oリングと名づけた(図6A)57).また,HリングとMSリングとの距離には多様性があり,Hリングは機械モーターの“ワッシャー”のように可変であると考えられる.さらにTリングの構造を観察し,13回回転対称の構造を持つことがわかった(図6A).これらを介すことでV. alginolyticusのNa+駆動型モーターでは,軸受け構造が強化されるとともに,固定子はペプチドグリカン層と軸受けに強固に固定される.
8. FliL:真核生物のセンサータンパク質との共通構造
べん毛モーターは単なる運動器官としてだけでなく,べん毛へとかかる負荷を感知するセンサー(いわゆるメカノセンサー)としての一面も持つ.つまり,周囲の環境が高い粘性を持つ場合(たとえばバイオフィルムを形成しているときや,宿主の細胞などの表面に固着しているとき),液体中などの低い粘性のときに比べ,べん毛モーターは高い出力を出す.この応答は主に,モーターへと集合する固定子の数が制御されることによって行われる.モーターの高出力回転を生み出すのに必要なべん毛モーター構成因子としてFliLが知られる.FliLがないとき,多くの場合,細菌は高粘性下でのべん毛運動能を損なう.FliLは1回膜貫通型のタンパク質で,固定子と相互作用してその機能を補助すると考えられており,またFliLは環境中の粘性を感知する機能も持つことが知られている.最近,著者らはX線結晶構造解析によりFliLのペリプラズム側領域の構造を明らかにし(図5C),10分子のFliLが固定子の周囲へと環状に集合して,FliL–固定子複合体を形成するモデルを提唱した58)(図5D).この際,FliLリングの内壁に存在する二つの保存されたバリン残基が,固定子との相互作用および固定子の活性制御に関与することがわかった.さらに興味深いことに,FliLは哺乳類の神経細胞や赤血球の中などにも存在するストマチン様タンパク質と共通の構造を持つことが明らかになった.ストマチン様タンパク質は膜貫通型あるいは膜結合型のタンパク質ファミリーで,機械刺激や酸刺激などのさまざまな外環境からのシグナルを受け取り,多様な膜タンパク質の活性を制御することが知られている59).FliLはこれまで見つかっていなかった新規ストマチン様タンパク質であり,この発見は,全生物に保存されたストマチン様タンパク質による膜タンパク質制御の共通メカニズムの解明のための大きな手がかりとして期待できる.
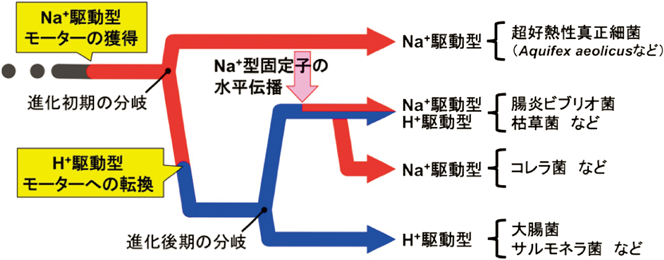
上述のように,べん毛モーターの研究は大腸菌やサルモネラ,ビブリオなどを含めたいくつかの細菌においてよく研究されているが,その機能についてより一般的かつ詳細に解析するためには,より多岐に及んだ細菌種へと視野を広げなければならない.アクウィフェクス門(Aquificae)の細菌は温泉や海底火山周辺に生息する細菌であり,16SリボソームRNAの解析から真正細菌の進化系統上で最も初期に分岐した細菌であると知られている.Aquifex aeolicusは代表的なアクウィフェクス門の細菌であり,その生育可能温度は67~95°C(至適生育温度は85°C)という超好熱性の細菌である60).A. aeolicusの全ゲノム配列はすでに報告されており61),そのリコンビナントタンパク質は構造解析の材料としてよく用いられている(Protein Data BankにはA. aeolicus由来のタンパク質構造が508件登録:2019年6月現在).A. aeolicusの近縁種であるAquifex pyrophilusは,細胞の極に複数本のべん毛を持つことが報告されている62).著者らはA. aeolicusを85°Cで培養,光学顕微鏡および電子顕微鏡を用いて観察し,A. aeolicusが細胞の極に生えた1本のべん毛を使って,高温環境で速い速度で泳ぐことを観察した(低温環境では非常にゆっくりとしか泳がない)63).このA. aeolicusが持つべん毛モーターの回転メカニズムの詳細を明らかにするため,菌体の調製が容易な大腸菌と組み合わせることでA. aeolicusの固定子の機能評価を行った.A. aeolicusと大腸菌の固定子を融合させたキメラ型固定子を大腸菌内で発現させたところ,A. aeolicusの固定子はNa+流を駆動力としており,原始細菌のべん毛モーターはNa+を使用していたことが示唆された.さまざまな細菌の固定子の遺伝子配列を比較した系統学的解析から,細菌の祖先はNa+を使ってモーターのエネルギー変換を行っていたこと,そのエネルギー変換機構は細菌の祖先から現在まで統一されていること,進化の過程においてNa+駆動型の固定子が一部の細菌へと水平伝播されたことなどがわかった(図7).
著者の一人である本間が,東京大学植物学教室の飯野徹雄先生の研究室で細菌べん毛の研究をスタートして40年が過ぎようとしている.「べん毛が理解できたときは生命を理解できたときだ」というのが,べん毛を“回転する生命”という見地から研究を行っていた飯野先生の言葉である.そのべん毛研究において,タンパク質で作られたナノ回転モーターをもう少しで原子レベルの構造解析ができそうなところまで来ている.共著者の竹川は,本間の研究室に所属して学位をとり,現在,大阪大学で助教としてべん毛研究を進めている.イオン流を回転エネルギーに変換する生物マシンの構造と機能の解明は間近であると思う.
謝辞Acknowledgments
名古屋大学理学部分子生物学科の分子第4講座から,今は,大学院理学研究科生命理学専攻生体膜機能グループと名前は変わったが,多くの共同研究者や学生がナトリウム駆動型のべん毛研究に携わってくれた.この最近の成果を支えてくれた紹介できなかった論文も多くある.名古屋大学のすばらしい研究環境と共同研究者に感謝したい.
引用文献References
1) von Ballmoos, C., Wiedenmann, A., & Dimroth, P. (2009) Essentials for ATP synthesis by F1F0 ATP synthases. Annu. Rev. Biochem., 78, 649–672.
2) Shrivastava, A., Lele, P.P., & Berg, H.C. (2015) A rotary motor drives Flavobacterium gliding. Curr. Biol., 25, 338–341.
3) Terashima, H., Kojima, S., & Homma, M. (2008) Flagellar motility in bacteria structure and function of flagellar motor. Int. Rev. Cell Mol. Biol., 270, 39–85.
4) Leake, M.C., Chandler, J.H., Wadhams, G.H., Bai, F., Berry, R.M., & Armitage, J.P. (2006) Stoichiometry and turnover in single, functioning membrane protein complexes. Nature, 443, 355–358.
5) Reid, S.W., Leake, M.C., Chandler, J.H., Lo, C.J., Armitage, J.P., & Berry, R.M. (2006) The maximum number of torque-generating units in the flagellar motor of Escherichia coli is at least 11. Proc. Natl. Acad. Sci. USA, 103, 8066–8071.
6) Li, N., Kojima, S., & Homma, M. (2011) Sodium-driven motor of the polar flagellum in marine bacteria Vibrio. Genes Cells, 16, 985–999.
7) Ito, M. & Takahashi, Y. (2017) Nonconventional cation-coupled flagellar motors derived from the alkaliphilic Bacillus and Paenibacillus species. Extremophiles, 21, 3–14.
8) Ueno, T., Oosawa, K., & Aizawa, S.I. (1992) M ring, S ring and proximal rod of the flagellar basal body of Salmonella typhimurium are composed of subunits of a single protein, FliF. J. Mol. Biol., 227, 672–677.
9) Francis, N.R., Irikura, V.M., Yamaguchi, S., DeRosier, D.J., & Macnab, R.M. (1992) Localization of the Salmonella typhimurium flagellar switch protein FliG to the cytoplasmic M-ring face of the basal body. Proc. Natl. Acad. Sci. USA, 89, 6304–6308.
10) Homma, M., Aizawa, S.-I., Dean, G.E., & Macnab, R.M. (1987) Identification of the M-ring protein of the flagellar motor of Salmonella typhimurium. Proc. Natl. Acad. Sci. USA, 84, 7483–7487.
11) Schoenhals, G.J. & Macnab, R.M. (1996) Physiological and biochemical analyses of FlgH, a lipoprotein forming the outer membrane L ring of the flagellar basal body of Salmonella typhimurium. J. Bacteriol., 178, 4200–4207.
12) Terashima, H., Fukuoka, H., Yakushi, T., Kojima, S., & Homma, M. (2006) The Vibrio motor proteins, MotX and MotY, are associated with the basal body of Na-driven flagella and required for stator formation. Mol. Microbiol., 62, 1170–1180.
13) Terashima, H., Koike, M., Kojima, S., & Homma, M. (2010) The flagellar basal body-associated protein FlgT is essential for a novel ring structure in the sodium-driven Vibrio motor. J. Bacteriol., 192, 5609–5615.
14) Zhao, X., Zhang, K., Boquoi, T., Hu, B., Motaleb, M.A., Miller, K.A., James, M.E., Charon, N.W., Manson, M.D., Norris, S.J., et al. (2013) Cryoelectron tomography reveals the sequential assembly of bacterial flagella in Borrelia burgdorferi. Proc. Natl. Acad. Sci. USA, 110, 14390–14395.
15) Francis, N.R., Sosinsky, G.E., Thomas, D., & Derosier, D.J. (1994) Isolation, characterization and structure of bacterial flagellar motors containing the switch complex. J. Mol. Biol., 235, 1261–1270.
16) Suzuki, H., Yonekura, K., & Namba, K. (2004) Structure of the rotor of the bacterial flagellar motor revealed by electron cryomicroscopy and single–particle image analysis. J. Mol. Biol., 337, 105–113.
17) Thomas, D.R., Morgan, D.G., & DeRosier, D.J. (1999) Rotational symmetry of the C ring and a mechanism for the flagellar rotary motor. Proc. Natl. Acad. Sci. USA, 96, 10134–10139.
18) Sato, K. & Homma, M. (2000) Multimeric structure of PomA, the Na+-driven polar flagellar motor component of Vibrio alginolyticus. J. Biol. Chem., 275, 20223–20228.
19) Yorimitsu, T., Kojima, M., Yakushi, T., & Homma, M. (2004) Multimeric structure of the PomA/PomB channel complex in the Na+-driven flagellar motor of Vibrio alginolyticus. J. Biochem., 135, 43–51.
20) Kojima, S. & Blair, D.F. (2004) Solubilization and purification of the MotA/MotB complex of Escherichia coli. Biochemistry, 43, 26–34.
21) Asai, Y., Kojima, S., Kato, H., Nishioka, N., Kawagishi, I., & Homma, M. (1997) Putative channel components for the fast-rotating sodium-driven flagellar motor of a marine bacterium. J. Bacteriol., 179, 5104–5110.
22) Zhou, J.D., Fazzio, R.T., & Blair, D.F. (1995) Membrane topology of the MotA protein of Escherichia coli. J. Mol. Biol., 251, 237–242.
23) Zhou, J., Sharp, L.L., Tang, H.L., Lloyd, S.A., Billings, S., Braun, T.F., & Blair, D.F. (1998) Function of protonatable residues in the flagellar motor of Escherichia coli: A critical role for Asp 32 of MotB. J. Bacteriol., 180, 2729–2735.
24) Morimoto, Y.V., Nakamura, S., Hiraoka, K.D., Namba, K., & Minamino, T. (2013) Distinct roles of highly conserved charged residues at the MotA-FliG interface in bacterial flagellar motor rotation. J. Bacteriol., 195, 474–481.
25) Sharp, L.L., Zhou, J.D., & Blair, D.F. (1995) Features of MotA proton channel structure revealed by tryptophan-scanning mutagenesis. Proc. Natl. Acad. Sci. USA, 92, 7946–7950.
26) Braun, T.F. & Blair, D.F. (2001) Targeted disulfide cross-linking of the MotB protein of Escherichia coli: Evidence for two H+ channels in the stator complex. Biochemistry, 40, 13051–13059.
27) Braun, T.F., Al-Mawsawi, L.Q., Kojima, S., & Blair, D.F. (2004) Arrangement of core membrane segments in the MotA/MotB proton-channel complex of Escherichia coli. Biochemistry, 43, 35–45.
28) Yakushi, T., Kojima, M., & Homma, M. (2004) Isolation of Vibrio Na+-driven flagellar motor complex composed of PomA and PomB solubilized by sucrose monocaprate. Microbiology, 150, 911–920.
29) Sudo, Y., Kitade, Y., Furutani, Y., Kojima, M., Kojima, S., Homma, M., & Kandori, H. (2009) Interaction between Na+ ion and carboxylates of the PomA–PomB stator unit studied by ATR-FTIR spectroscopy. Biochemistry, 48, 11699–11705.
30) Terashima, H., Kojima, S., & Homma, M. (2010) Functional transfer of an essential aspartate for the ion-binding site in the stator proteins of the bacterial flagellar motor. J. Mol. Biol., 397, 689–696.
31) Takekawa, N., Terauchi, T., Morimoto, Y.V., Minamino, T., Lo, C.J., Kojima, S., & Homma, M. (2013) Na+ conductivity of the Na+-driven flagellar motor complex composed of unplugged wild-type or mutant PomB with PomA. J. Biochem., 153, 441–451.
32) Terauchi, T., Terashima, H., Kojima, S., & Homma, M. (2011) A conserved residue, PomB-F22, in the transmembrane segment of the flagellar stator complex, has a critical role in conducting ions and generating torque. Microbiology, 157, 2422–2432.
33) Stolz, B. & Berg, H.C. (1991) Evidence for interactions between MotA and MotB, torque-generating elements of the flagellar motor of Escherichia coli. J. Bacteriol., 173, 7033–7037.
34) Hosking, E.R., Vogt, C., Bakker, E.P., & Manson, M.D. (2006) The Escherichia coli MotAB proton channel unplugged. J. Mol. Biol., 364, 921–937.
35) Morimoto, Y.V., Che, Y.S., Minamino, T., & Namba, K. (2010) Proton-conductivity assay of plugged and unplugged MotA/B proton channel by cytoplasmic pHluorin expressed in Salmonella. FEBS Lett., 584, 1268–1272.
36) Li, N., Kojima, S., & Homma, M. (2011) Characterization of the periplasmic region of PomB, a Na+-driven flagellar stator protein in Vibrio alginolyticus. J. Bacteriol., 193, 3773–3784.
37) Fukuoka, H., Wada, T., Kojima, S., Ishijima, A., & Homma, M. (2009) Sodium-dependent dynamic assembly of membrane complexes in sodium-driven flagellar motors. Mol. Microbiol., 71, 825–835.
38) Kojima, S., Imada, K., Sakuma, M., Sudo, Y., Kojima, C., Minamino, T., Homma, M., & Namba, K. (2009) Stator assembly and activation mechanism of the flagellar motor by the periplasmic region of MotB. Mol. Microbiol., 73, 710–718.
39) Kojima, S., Nonoyama, N., Takekawa, N., Fukuoka, H., & Homma, M. (2011) Mutations targeting the C-terminal domain of FliG can disrupt motor assembly in the Na+-driven flagella of Vibrio alginolyticus. J. Mol. Biol., 414, 62–74.
40) Morimoto, Y.V., Nakamura, S., Kami-ike, N., Namba, K., & Minamino, T. (2010) Charged residues in the cytoplasmic loop of MotA are required for stator assembly into the bacterial flagellar motor. Mol. Microbiol., 78, 1117–1129.
41) Onoue, Y., Iwaki, M., Shinobu, A., Nishihara, Y., Iwatsuki, H., Terashima, H., Kitao, A., Kandori, H., & Homma, M. (2019) Essential ion binding residues for Na+ flow in stator complex of the Vibrio flagellar motor. Sci. Rep. (印刷中).
42) Sowa, Y. & Berry, R.M. (2008) Bacterial flagellar motor. Q. Rev. Biophys., 41, 103–132.
43) Zhou, J.D., Lloyd, S.A., & Blair, D.F. (1998) Electrostatic interactions between rotor and stator in the bacterial flagellar motor. Proc. Natl. Acad. Sci. USA, 95, 6436–6441.
44) Yorimitsu, T., Sowa, Y., Ishijima, A., Yakushi, T., & Homma, M. (2002) The systematic substitutions around the conserved charged residues of the cytoplasmic loop of Na+-driven flagellar motor component PomA. J. Mol. Biol., 320, 403–413.
45) Yorimitsu, T., Mimaki, A., Yakushi, T., & Homma, M. (2003) The conserved charged residues of the C-terminal region of FliG, a rotor component of Na+-driven flagellar motor. J. Mol. Biol., 334, 567–583.
46) Yakushi, T., Yang, J., Fukuoka, H., Homma, M., & Blair, D.F. (2006) Roles of charged residues of rotor and stator in flagellar rotation: comparative study using H+-driven and Na+-driven motors in Escherichia coli. J. Bacteriol., 188, 1466–1472.
47) Fukuoka, H., Yakushi, T., & Homma, M. (2004) Concerted effects of amino acid substitutions in conserved charged residues and other residues in the cytoplasmic domain of PomA, a stator component of Na+-driven flagella. J. Bacteriol., 186, 6749–6758.
48) Obara, M., Yakushi, T., Kojima, S., & Homma, M. (2008) Roles of charged residues in the C-terminal region of PomA, a stator component of the Na+-driven flagellar motor. J. Bacteriol., 190, 3565–3571.
49) Takekawa, N., Kojima, S., & Homma, M. (2014) Contribution of many charged residues at the stator-rotor interface of the Na+-driven flagellar motor to torque generation in Vibrio alginolyticus. J. Bacteriol., 196, 1377–1385.
50) Onoue, Y., Takekawa, N., Nishikino, T., Kojima, S., & Homma, M. (2018) The role of conserved charged residues in the bidirectional rotation of the bacterial flagellar motor. MicrobiologyOpen, 7, 00587.
51) Takekawa, N., Terahara, N., Kato, T., Gohara, M., Mayanagi, K., Hijikata, A., Onoue, Y., Kojima, S., Shirai, T., Namba, K., et al. (2016) The tetrameric MotA complex as the core of the flagellar motor stator from hyperthermophilic bacterium. Sci. Rep., 6, 31526.
52) Kojima, S., Takao, M., Almira, G., Kawahara, I., Sakuma, M., Homma, M., Kojima, C., & Imada, K. (2018) The helix rearrangement in the periplasmic domain of the flagellar stator B subunit activates peptidoglycan binding and ion influx. Structure, 26, 590–598.e595.
53) Miyanoiri, Y., Hijikata, A., Nishino, Y., Gohara, M., Onoue, Y., Kojima, S., Kojima, C., Shirai, T., Kainosho, M., & Homma, M. (2017) Structural and functional analysis of the C-Terminal region of FliG, an essential motor component of Vibrio Na+-driven flagella. Structure, 25, 1540–1548.
54) Nishikino, T., Hijikata, A., Miyanoiri, Y., Onoue, Y., Kojima, S., Shirai, T., & Homma, M. (2018) Rotational direction of flagellar motor from the conformation of FliG middle domain in marine Vibrio. Sci. Rep., 8, 17793.
55) Nishikino, T., Zhu, S., Takekawa, N., Kojima, S., Onoue, Y., & Homma, M. (2016) Serine suppresses the motor function of a periplasmic PomB mutation in the Vibrio flagella stator. Genes Cells, 21, 505–516.
56) Terashima, H., Li, N., Sakuma, M., Koike, M., Kojima, S., Homma, M., & Imada, K. (2013) Insight into the assembly mechanism in the supramolecular rings of the sodium-driven Vibrio flagellar motor from the structure of FlgT. Proc. Natl. Acad. Sci. USA, 110, 6133–6138.
57) Zhu, S., Nishikino, T., Hu, B., Kojima, S., Homma, M., & Liu, J. (2017) Molecular architecture of the sheathed polar flagellum in Vibrio alginolyticus. Proc. Natl. Acad. Sci. USA, 114, 10966–10971.
58) Takekawa, N., Isumi, M., Terashima, H., Zhu, S., Nishino, Y., Sakuma, M., Kojima, S., Homma, M., & Imada, K. (2019) Structure of Vibrio FliL, a new stomatin-like protein that assists the bacterial flagellar motor function. MBio, 10, e00292–e19.
59) Lapatsina, L., Brand, J., Poole, K., Daumke, O., & Lewin, G.R. (2012) Stomatin-domain proteins. Eur. J. Cell Biol., 91, 240–245.
60) Reysenbach, A.L., Banta, A., Civello, S., Daly, J., Mitchel, K., Lalonde, S., Konhauser, K.O., Rodman, A., Rusterholtz, K., & Takacs-Vesbach, C. (2005) The Aquificales of Yellowstone National Park, in Geothermal biology and geochemistry in Yellowstone National Park (Inskeep & McDermott eds.), pp. 129–142, Thermal Biology Institute, Montana State University, Bozeman.
61) Deckert, G., Warren, P.V., Gaasterland, T., Young, W.G., Lenox, A.L., Graham, D.E., Overbeek, R., Snead, M.A., Keller, M., Aujay, M., et al. (1998) The complete genome of the hyperthermophilic bacterium Aquifex aeolicus. Nature, 392, 353–358.
62) Behammer, W., Shao, Z.X., Mages, W., Rachel, R., Stetter, K.O., & Schmitt, R. (1995) Flagellar structure and hyperthermophily: Analysis of a single flagellin gene and its product in Aquifex pyrophilus. J. Bacteriol., 177, 6630–6637.
63) Takekawa, N., Nishiyama, M., Kaneseki, T., Kanai, T., Atomi, H., Kojima, S., & Homma, M. (2015) Sodium-driven energy conversion for flagellar rotation of the earliest divergent hyperthermophilic bacterium. Sci. Rep., 5, 12711.
著者紹介Author Profile
 竹川 宜宏(たけかわ のりひろ)
竹川 宜宏(たけかわ のりひろ)大阪大学大学院理学研究科助教.博士(理学).
略歴1988年愛知県に生る.2011年名古屋大学理学部卒業.15年同大学院理学研究科博士後期課程修了.同年同研究科日本学術振興会特別研究員(PD).16年大阪大学大学院理学研究科同特別研究員(PD).19年より現職.
研究テーマと抱負細菌のべん毛の構築と機能に関するタンパク質の構造・変異体の研究を行っている.
 本間 道夫(ほんま みちお)
本間 道夫(ほんま みちお)名古屋大学大学院理学研究科教授.理学博士.
略歴1955年新潟県に生る.79年国際基督教大学教養学部卒業,85年東京大学大学院理学研究科植物学教室博士課程修了.同年4月米国エール大学分子生物物理生化学科博士研究員.88年9月名古屋大学付属病態制御研究施設講師.92年9月名古屋大学理学研究科助教授.97年11月より現職.
研究テーマと抱負大学院から,べん毛モーター回転機構解明を目指して研究を始めた.学位を取得した後,米国に留学した.名古屋大学医学部に職を得て,病原性酵母の研究を行ったが,理学部に移ることができ,べん毛研究を再開した.あと定年まで約1年半,最後までアクティブに研究を進めたい.
ウェブサイトhttp://bunshi4.bio.nagoya-u.ac.jp/~bunshi4/fourth.html
趣味サイエンス,水泳,フルート(最近は吹いていない).