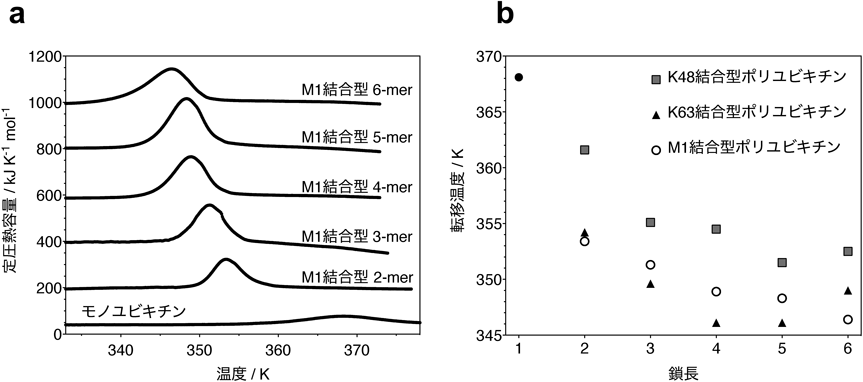
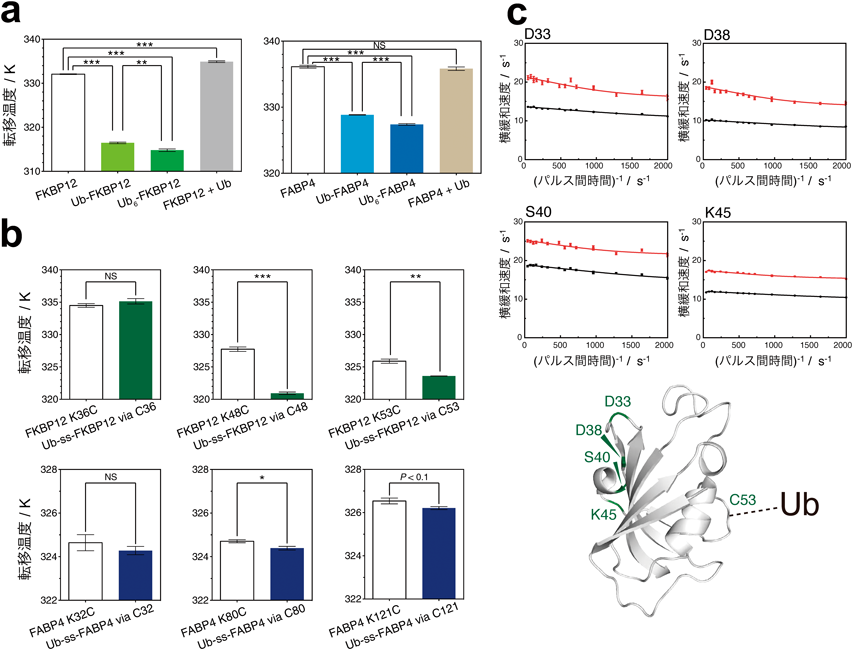
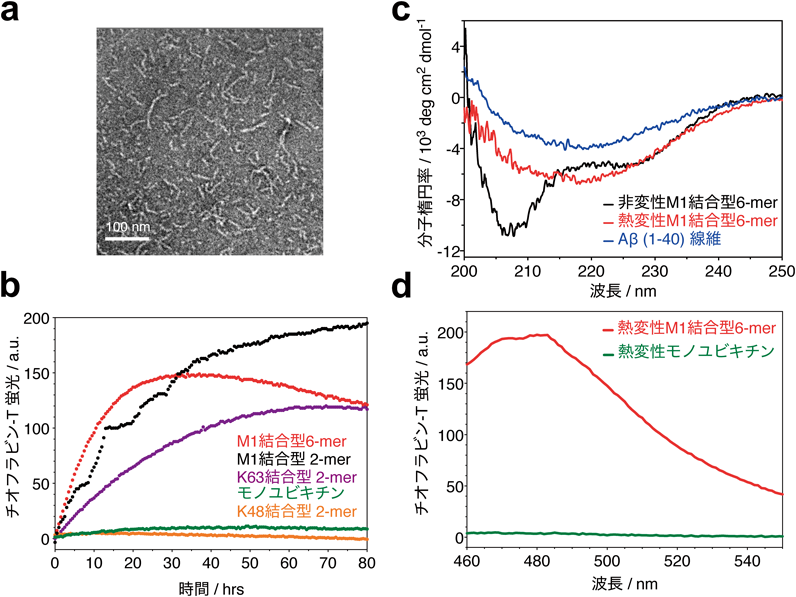
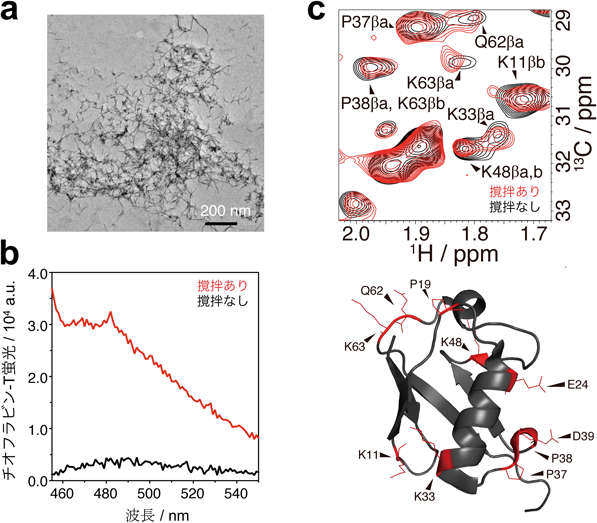
ユビキチン鎖の鎖長に依存した物性と分解Chain-length dependent physical properties and degradation of ubiquitin chains
1 京都大学大学院工学研究科分子工学専攻Department of Molecular Engineering, Graduate School of Engineering, Kyoto University ◇ 〒615–8510 京都府京都市西京区京都大学桂 ◇ Kyoto University Katsura, Nishikyo-ku, Kyoto, 615–8510, Japan
2 量子科学技術研究開発機構量子生命科学領域領域研究統括Scientific Director General, Institute for Quantum Life Science, National Institutes for Quantum and Radiological Science and Technology ◇ 〒263–8555 千葉県稲毛区穴川4–9–1 ◇ 4–9–1 Anagawa, Inage-ku, Chiba, 263–8555, Japan