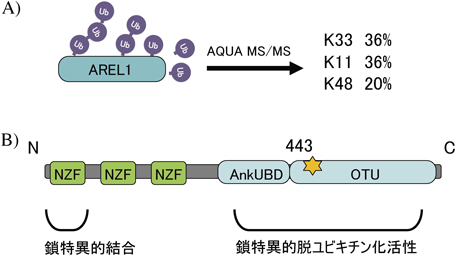
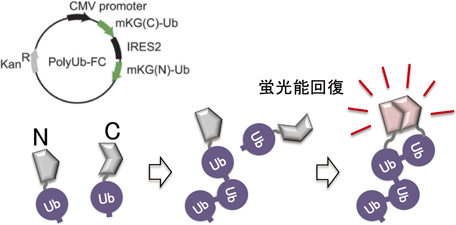
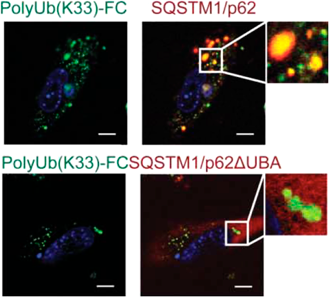
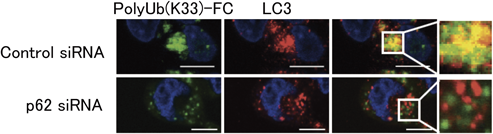
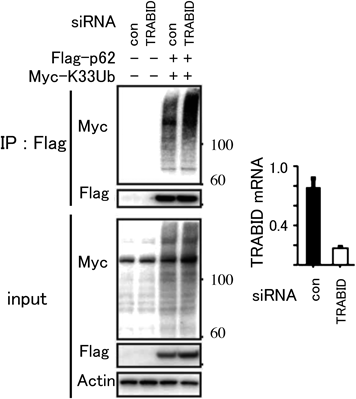
K33ユビキチン鎖の機能The role of K33-linked polyubiquitin
東京医科歯科大学医学部附属病院消化器内科Department of Gastroenterology and Hepatology Medical Hospital,Tokyo Medical and Dental University (TMDU) ◇ 〒113–8510 東京都文京区湯島1–5–45 ◇ 1–5–45 Yushima, Bunkyo-ku, Tokyo, 113–8510, Japan