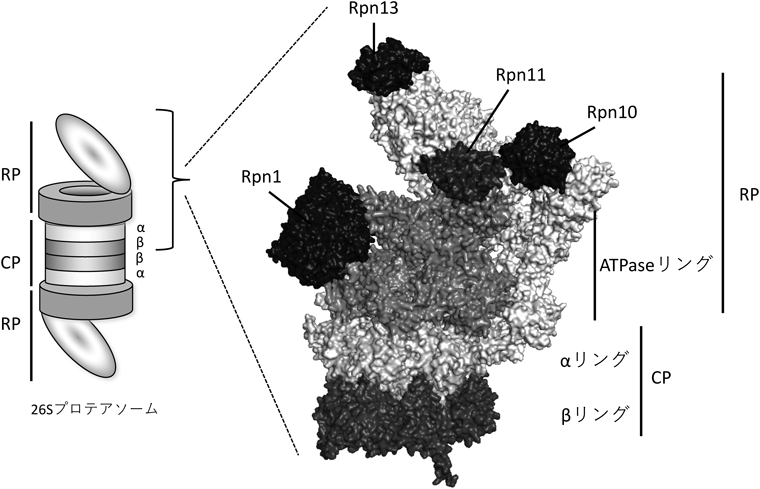
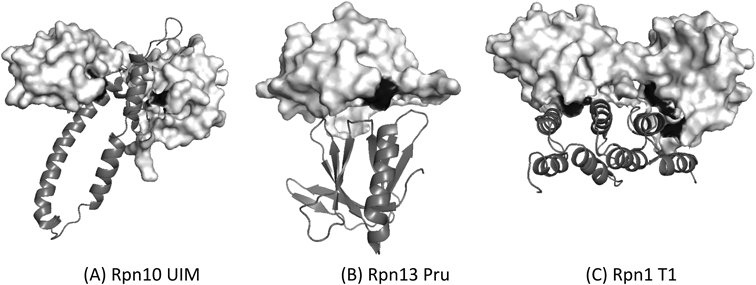
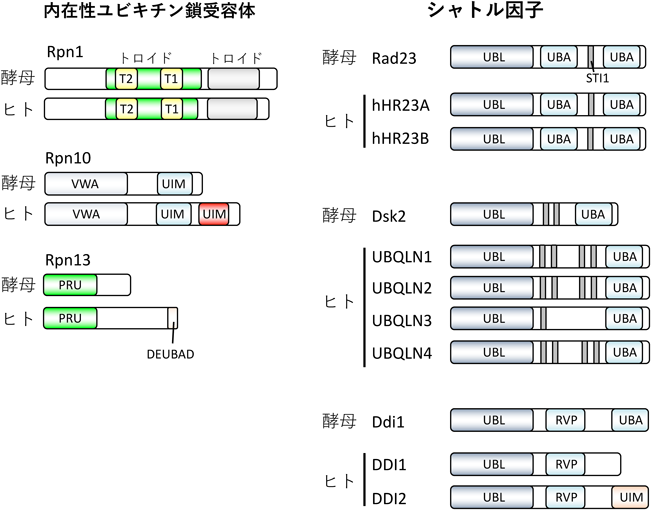
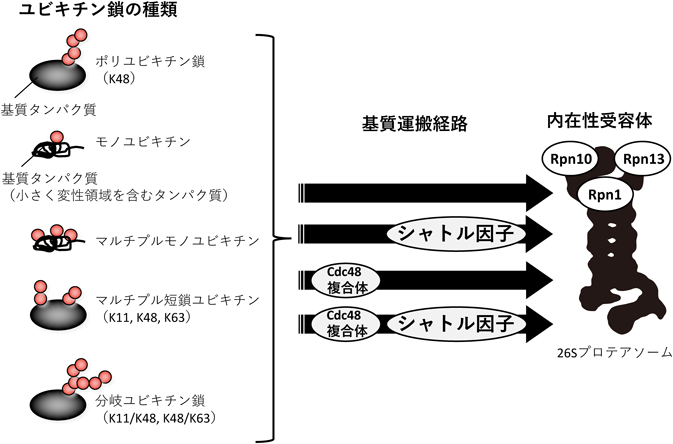
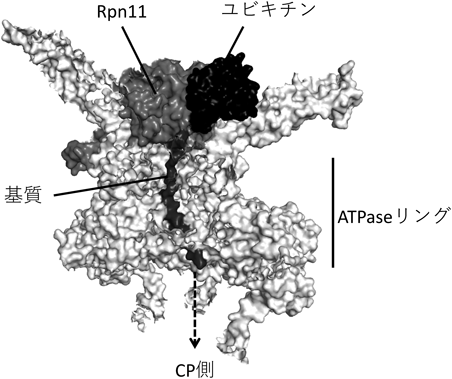
26Sプロテアソームによるユビキチン化タンパク質認識・分解機構Recognition and degradation of ubiquitinated proteins by the 26S proteasome
東京大学大学院薬学系研究科蛋白質代謝学教室Laboratory of Protein Metabolism, Graduate School of Pharmaceutical Sciences, The University of Tokyo ◇ 〒113–0033 東京都文京区本郷7–3–1東京大学薬学部総合研究棟7階 ◇ 7–3–1 Hongo, Bunkyo-ku, Tokyo 113–0033, Japan