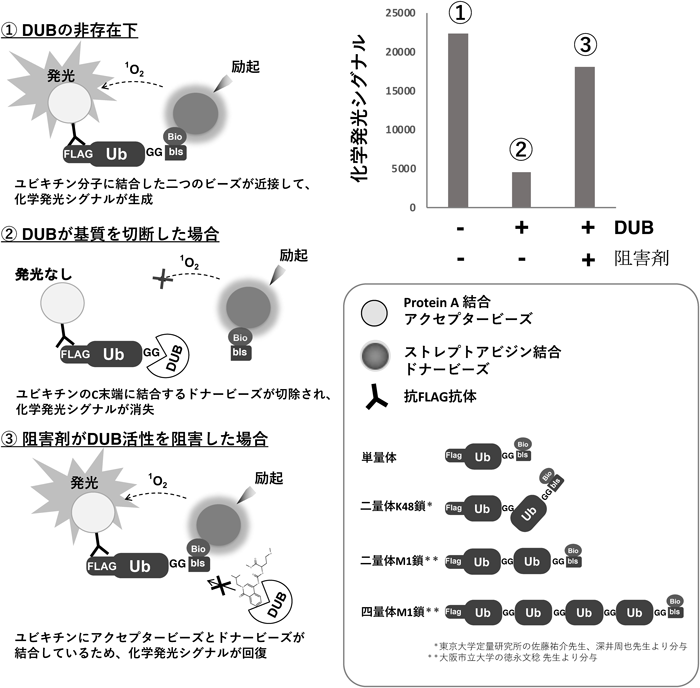
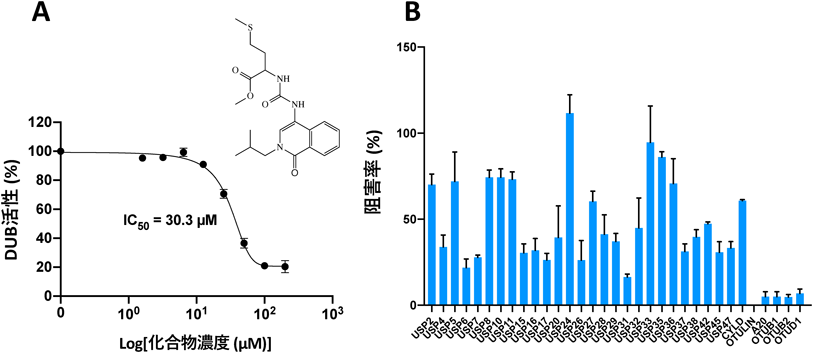
ヒト脱ユビキチン化酵素タンパク質アレイの開発とその応用例Development and application of a protein array of human deubiquitinating enzyme
1 愛媛大学プロテオサイエンスセンターProteo-Science Center, Ehime University ◇ 愛媛県松山市文京町3番 ◇ Japan, 3 Bunkyo-cho, Matsuyama, Ehime 790–8577, Japan
2 大阪市立大学大学院医学研究科分子病態学Department of Pathobiochemistry, Graduate School of Medicine, Osaka City University ◇ 大阪府大阪市阿倍野区旭町1–4–3 ◇ 1–4–3 Asahi-machi, Abeno-ku, Osaka 545–8585, Japan