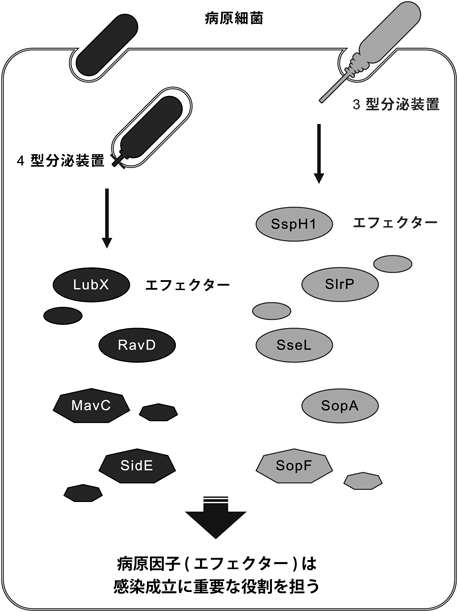
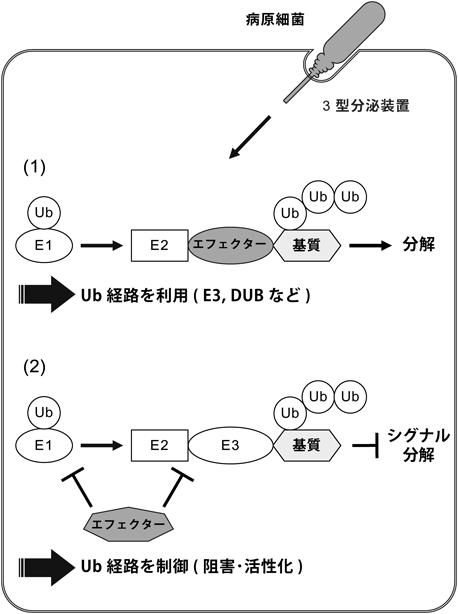
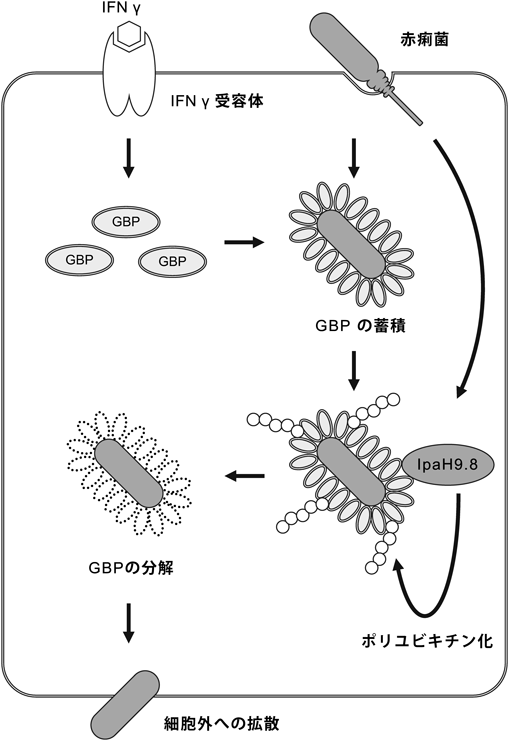
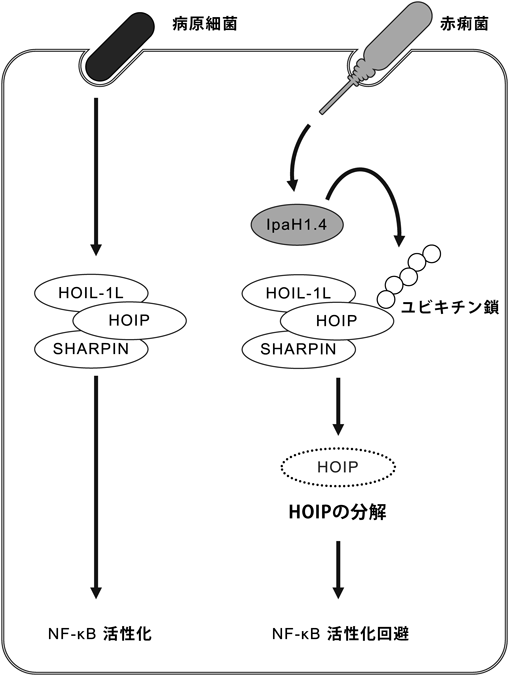
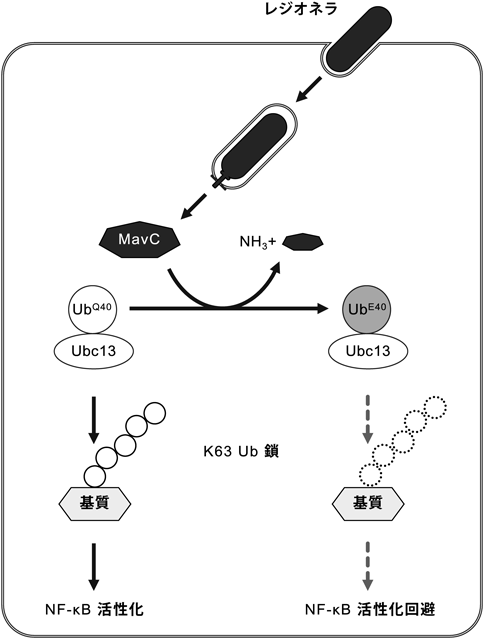
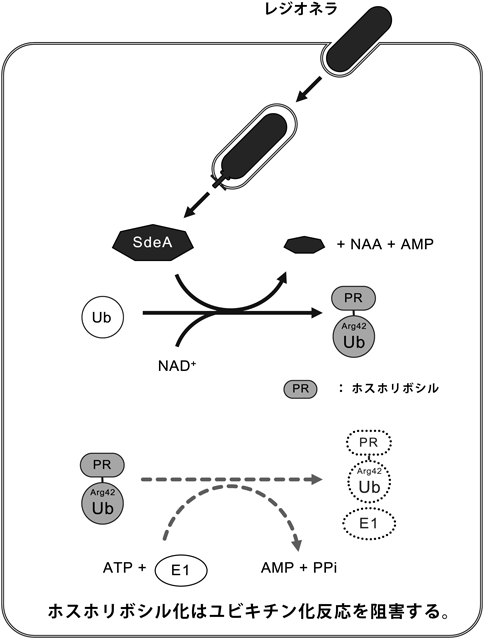
病原細菌によるユビキチン修飾系撹乱の分子機構Hijacking of the host ubiquitin system by pathogenic bacteria
1 京都大学・大学院医学研究科・細胞機能制御学Department of Molecular and Cellular Physiology, Graduate School of Medicine, Kyoto University ◇ 〒606–8501 京都市左京区吉田近衛町 ◇ Yoshida-konoe-cho, Sakyo-ku, Kyoto-shi, Kyoto 606–8501, Japan
2 京都大学・白眉センターThe Hakubi Center for Advanced Research, Kyoto University ◇ 〒606–8501 京都市左京区吉田近衛町 ◇ Yoshida-konoe-cho, Sakyo-ku, Kyoto-shi, Kyoto 606–8501, Japan