多くの生命現象は,分子構造から組織構築に至る多階層での形の変化を伴う.細胞表層に発達するアクチン細胞骨格は,アクチン重合やミオシンモーターの働きで力を発生するとともに,物理ストレスを含めた種々の外来刺激に応答し,その形態を変化させる.この内外の力のバランスにより,細胞や組織が形づくられる.
アクチンリモデリングのような動的な複雑系においては,ある分子の活性を変動させた場合,系の挙動が広く変化し,分子の直接作用を超えた範囲にまで効果が波及することがある.このような複雑系における個々の分子の働きを正確に捕捉するためには,システムの揺さぶりに応答して刻々と変化する個々の分子現象を捉えることが大きな意味を持つ.
この目的において,細胞内蛍光単分子イメージングは大きなポテンシャルを持つ.当初この手法は,EGFPを融合したアクチンを用いて実現された.しかし,プロセッシブ(重合するアクチン線維端に結合したまま移動し伸長速度を制御する)なアクチン重合促進分子であるフォルミンファミリーが作るアクチン線維に,蛍光タンパク質融合アクチンは取り込まれにくい.現在に至るまで,単分子イメージングに限らず,多くの知見が蛍光タンパク質融合アクチンを用いたライブセルイメージングによって得られているが,もしかするとこれらの知見の多くは,フォルミンファミリーの働きを見過ごしているのかもしれない.では,線維に結合するモチーフをつけたLifeActのようなプローブを用いれば,細胞内線維をきちんと定量イメージングできるのであろうか.
最近我々は,フォルミンファミリーに依存するアクチンダイナミクスを解明する目的で,蛍光色素で化学修飾したアクチンを評価し,フォルミンファミリーの活性についても捕捉可能な新規の単分子イメージング手法を開発した.副産物として,電気穿孔法による100%近い高効率な細胞標識やトランスフェクションの難しい初代培養細胞への応用,高分解能の分子位置捕捉,近赤外蛍光アクチンプローブの開発などが実現した.
本稿の前半では,この手法の優位性を明確に示すため,既存の蛍光イメージング手法のデータ解析で留意すべき問題点をいくつかあげる.特に,2節2)項で述べる細胞辺縁の求心性アクチンフローが引き起こすアクチン結合型プローブのミスローカリゼーションエラーは,発見者の我々にとっても,直感に反する強い影響力には驚かされるものがあった.後半では,単分子アクチンイメージングの改良を通して得られた知見,フォルミンファミリーが関与する二つの物理ストレスに対抗するメカニズムと細胞先導端におけるアクチン重合と外部からの牽引力とのカップリングについて紹介する.
2. アクチンの可視化から学んだライブセルイメージングの落とし穴
1)Tardyモデルが指摘する蛍光退色後回復法(FRAP)の問題点
アクチンは,細胞辺縁で中心へ向かって絶えず流動(レトログレードフロー)する1).この流動はほとんどの培養細胞の自由端にみられるが,一体何に役立っているのであろうか.
最初にこれが可視化されたのは,蛍光退色後回復法(fluorescence recovery after photobleaching:FRAP)を用いてであった2).FRAPは,共焦点レーザー顕微鏡に実装され,気軽に利用できる.構造の動きのみならず,FRAPの回復速度から分子の構造からの解離(入れ替わり)速度が測定されるが,果たして正しくデータが解釈されているのだろうか.
Tardyらは,数理モデルを用い,フォトブリーチングやフォトアクチベーションで印づけられた蛍光分子が印の中に再取り込みされ,見かけ上の回復速度が分子の解離速度より遅くなるケースがあることを指摘した3).考慮すべきは,分子の解離速度だけでなく,拡散速度やどれくらいの割合の分子が拡散状態にあるかが含まれる.これらの性質を知らずして,このアーティファクトは除外できない4, 5).詳細については,原著3)を参照されたい.
葉状仮足では,線維状アクチン(Fアクチン)は,サブユニット濃度で1000 μMもの高密度に達する6).対して,単量体アクチン(Gアクチン)は,100~200 μMほどであり,Gアクチンでいる時間は,Fアクチンの寿命より1桁ほど短いと考えられる.Gアクチンは,コフィリンからの速い解離とプロフィリンによるADP/ATP交換促進により1秒ほどで次の重合の準備が整う.これらをTardyモデルに繰り込むと,葉状仮足ではFRAPの回復が線維の入れ替わりより2倍近く遅くなることが予想されていた7, 8).
この予想が最近実証された.高速で移動する魚類ケラトサイトは,細胞運動のモデルとして頻用される.その葉状仮足で,ケージ化蛍光アクチンのフォトアクチベーションを用いて,Fアクチンの半減期が23秒と短いことが示されてきた9).一方,蛍光単分子アクチンを用い,脱重合を直接観察すると,半減期が12.6秒とさらに半分近く短いことが判明した10).一部は,温度の差など観察系の違いによるかもしれないが,Tardyモデルで予測された2倍近い差が同じケラトサイトを用いて観察されたことは,アクチンのみならず種々の分子のFRAPを実践する上で,再取り込みを吟味する重要性をあらためて強調すべき結果が得られたと我々は考えている.構造に結合している時間の割合が高く,かつ結合と解離を頻回に繰り返している分子では,FRAPを用いて報告されたデータがある場合でも単分子イメージングを用いて再検証すべきである.それによって,正確なダイナミクスが明らかになる場合が少なからずあると予想している.
葉状仮足内では,ケラトサイトで37%10),XTC細胞で34%8, 11)のFアクチンが10秒以下の寿命しか持たない.古くから,葉状仮足ではアクチン線維がトレッドミル様の伸長→脱重合を行うといった仮説があるが,各線維がトレッドミルを行うわけではないことは,これらの速い線維回転から明白である.反矢じり端に結合するキャッピングプロテイン12)やAIP113)のアクチン崩壊に依存した速い解離は,反矢じり端近くでの速いFアクチン崩壊を示唆している7, 14).
2)アクチン流動が引き起こす線維結合型プローブの局在ミス
アクチン動態を観察する際,GFPや蛍光色素をアクチンに直接付加したプローブ以外にも,種々のFアクチン結合モチーフの標識体が用いられる.しばしば,それらの細胞内局在が互いに異なることが報告されるが,その理由は,特定のFアクチン構造との相性の違いによると考えられてきた.この局在の相違にヒントを得て,我々は,構造のダイナミクスに由来するアクチン結合タンパク質やアクチン標識プローブの濃度勾配形成機構を見いだした.
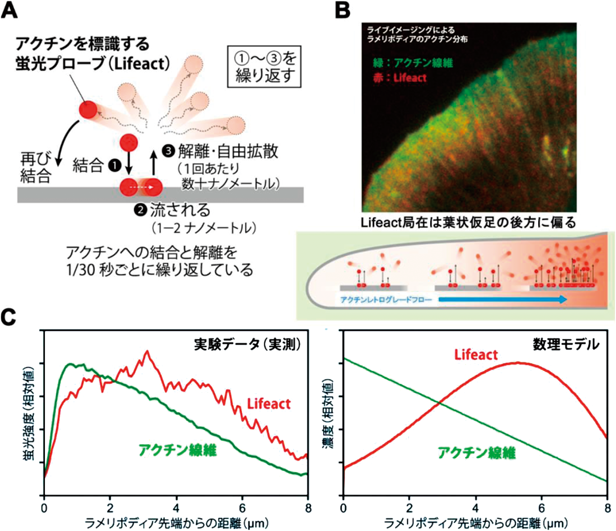
アクチン結合型プローブの代表的なものとして,LifeActがある15).LifeActは酵母のABP140の14アミノ酸からなるアクチン結合モチーフに由来し,細胞内のほとんどのFアクチン構造に結合する.線維からの解離が100ミリ秒以内と短く,アクチン動態への影響がほとんどないとされる.当研究室が開発した結合・解離プローブを用いた分子ローカリゼーション超解像顕微鏡IRISにおいても,固定標本におけるFアクチンの網羅的な超解像可視化に有効に利用されている16).
しかし,ライブセルの葉状仮足においては,蛍光LifeActの分布はFアクチンのそれと一致せず,固定後のファロイジン染色と比べるとLifeActが葉状仮足の後端に偏って分布することに気づいた17).EGFPアクチンでは,このような差異はみられない.興味深いことに,ライブセルでみられたLifeActの仮足後方への偏りは,固定した細胞を蛍光LifeActで染色した際にはみられない.つまり,生きた細胞内特有の現象である.
葉状仮足に内在する高密度のFアクチンは,レトログレードフローによって絶え間なく流動する.このFアクチンによる運搬の影響を,結合・解離を含めた数理モデル(convection-induced gradient distribution model)を構築し解析したところ,LifeActのように結合・解離の非常に速いプローブでも,流動に従ってアクチン結合プローブの分布に後方バイアスがかかることが判明した17, 18).解離のより遅いファロイジンでは,より大きな後方への局在バイアスがかかる.直感に反するのは,LifeActでは,1回の結合あたり1~2 nmしか後方に運搬されず,一方で1回の解離から再結合に至るまでに数10 nmの距離を移動するにもかかわらず,Fアクチンと解離した明白な分布異常が起こることである(図1).
この発見は,標的結合型蛍光プローブの使用において,レトログレードフローや原形質流動のような大きな流動に会合する分子が標的の場合,プローブの分布と標的の実際の分布との間に解離が生じることが普遍的に起きうることを示しており,定量イメージングにおける結合型プローブの使用およびデータ解釈に注意を喚起する.加えて,生物学的にも同様なメカニズムを利用して,Fアクチン結合分子がレトログレードフローへの会合頻度を変化させることで,葉状仮足や糸状仮足で異なった分布勾配を形成している可能性を示唆する.これに類似した考え方はすでに報告されているが,LifeActとFアクチンとの局在の相違に気づいておらず19),イメージングの定量性や数理モデルの構築にあいまいな部分が残されていた可能性が疑われる.また,仮足の先端に局在し機能するFアクチン結合分子にとっては,アクチン流動からの強い影響はやっかいな問題となりうるため,それを回避するための何らかの分子機構を個別に備えている可能性も暗示している.
3)フォルミンファミリーと干渉する蛍光タンパク質融合アクチン
フォルミンファミリーは,真核生物に広く存在するアクチン重合促進分子で,重合するアクチン反矢じり端に結合したまま移動し,連続的に線維を伸長するユニークな能力を持つ20–22).多くのメンバーが細胞質分裂,胚や酵母の極性形成に必須な遺伝子の産物として同定されてきた23).最近の個体レベルの研究では,発生時の神経細胞の移動24)や神経上皮細胞の極性形成25),精子形成26),T細胞分化27)におけるmDia1/3,赤芽球の細胞質分裂におけるmDia228),プルキンエ細胞の樹状突起形成におけるDaam129),心筋サルコメアの形成とリモデリングにおけるFhod330)の重要性がマウスで報告され,ヒトでも難聴を引き起こす新たな変異がhDia131)で報告されている.
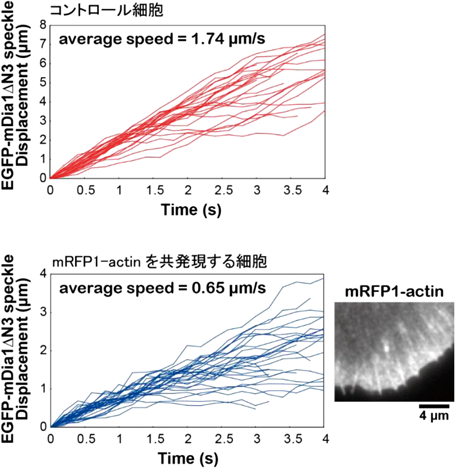
細胞内では,FH1ドメインの多数のポリプロリン配列を介してプロフィリンアクチンを呼び込むことで32, 33),mDia1においては,毎秒700サブユニットを超える速度でFアクチンを伸長させる20).このプロセッシブなアクチン伸長過程に蛍光タンパク質を付加したアクチンが部分的に干渉することが知られている.我々は,mRFP1アクチンを過剰発現する細胞内では,mDia1の分子移動が頻回に停止するようになり,Fアクチンの形態が一見正常にみえる細胞でも,mDia1の速度を停止も含めて計算すると約3分の1まで低下することを報告した11, 34)(図2).また,EGFPアクチンはストレス線維に取り込まれるものの8),よりサイズの大きい蛍光タンパク質を融合したアクチンの過剰発現では,ストレス線維の形態や安定性に異常を来すことも経験している(未発表).分裂酵母に用いられるアクチンGFPは,収縮環に取り込まれない35).これらを合わせると,蛍光タンパク質融合アクチンは,さまざまな程度でフォルミンファミリーの作る線維への取り込みに障害を来し,その可視化には不向きであると考えられる.また,フォルミンファミリーの機能を部分的に抑制するため,FRAP等の実験も含め,蛍光タンパク質融合アクチンを大量に発現した細胞でのアクチンダイナミクスの解析は避けるべきと考える.
我々は,フォルミンファミリーが作る線維のダイナミクスを解明する目的で,DyLight550をリシン残基に低ストイキオメトリーで標識したアクチン(以下,DL-actin)を評価し,通常の線維の80%の効率でmDia1が作る線維に取り込まれることを確認した11).ちなみに,アクチンで頻用されるCys-374の標識体は,その多く(ピレンやビオチンによる標識体を含む)がプロフィリンに対する親和性を失うため,フォルミンファミリーが作るFアクチンへの取り込みが大きく損なわれる33).
このDL-actinは,電気穿孔法を用いると培養細胞にほぼ均一に一分子観察に適した低濃度で導入することができる.EGFPより退色しにくいため,単分子観察の撮影時間設定の自由度が増し,高い輝度により分子の位置測定精度も向上した.この手法をeSiMS(electroporation-based single-molecule speckle)顕微鏡法と命名している11, 36).加えて,自家蛍光の低い近赤外光領域での一分子可視化を可能とするアクチンとして,CF680Rによる標識体も開発した37).1)~3)項で述べた理由により,以前の研究では正確に捉えられてこなかった細胞内アクチンダイナミクスを,eSiMS法を用いることでより正確な定量性をもって検証することができる.細胞運動や組織構築に付随した多くの細胞構造変換の仕組みを,あらためて捉えることに挑むことができるはずである18).
3. 単分子可視化が明らかにしたアクチンダイナミクスがもたらす細胞メカノセンス
1)G/Fアクチンの恒常性が制御する即時型アクチン線維回生
アクチンは真核細胞における最も豊富なタンパク質の一つであり,その細胞内濃度は200~500 μMほどに達する.典型的な培養細胞ではGアクチンとFアクチンはおおむね1:1の割合で存在する.このように豊富な分子が秒単位の細胞応答の引き金として機能するとは,にわかに信じがたいかもしれない.しかし,GアクチンとFアクチンとの変換はかなり動的に起きており,そのバランスの崩れは,アクチン線維再生を急速に促すトリガーとなることが判明している.
この仕組みは,当初Gアクチンの阻害剤であるlatrunculinを低濃度(100 nM前後)で用い細胞を処理すると,mDia1によるアクチン重合核形成の頻度が顕著に上昇することから見つかった38).同じことが,細胞表層を数マイクロメートルほど人為的に変形させた場合でも起こる.後者の反応は,10秒ほどでピークに達し30秒ほどで消失する迅速なものであり,カルシウムイオンを要しない.重要なことは,異なる制御ドメインを持つ複数のフォルミンファミリーが細胞表層の物理ストレスに応答して,アクチン重合を開始することである39).
このことから,フォルミンファミリーによる重合核形成を促進する引き金が反応の基質である遊離Gアクチンの濃度変動であることが予想された7, 38).細胞内におけるGアクチンの高い濃度は,重合核形成を妨げるサイモシンβ4ファミリーやプロフィリンに代表される単量体隔離タンパク質が大量に存在することで実現している40).しかしながら,それらによる隔離作用はGアクチン濃度が高くなるにつれて急速に力を失い,10%程度のGアクチンの総量増加が,隔離タンパク質と結合していない遊離Gアクチンの濃度を2倍以上上昇させるようになる.mDia1は,in vitroで遊離Gアクチン濃度の3乗に比例した頻度で重合核を形成する41).実際に,物理ストレスを負荷した細胞では,コフィリンによる線維切断に応じてFアクチンに会合する42, 43)AIP1が21%上昇し,sFDAPplus法で計測したところ,総Gアクチン濃度が速やかに10~20%上昇することが判明した39).これらは,遊離Gアクチンの濃度サージを十分起こしうる上昇である.さらに,II型ミオシンの阻害薬のアクチン脱重合速度への影響を厳密に検討した我々の最近の研究10, 18)から,ミオシンが生じる張力がFアクチンを安定化していることが判明した.おそらく,細胞表層にかかる物理ストレスが一部の線維の張力を緩め,脱重合を促進し遊離Gアクチンの速やかなサージを引き起こすのであろう.
mDia1は,自己抑制的分子内結合が低分子量Gタンパク質Rhoの結合により解除される分子活性制御を受ける44).しかし,C末端を欠失し自己抑制がはずれた変異体でも,細胞内で連続的アクチン伸長を行う分子は10%ほどであり,細胞膜などに会合した分子の多くが,いわばアイドリング状態というべきランダムな拡散状態にある動きを示す45).このことは,通常の細胞環境下では,mDia1が活性化してもその大半が材料不足のためアクチン重合を開始せず,しばらく待機していることを示唆している.おそらく,何らかの刺激や物理ストレスでFアクチンが壊れると,局所のGアクチンの上昇がトリガーとなり,すでに活性化状態にあるmDia1や他のフォルミンファミリーによるアクチン重合バーストを誘発する二重支配機構が想定される.すなわち,フォルミンファミリーの介在によって動的に保たれるGアクチンとFアクチンとの間の恒常性が存在し,それによって,細胞障害時等に迅速なFアクチンの回復が行われることが,分子可視化によって直接捉えられたのである39, 46).
2)フォルミンタンパク質の回転重合による線維の長距離安定化
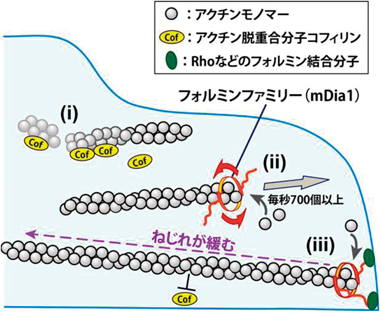
前項で述べたフォルミンファミリーが介在するFアクチン回生機構は,つくられた線維の安定化を伴う可能性が,続く研究によって明らかとなった.当研究室では,Cys-374をテトラメチルローダミンでラベルしたアクチンによる一分子蛍光偏光観察法47)を応用し,mDia1がアクチン線維のらせん構造に沿って回転することを可視化証明したが48),この回転重合が線維のねじれと安定性に影響することが判明した45).
アクチン脱重合因子コフィリンが結合した線維では,約30%ねじれが強くなり49, 50),線維切断が促進される.自由に回転しない状態で固定されたフォルミンファミリーがFアクチンを伸長させた場合,線維のねじれが緩む方向にトルクがかかる.この可能性を電子顕微鏡で検証したところ,10%以内であるがFアクチンのロングピッチらせんのねじれが緩和することがわかった.光学顕微鏡下でのin vitroの実験では,ねじれの緩和がコフィリンによる線維切断に抵抗性をもたらすことが確認された.過剰発現の効果を検証したところ,細胞構造に会合しながらFアクチンを伸長するタイプのmDia1の活性化変異体のみ,細胞内Fアクチンの寿命を延長し,コフィリン分子のFアクチンからの解離速度を速めることが判明した45).このFアクチン寿命への影響は,DL-actinによるeSiMS法を開発したことで初めて検証可能となったことを強調しておきたい.
この機構の発見は,アクチンらせんを変化させる二つ目の分子機構を見いだしただけでなく,コフィリンによる線維ねじれの増強が実際,細胞内でもFアクチン崩壊を促していることを示す証拠を提供している.このフォルミンの作用をHELPPS(HELical Polymerization-mediated Polymer Stabilization)と名づけた46).この線維のねじれを介するアクチン重合因子と脱重合因子のせめぎ合いは,いくつかのユニークな特徴を有している(図3).
まず,フォルミンファミリーが特定のFアクチンのみを安定化することがあげられる.細胞質では,コフィリン,AIP1,コロニンからなるアクチン崩壊機構が強力に働いている43, 51).一方で,mDia1は,アクチンストレス線維とは離れた部位で線維を伸長させる20, 39).また,コフィリンの阻害作用を持つ52)トロポミオシンは,線維に沿って堅く結合するのに相当な時間を要する53).よって,5分程度で誘導されるRhoに依存したストレス線維の形成54)の際に,重合したばかりの線維が壊されずに,十分な確率をもって安定なアクチン線維束に組み込まれるには何らかの仕組みが必要かもしれない.mDia1やフォルミンファミリーは,速いアクチン伸長とともに線維を安定化する機能を発揮することで,ストレス線維や収縮環の形成の際に,効率を上げてFアクチンを供給している可能性が考えられる.
二つ目は,Fアクチンの一端に結合した一つの分子が長距離にわたって線維の性質を変える点である.細胞内での線維安定化やコフィリンの解離促進がはっきり捉えられることから,数マイクロメートル以上にわたって影響を及ぼすと推測される.通常のシグナル伝達では,直接結合して相手の性質を変えるか,リン酸化などの修飾を多数の標的に施し周囲へ影響を及ぼす様式が一般的である.特定のFアクチンに沿って,遠く離れた分子の活性に影響を及ぼすHELPPSのような生化学的作用の伝播様式は,他にあまり例をみない.
三つ目は,線維ねじれが反矢じり端にあるフォルミンファミリーを含む,複数の因子の協調作用によって制御されうる点である.たとえば,細胞内のプロフィリンアクチンの局所濃度が変動すると,重合力の変動からねじれのトルクも変動する.アクチン線維の多くは細胞内でクロスリンクされるが,クロスリンクの頻度やそのオン・オフのダイナミクスによって,ねじれの程度や軸周りのトルクがさまざまに変動することが予想される.関連がありそうな生物の仕組みとして,キラリティー・左右非対称の制御があげられる.巻き貝は,何らかの進化上の理由で左巻き,右巻きが同一種内に存在するまれな生物である.最近,そのキラリティーを決定する遺伝子がDiaphanous近縁のフォルミンファミリーであるLsDia1をコードすることが判明した55, 56).巻き貝のキラリティーは,発生の初期,卵割の4→8細胞期への第3分裂期に割球間でねじれが生じることで決定される.このような表現型を誘導するメカニズムとして,LsDia1が発生するトルクが直接キラリティー誘導に働く,あるいは特殊なねじれを持つアクチン束を生み出すためである,など興味深い可能性がいくつか考えられる.その分子レベルの仕組みの解明には時間を要するであろう.もう一つ関連する知見として,特殊な環境下の培養細胞内に一定の向きで形成される渦状のアクチン細胞骨格があげられる.興味深いことに,Fアクチンのクロスリンカーであるαアクチニンを過剰発現させると渦の向きが逆転する57).上述したフォルミンファミリーとクロスリンカーとの協調ないし拮抗作用が,細胞スケールで形成されるキラリティーの制御につながっている可能性を暗示している.
3)細胞先導端のブラウンラチェットフォースセンサー
葉状仮足では,アクチン重合の力が細胞先端を伸展させる.この力は,細胞膜とアクチン反矢じり端との間に生じる間隙に,確率的にGアクチンが入り込むことで膜を前方に押し出すブラウンラチェットモデルによって,理論的に説明されてきた58, 59).いわば“polymerization motor”である.
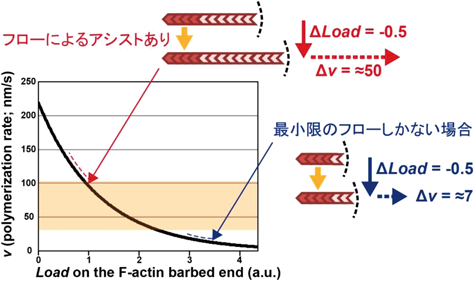
一方,細胞は,外からの牽引力60, 61)や硬度の高い基質の方向62)へ伸展する方向を変えることが知られている.前述したようにブラウンラチェットモデルは,アクチン伸長速度が障壁で押さえられると発生する力が増強することを明示している.これを逆の視点からみると,力が弱まると重合速度が速くなることが予測される.特に,重合速度が速く力が弱い領域では,力の低下がアクチンの伸長速度を大きく加速させる関係がみてとれる.これが,牽引力や硬い基質に向かって細胞が伸展する仕組みである可能性を考え,アクチン重合を直接定量できるeSiMS法を用い,細胞先端を牽引したときの応答を検証した.
この実験において肝要なことは,先導端の張力を変化させる目的で,細胞表面を直接操作しないことである.この操作はフォルミンファミリーの急激な活性化を引き起こしうる39).そこで,我々は柔らかい基質の上に細胞を播種したうえで,マイクロニードルで基質を変形することで,細胞先導端を間接的に牽引する実験系を構築した.結果は,牽引力に即応して,数秒以内に葉状仮足の先端のみでアクチン重合の頻度が上昇することが確認された.この応答には,同部の主要なアクチン重合促進因子と考えられているEna/VASPファミリーやWAVE複合体を必要としない.フォルミンファミリーについても,それらが重合する線維への取り込みが悪いGFPアクチンを用いても応答が確認できるため,関与しないと考えられる.重合促進の程度は,ブラウンラチェットモデルでの予測に一致して,速い牽引力の領域でより急峻な重合の増加がみられた.また,増加したアクチン重合は,20%程度以内の線維密度上昇を伴うが,大半は前方への線維伸長によるもので,細胞先導端が徐々に前方へ伸展する45).これらの結果から,アクチン反矢じり端が直接外力のセンサーとして働き,線維伸長速度を変化させ,細胞を牽引力に向かって伸展させる機構が先導端に存在することが判明した.
このブラウンラチェットフォースセンサーは,もしかすると多くの細胞が薄べったい葉状仮足を形成し,その中にアクチンレトログレードフローを普遍的に形成する理由なのかもしれない.葉状仮足は,その厚さが180 nm以下と薄く,Fアクチンは,その中にサブユニット濃度で1000 μM以上の高密度でパックされている6).1 μm幅あたり300本近いFアクチンが並んでいると推測される.Fアクチンは,互いにクロスリンクされ平行に細胞中心へ向かって絶えず流動する8, 11).アクチン流動を駆動するのは,一部が先導端でのアクチン伸長の反発力で,一部が未知のモータータンパク質の力であり,およそ20~150 nm/sの範囲でほぼ一定速度で駆動されている.これらを合わせると,葉状仮足先端の各々のFアクチン伸長端は,非常に弱い力がかかるようにアクチンフローによって,あらかじめプライミングされた状態にあると考えられる.ブラウンラチェットモデルでは,力の増大により重合速度が指数関数的に減衰するが,この関係においては,弱い力と速い線維伸長が均衡する領域においてこそ,外部からの力の変化に対応して,大きく伸長速度が変化することがわかる(図4).おそらく葉状仮足は,細胞種を超えてこの弱い力と速い伸長とが均衡する状況を作り出すことで,外部の力や硬さに敏感に応答して細胞の伸展方向を舵取りする機能を提供しているのであろう.普遍的かつ最も原始的な細胞力覚センサーして,先導端のFアクチン反矢じり端が機能していると考えられる34).
関連する概念として,ブラウンラチェットのような確率的動作機構は,情報を与えるとエネルギーに変換できることが想定されている63).この変換は,実際にモデル実験を用いて実証されてきた64, 65).つまり,情報とエネルギーは,相互に変換されうるものであり,ラチェット機構はそれらを変換する仕組みとして働くことができる.今回の論文では,牽引された細胞縁に沿って,30秒間に細胞内のおよそ5%相当する量のアクチンの重合が増えると見積っている34).もちろん,一部は速やかに脱重合するので,そのすべてが局所にFアクチンとして蓄積するわけではない.また,アクチンは1分子が重合するごとにATPを一つ分解するので,牽引力から特段のエネルギーが与えられたわけではない.しかし,この量のアクチンの重合が短時間に細胞のある部分で偏在して増加することは,細胞の伸展や移動の方向決定に大きなインパクトを与えるはずであり,実際に牽引した先導端が徐々に突出することも観察されている34).これは,細胞にとって外部の物理力からの情報の獲得を意味している.一見,非効率に思えるアクチン重合によるATPの浪費も,休みなくレトログレードフローを駆動することも,薄く平坦な葉状仮足の形態と高密度のFアクチンを維持することも,外界からの接触や力を感知して的確に行動するために,すなわちブラウンラチェットセンサーを効率よく作動させるために,細胞は維持し続けているのであろう.