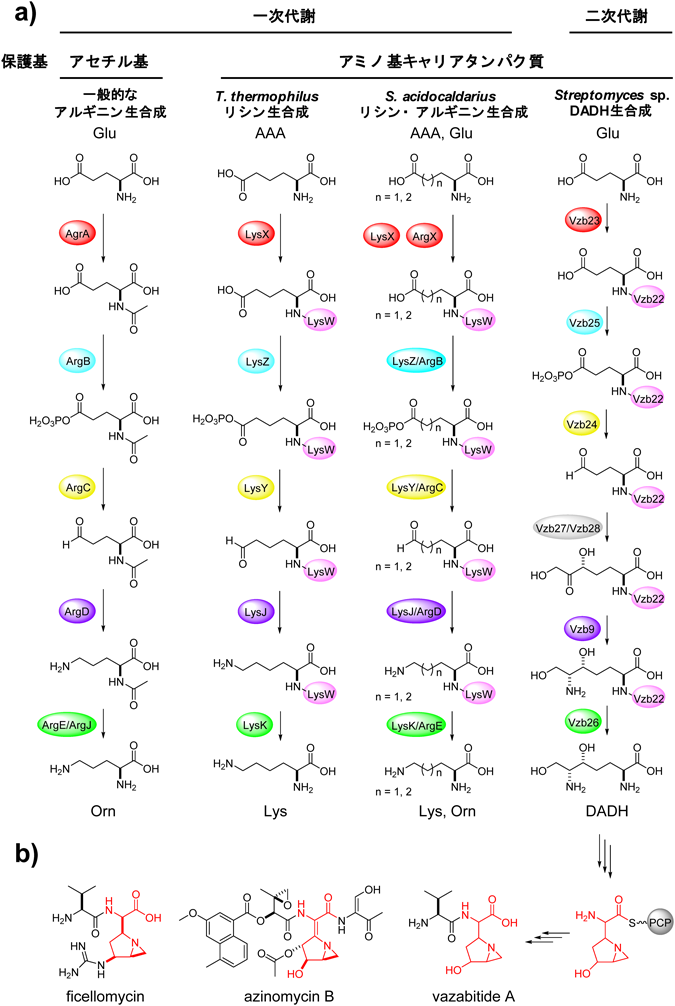
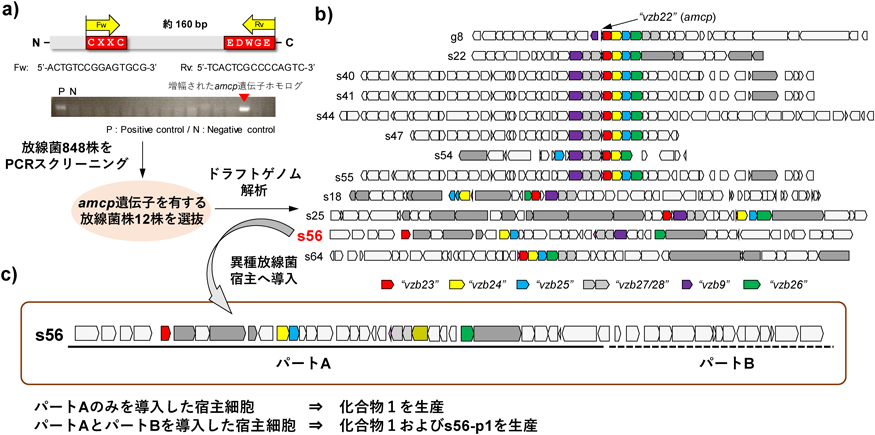
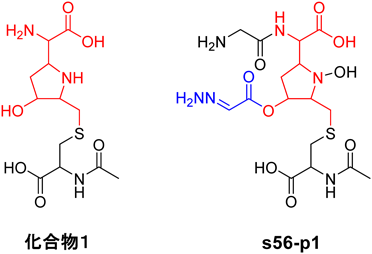
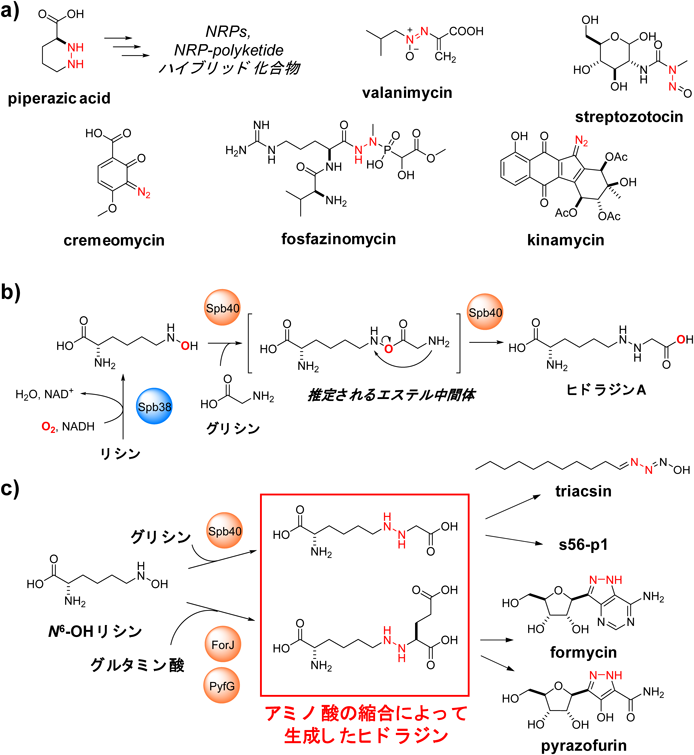
アミノ基キャリアタンパク質を介して生合成される新規天然物の探索と特異なN–N結合形成機構の発見Genome-mining of amino group carrier protein-mediated machinery led to the discovery of an unprecedented hydrazine-forming enzyme in bacteria.
東京大学生物生産工学研究センターBiotechnology Research Center, The University of Tokyo ◇ 〒113–8657 東京都文京区弥生1–1–1 ◇ Yayoi 1–1–1, Bunkyo-ku, Tokyo 113–8657, Japan