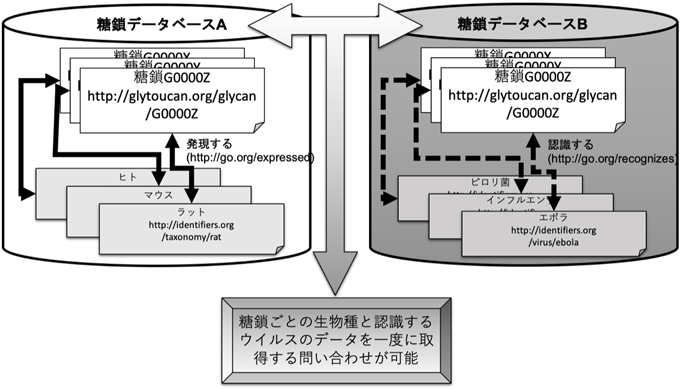
糖鎖インフォマティクスの世界統合World-wide integration of glycome informatics
創価大学・理工学部・糖鎖生命システム融合センターGlycan & Life System Integration Center (GaLSIC), Faculty of Science and Engineering, Soka University ◇ 〒192–8577 東京都八王子市丹木町1–236 ◇ 1–236 Tangi-machi, Hachioji, Tokyo 192–8577, Japan