我々ヒトを含めた真核生物のゲノムには各々のmiRNAをコードする遺伝子が存在する.一方,各々のpiRNAをコードする遺伝子は存在しない.たとえばショウジョウバエのpiRNAの塩基配列をゲノムにマッピングすると,piRNA配列が極端に集中する遺伝子間領域が現れる.この領域は“piRNAクラスター”として知られており1, 2),トランスポゾンの断片が密集することを特徴とする.ショウジョウバエゲノムには150程度のpiRNAクラスターが存在するが,各クラスターの発現は一様ではなく,約15のクラスターから全体の50~70%程度のpiRNAが産生される2).各クラスターの長さはまちまちだが,時には数十万塩基長にわたるものも存在する.
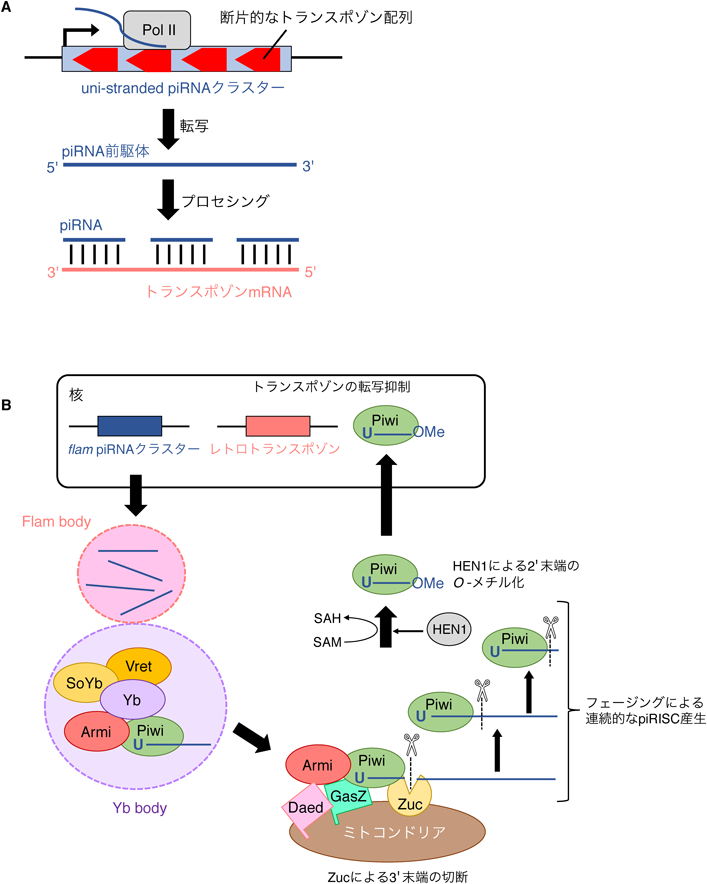
これらpiRNAクラスターはいずれもRNAポリメラーゼII(RNA polymerase II:Pol II)によって転写されるが,ショウジョウバエの場合,それらは片鎖のみ転写されるuni-strandクラスターと双方向に転写されるdual-strandクラスターに大別される(図1A,図2).いずれの場合も,クラスターの転写産物(RNA)はトランスポゾン由来の配列に富むが,その配列は断片的であることから,転移に必要なタンパク質を発現することができず,よって転移活性を有しない.このことから,piRNAクラスターの転写産物は長鎖非コードRNAに分類される.piRNAクラスターの転写産物は,特別な“シス配列”を持つなどの理由からpiRNA前駆体として認識され,その後複雑なプロセシングを経て成熟型piRNAになる(図3).つまり,piRNAはpiRNAクラスターを由来とする小分子RNA群であり,その配列は多様性に富む一方,一つ一つのpiRNAのコピー数(量)はmiRNAのそれに比べ非常に少ないことを特徴とする.
piRNA研究はショウジョウバエ卵巣を用いた遺伝学から始まった.そして今なおショウジョウバエ卵巣が広く用いられる.ショウジョウバエ卵巣は生殖細胞とそれを取り囲む体細胞(濾胞細胞)からなるが,興味深いことにこれら2種類の細胞におけるpiRNA機構はトランスポゾンの発現抑制という目的こそ共有されているものの,その仕組みは一様ではない.以下に,ショウジョウバエ卵巣内体細胞と生殖細胞におけるpiRNA生合成の仕組みを概説する.
1)ショウジョウバエ卵巣内体細胞におけるpiRNA生合成機構
ショウジョウバエ卵巣内の体細胞(濾胞細胞)では主にuni-strand piRNAクラスターが発現する.uni-strandクラスターは固有のプロモーターを持ち,転写は一定方向に起こる.uni-strandクラスターに集積するトランスポゾン断片の大半は転写と逆方向に挿入されており,よって最終産物であるpiRNAの多くはトランスポゾンmRNAに対して相補的なアンチセンス配列を持つ(図1A).
現在,最も解析が進んでいるuni-strandクラスターはflamenco(flam)であり,ショウジョウバエ卵巣内体細胞のpiRNAの大半はflamを由来とする.flamの転写産物は核外へ輸送された後に細胞質顆粒体Flam bodyにいったん集積し,その後,Flam bodyに隣接する細胞質顆粒体Yb bodyへと移行する(図1B).flamは180 kbにもおよび,その転写産物はスプライシングを受けるものの非常に長いと見積もられているが,Flam bodyあるいはYb bodyを経由することによって粗く断片化される2–6).なお,この反応に関わる因子はいまだ不明である.PiwiはYb bodyにおいてこれら断片化されたflam RNAの5′末端に結合し,Piwi-piRISC前駆体(pre-Piwi-piRISC)となる.続いてpre-Piwi-piRISCはミトコンドリアへ移行し,ミトコンドリア外膜上のGasZとDaedalus(Daed)に係留される7, 8).ミトコンドリア外膜に局在するエンドリボヌクレアーゼZucchini(Zuc)は,ミトコンドリア上に移行したpre-Piwi-piRISC中のpiRNA前駆体を,成熟型piRNAの3′末端となる位置で切断することで,30塩基程度の成熟型piRNAの結合したpiRISCを形成する(図1B)9, 10).最終的にpiRNAの3′末端はHEN1/Pimetメチルトランスフェラーゼによってメチル化され,PiwiのpiRNA 3′末端結合ポケットに格納される11, 12).同時に,ZucによるpiRNA前駆体の切断によって,新たにPiwiが結合することが可能な5′末端をもつpiRNA前駆体が形成され,二つ目のPiwi-piRISCが生み出される.このように,piRNA前駆体1分子から連続的にPiwi-piRISCが生成される仕組みをフェージングという13)(図1B).
最近の研究から,Yb bodyは非膜性の顆粒構造体であり,液–液相分離(liquid–liquid phase separation:LLPS)によって形成されることが示された.Yb bodyの形成には,その中核因子であるfs(1)Yb(Yb)どうしの結合のみならず,YbのpiRNA前駆体への結合が必要である14, 15).YbはpiRNA前駆体内のシス配列を認識することによってpiRNA前駆体に選択的に結合する.Yb bodyの他の構成因子としてはVreteno(Vret),Sister of Yb(SoYb),Armitage(Armi)がある16–18).ArmiはATP結合性RNAヘリカーゼで,pre-Piwi-piRISCのYb bodyからミトコンドリアへの移行に寄与する19).なお,piRNA生合成が,非膜性オルガネラ(Yb body)および膜性オルガネラ(ミトコンドリア)で段階的に行われる理由はいまだ理解されていない.
2)ショウジョウバエ卵巣内生殖細胞におけるpiRNA生合成機構
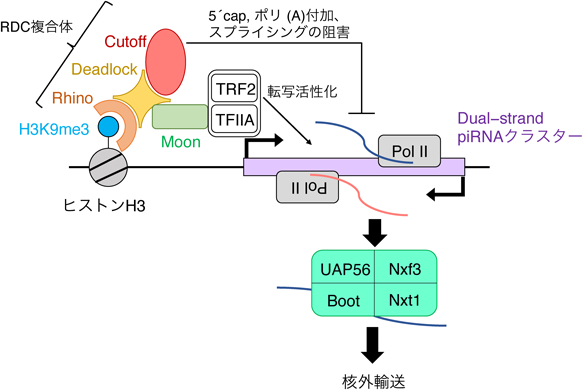
ショウジョウバエ卵巣内の生殖細胞では主にdual-strand piRNAクラスターが用いられる.興味深いことに,dual-strandクラスターは明確なプロモーターを持たず,また,その転写は9番目のリシンがトリメチル化された転写抑制型ヒストンH3(H3K9me3)に依存する20)(図2).通常,H3K9me3にはヘテロクロマチン構成因子HP1が結合するが,dual-strandクラスターではHP1ホモログRhinoがH3K9me3に結合する21).RhinoはH3K9me3上でDeadlockおよびCutoffと結合し,三者複合体RDC(各因子の頭文字をとってRDCと呼ばれる)を形成する.RDC複合体にはさらにMoonshiner(Moon)やTFIIA-S, TRF2が結合することで,dual-strand piRNAクラスターの転写が開始する22).Dual-strandクラスターの転写産物の核外輸送にはBootleggerとRNA核外輸送因子Nxf1, Nxf3, UAP56が関わる23–25).興味深いことに,このRhino依存的なdual-strandクラスターの転写機構はショウジョウバエに特異的である.ゲノム中のレトロトランスポゾンの増加とpiRNA機構の共進化の結果,ショウジョウバエが独自に獲得した機構なのかもしれない.
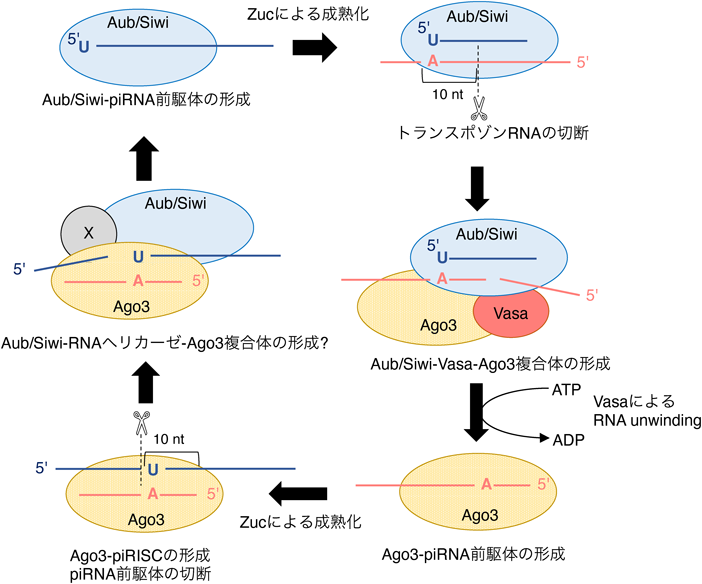
ショウジョウバエ濾胞細胞は三つのPIWIのうちPiwiのみを発現するが,生殖細胞はそのすべて,つまりPiwi, Aubergine(Aub),Ago3を発現する.PiwiとAubはトランスポゾンmRNAに対して相補的な,いわゆるアンチセンスpiRNAとpiRISCを形成する.このアンチセンスpiRNAは卵巣内体細胞におけるpiRNA生合成経路と似た機構で生成されると考えられているが,Ybは生殖細胞では発現しておらず,よってYb/Yb body非依存的に生成される.一方,Ago3に結合するpiRNAはAub-piRISCによって切断されたトランスポゾンmRNAから作られるため,センスpiRNAとなる.つまり,Aub-piRISCとAgo3-piRISCは互いに逆鎖のpiRNAに結合し,互いに逆鎖のトランスポゾン転写産物を切り合う.この仕組みはそのレシプロカルな様相からピンポンサイクルと名づけられた2, 26)(図3).ピンポンサイクルではトランスポゾンの転写産物がpiRNA前駆体となりAub-piRISCとAgo3-piRISCが大量に産生(増幅)される.その際,トランスポゾンの転写産物が消費されるため,ピンポンサイクルはトランスポゾンの転写後抑制機構であるともいえる.
ピンポンサイクル関連因子はこれまで複数同定されているが,その多くは生殖顆粒Nuageに局在する.NuageもYb bodyと同様に非膜性の顆粒構造体で,Nuageの形成阻害はpiRNA生成を阻害するため,Nuageはピンポンサイクルの反応場であると考えられる.Nuageに局在するピンポンサイクル因子の代表例であるVasaは,生殖細胞特異的に発現するDEAD-box型RNAヘリカーゼで,その欠損はNuage形成を阻害する27).カイコ卵巣由来生殖細胞株BmN4を用いた解析から,Vasaは,ピンポンサイクルにおいてSiwi-piRISC(ショウジョウバエAub-piRISCのホモログ)が切断したRNA断片をATP加水分解依存的にSiwi-piRISCから解離し,Ago3に受け渡す機能を持つことが示された28).Vasaがヘリカーゼ活性を失い正常に機能しない場合,Siwiによって切断されたトランスポゾンmRNAはSiwi-piRISCにとどまり,ピンポンサイクルは不活性化される.
最近のヒトVasaホモログDDX4の解析から,そのN末端には天然変性領域があり,DDX4はこの領域を介してLLPSを誘導することが示された29).この反応はDDX4の翻訳後修飾やRNA結合によって左右されるようであるが,その詳細はいまだ不明である.
最近のカイコ卵巣由来生殖細胞株BmN4を用いた解析から,ミトコンドリア外膜に局在するPapiのpiRISC形成への関与も提唱された30).しかし,ショウジョウバエpiRISC形成においてPapiは必須ではなく,Papi欠損時の不妊率も低い31).前述のRhinoもそうであるが,このようにpiRISC形成機構は時として生物種特異性を示すことを特徴とする.その最たるは線虫であるが,そのpiRNA生合成機構はショウジョウバエやマウスの仕組みとの相似性が非常に低い32).
3. Piwi-piRISCによるトランスポゾン転写抑制機構
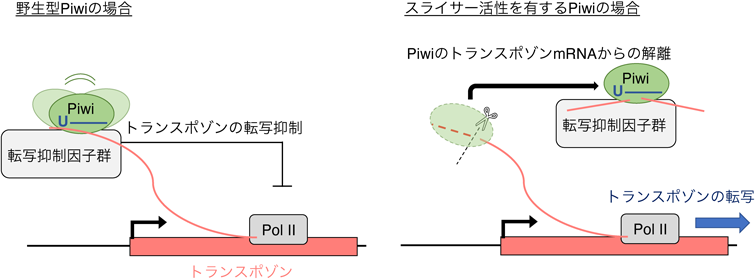
piRISCには,核に移行したのちに転写レベルでトランスポゾンの発現を抑制するものと,細胞質にとどまって転写後レベルでトランスポゾンの発現を抑制するものがある.これは単純にpiRISCを構成するPIWIメンバーに核移行シグナル(nuclear localization signal:NLS)があるかないかによって決定される.ショウジョウバエのPIWIのうち,PiwiはNLSを持つが,AubとAgo3は持たない.AubとAgo3はAGOメンバーであるAgo1とAgo2のようにRNA切断活性(スライサー活性)を持ち,この機能を発揮することによってトランスポゾンを転写後レベルで抑制する(2-2)項参照).この点においては,PIWI(AubとAgo3)とAGO(Ago1とAgo2)は等価であるといえる.一方,Piwiはスライサー活性を持たず,トランスポゾンの発現を転写レベルで抑制する.以下に,核局在型Piwi-piRISCによるトランスポゾンの転写抑制機構に関して概説する.
翻訳されたばかりの,piRNAと結合していないPiwiのNLSは分子内に収納されており,よってpiRNAと結合していないPiwiは核移行することができない.しかし,piRNAの結合後にPiwiのNLSは露出し,これにImportin αが結合することによってPiwi-piRISCは核へと輸送される33).核に移行したPiwi-piRISCは,piRNAに相補的なトランスポゾンmRNAに結合し,複数の補因子とともにその転写抑制を誘発する.最近の研究から,Piwiに変異を導入し人工的にスライサー活性を持たせると,標的RNAから解離しやすくなることが示された6)(図4).自然界においては,トランスポゾンmRNAとpiRNAの相補性は時として不完全な場合があるが,この実験結果はPiwiがスライサー活性を持つ場合,piRISCの標的への結合,ひいては抑制効果が低下することを示唆する.Piwiは十分な転写抑制効果を担保するためにRNA切断能を進化の過程で自ら放棄したのではないかと考えられる.
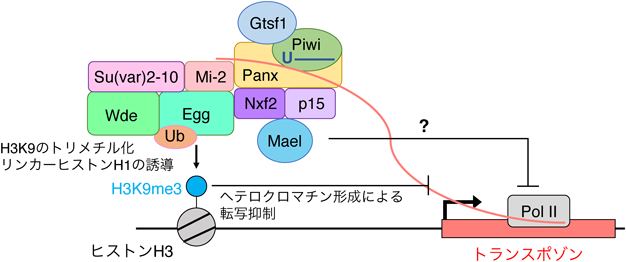
Piwi-piRISCによる転写抑制には補因子が必要である.補因子としてはこれまでPanoramix(Panx),Gtsf1, p15, Nxf2, Maelstrom(Mael),Eggless/Setdb1(Egg),リンカーヒストンH1, Su(var)2-10, Mi-2が同定されている.Panx/Nxf2/p15は複合体を形成し,Piwi-piRISCの標的RNAとの結合を安定化する.また,ヒストンメチルトランスフェラーゼEggはトランスポゾン座位周辺にH3K9me3修飾を導入する役目を果たす34–37)(図5).なお,Eggはモノユビキチン修飾を受け,この修飾がEggのメチルトランスフェラーゼ活性に大きな影響を与えることが示されている38).また,Eggの補因子Windei(Wde)はEggを介してヘテロクロマチン形成に大きく寄与する39).リンカーヒストンH1はクロマチン構造をより強固にする役割を果たす40).Su(var)2-10やMi-2は転写抑制型ヒストン修飾を誘導し,ヘテロクロマチン形成に寄与する41, 42).一方,Maelはトランスポゾンの転写抑制に必要であるにも関わらず,H3K9me3修飾には関与しないことが示されており,その機能的寄与はいまだ不明である.Gtsf1もPiwi-piRISC依存的なトランスポゾンの発現抑制に必須であるが43),その詳細な分子機能は不明である.
Piwi-piRISCが補因子とともに転写中のトランスポゾンmRNAに結合したとしても,Pol IIの活性が直ちに阻害されなければ転写は続行し,トランスポゾンmRNAがPiwi-piRISCに結合したままゲノムから解離してしまうため,トランスポゾンの転写抑制は起こらない.よって,Pol IIの活性を“瞬時に凍結する”因子の存在が示唆されるが,そのような因子はいまだ同定されていない.Piwi機能の欠損は速やかにトランスポゾンの脱抑制を引き起こす.これはPiwi-piRISCによる転写抑制は恒常的ヘテロクロマチン形成には至っていないことを物語る.
4. Piwi-piRISCとSiwi-piRISCの立体構造
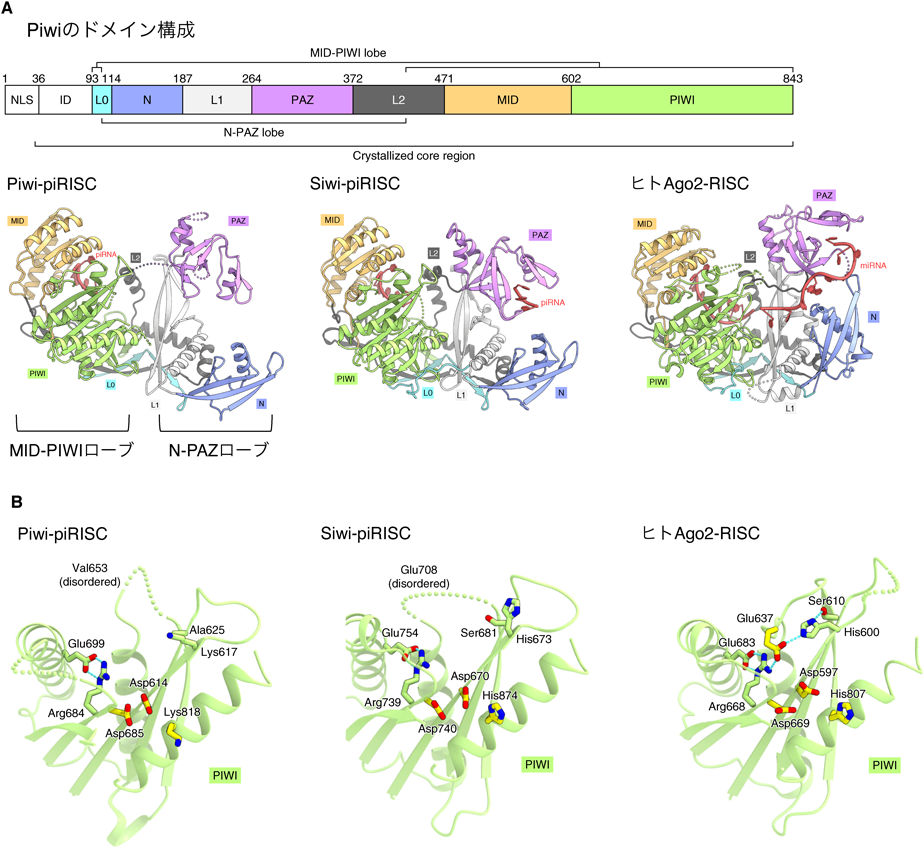
最近,X線結晶構造解析によってPiwi-piRISCおよびSiwi-piRISCの立体構造が明らかとなった6, 44).PIWIはAGOと同様のドメイン構成を有するが,各ドメイン(Nドメイン,PAZドメイン,MIDドメイン,PIWIドメイン)の構造自体は類似していることが判明した(図6A).Siwi-piRISCのMIDドメインはpiRNAの5´末端のウリジンおよびリン酸基を特異的に認識し,PAZドメインはpiRNAの3´末端のメチル基を認識していた.一方,piRNAの中間領域である約20塩基の構造は不明瞭であったことから,piRNAの中間領域は柔軟性を持つことが示唆された.既存のAGO-RISCの構造と比較すると,N-PAZドメインとMID-PIWIドメインの配向が異なっており,この配向の差がAGO-RISCとPIWI-piRISCの生合成機構の違いを反映すると考えられた.PiwiのPAZドメインはpiRNA 3´末端を認識するものの,その立体構造は不明瞭であった.PiwiのPAZドメインはより柔軟な構造をとることによってpiRNAの長さの多様性に対応できるようになっているのかもしれない.
AGO-RISCのPIWIドメインはRNase H構造をとり,その活性中心はAsp-Glu-Asp-Asp/Hisからなる45, 46).スライサー活性を示すSiwiにはこの4残基が保存されているが(図6B),PiwiではAsp-Val-Asp-Lysに変異している.これを人工的にAsp-Glu-Asp-Hisへと変異させるとPiwiはスライサー活性を示すようになる6).ただし,先に述べたように,このPiwi変異体は標的RNAからはがれやすいため,進化の過程でPiwiはスライサー活性を捨てたのだと考えられる.
Piwi-piRISCによるトランスポゾンの転写抑制機構の分子レベルでの解明には,Panoramix(Panx),Gtsf1, p15, Nxf2, Maelstrom(Mael)など補因子との高次複合体の立体構造解析が必須であるが,それはまだ解かれていない.今後の研究の発展が待たれるところである.