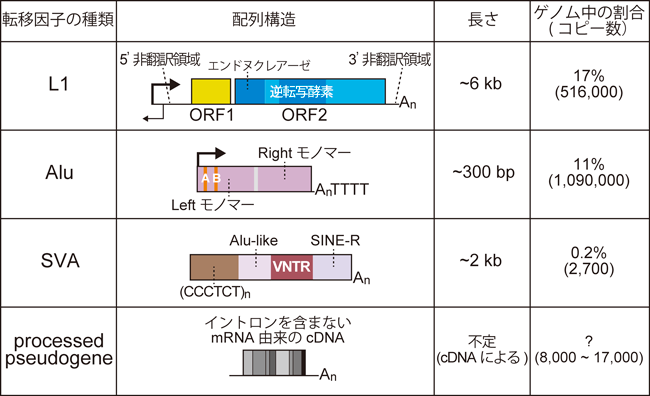
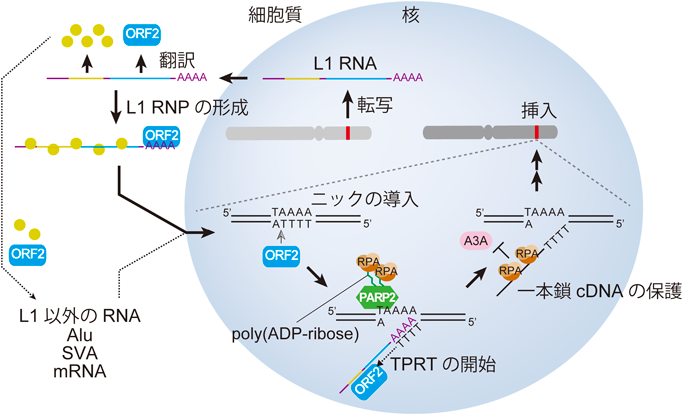
ヒトレトロトランスポゾンと宿主因子との間で繰り広げられる攻防と連携A battleground between human retrotransposons and host factors
京都大学大学院生命科学研究科Kyoto University, Graduate School of Biostudies ◇ 〒606–8501 京都府京都市左京区吉田近衛町 医学・生命科学総合研究棟 ◇ Yoshida-Konoe-cho, Kyoto-shi, Kyoto 606–8501, Japan