1)細菌とアーキア
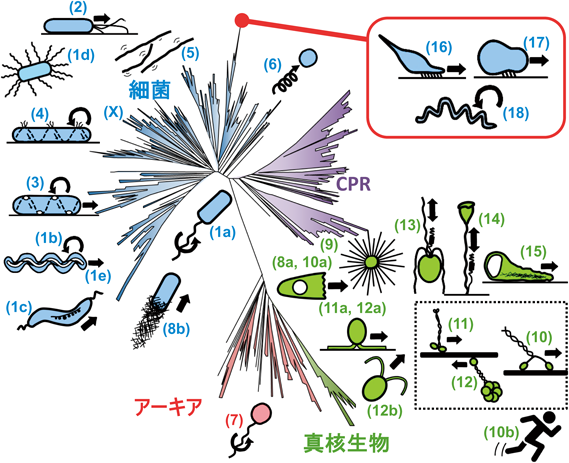
三つの運動能を持つことはモリクテス綱の大きな特徴の一つだが,それでは全生命において運動能はいくつ存在しているのだろうか? 著者らは新学術領域研究「運動超分子マシナリーが織りなす調和と多様性(略称:運動マシナリー)」で個別の研究と俯瞰のための議論を行い,これまでに報告されている細胞運動が18種類に分類できることを明らかにした(図1)5, 7).ここでの分類は力発生ユニットの構造を基にしている.たとえば,ヒトの体にある骨格筋も心筋も平滑筋も同じメカニズムで動いているが,精子の遊泳や咽頭や輸卵管の繊毛運動は異なるメカニズム,といった具合である.さらに細菌,アーキア,真核生物の三つのドメインそれぞれに運動能進化のストーリーがあり,細胞フォーマットとともに全体としてつながっていることが浮き彫りになった.
細菌では大半の種がべん毛か線毛で動いている.べん毛は細胞の表面に生えた,らせんの繊維構造で,細胞膜に埋め込まれた基部体と呼ばれるモーターにつながっている[図1:(1)].プロトンなどのイオンが膜の外側から内側へ向けモーターを通って流れることでモーターが回転し,べん毛が回転する.べん毛はらせん状なので,水を後ろに押しやって細菌が遊泳する.べん毛運動にはべん毛を自身の細胞に巻きつけて自身が変形しながら動くものや,多数のべん毛を動かして水のほとんどない固形物表面を動くものなど,多くのバリエーションがある.線毛はやはり基部体から細胞外へのびた繊維構造で,こちらはATPのエネルギーとAAA+ ATPaseに依存して伸長と短縮を行う[図1:(2)].すなわち伸長した線毛が固形物にはりついて基部体が張力を感じると,線毛が細胞側に引き込まれるために細胞が移動するのである.べん毛や線毛の構成タンパク質構造は膜に存在する輸送装置と相同性があるため,べん毛や線毛と輸送装置が共通の祖先を持つ可能性が高い.細菌の運動能には,モリクテス綱以外にもべん毛でも線毛でもないものが5種類知られている[図1:(3)~(6),(8b)].この5種類はべん毛や線毛のような大きな付属物が含まれておらず(8b)以外には門を超えた分布は見つかっていない.付属物を振り回すことが困難な,入り組んだ環境を進むための解決策として進化したものと考えられる.べん毛と線毛を含むほとんどの運動能がペプチドグリカン層を必要とするが,運動の装置は細胞表面に存在し,機能するには力を受け止める足場が必要であるため,このことはしごく当然なのかもしれない.
アーキアの運動能についてはこれまで,アーキアべん毛(archealla)と呼ばれるもの1種類しか見つかっていない[図1:(7)].アーキアべん毛は名前のとおり,らせん状の繊維が基部体につながったもので,やはりスクリューのように回転して水を押して細胞を前に進める.見た目は細菌のべん毛と似ているが,実際には基部体も含めて細菌の線毛基部体と相同性がある.一般的なアーキアは,環境や細胞の大きさなどで細菌と共通点も多い.なぜ数多くある運動能の中で,線毛のみがアーキアべん毛に変化して使われているのかは興味のあるところである.アーキアにはペプチドグリカン層がないために,細菌の運動能がアーキアでは働きにくいことが大きな原因と考えられる.また,細菌の運動器官の中で線毛がペプチドグリカン層への依存度が少ないために,アーキアで機能しやすかったのかもしれない.あるいはアーキアの細胞の構造や挙動が研究対象となってまだ日が浅いせいで,アーキアべん毛以外の運動能がまだ見つかっていないだけかもしれない8).
2)真核生物
真核生物の直近の祖先と考えられているAsgardアーキアは,ペプチドグリカン合成酵素やDNAを輸送するために細菌が作った細胞骨格タンパク質,MreBやParMなどからアクチンを進化させることで,細胞骨格重合・脱重合による膜構造を形成する能力(膜リモデリング能)を獲得して細胞巨大化への道をたどった3, 9).そして,巨大化した真核生物は細菌やアーキアのようにスクリューを回転して泳ぐことはもはやできなくなった.その代わりに獲得したのは,膜リモデリング能を活用してアメーバや太陽虫のように仮足ではうことである[図1:(8a),(9)].一般的に細菌は小さく,多くの物質は拡散のみで細胞の隅々まで届くため,細胞内で積極的に物質を輸送する必要はなかったが,細胞が巨大化すると拡散のみでは不十分になった.そのため,細菌がペプチドグリカン合成酵素を運ぶために作ったFtsZなどからチューブリンを進化させ,アクチンとともに重合・脱重合を行い細胞内輸送を行うようになった.アクチンとチューブリンという細胞骨格を用いた細胞内物質輸送をさらに効率化するために,真核生物はミオシン,キネシン,ダイニンなど細胞骨格の上で力を発生する“モータータンパク質”を進化させた[図1:(10)~(12)].モータータンパク質の動きが細胞表面に伝わって新しい運動能となり,また細胞骨格間で働いた場合には収縮力となりアメーバ運動やさらには筋肉の運動へと進化していった[図1:(10ab)(11a)(12ab)].ここで,真核生物が必ずしも細胞壁を持っていないことと,細胞の複雑化のためにハウスキーピングタンパク質の種類が増えたことも有利に働いている.このことは真核生物が既存の細胞骨格に依存しない三つもの運動能を獲得していることからもみてとれる[図1:(13)~(15)].
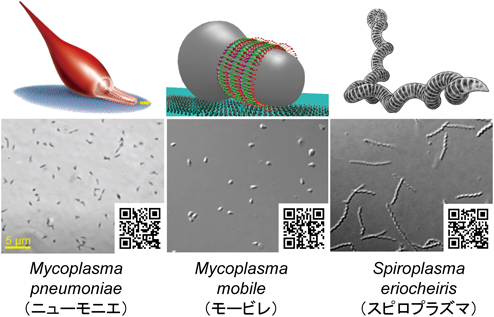
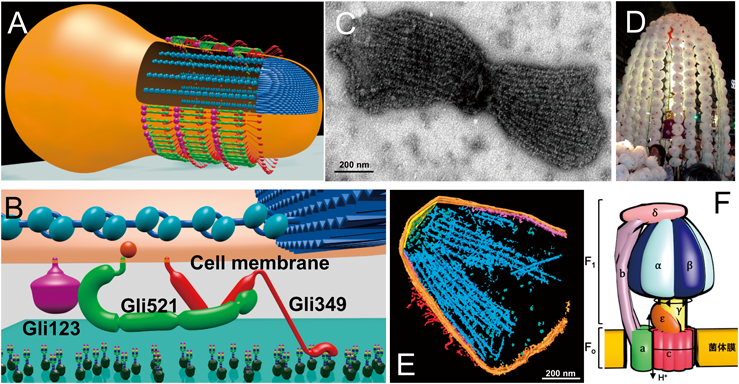
以上から,真核生物とモリクテス綱のみが,細胞の基本機能の多様性の観点からみてごく小さなグループであるにもかかわらず,多くの運動能を確立していることがわかる5–7).これまでに見つかっているモリクテス綱のほとんどは動植物に寄生するため,モリクテス綱の始まりはカンブリア大爆発以降,現在から5億年以内である可能性がある.真核生物が誕生したのが23億年前とすると,モリクテス綱が三つもの運動能を獲得していることは特徴といってよい.以下に著者らが1997年ごろから研究してきた三つの運動メカニズムを紹介する.すなわち,ヒト肺炎菌Mycoplasma pneumoniae(マイコプラズマ・ニューモニエ,以下ニューモニエと表記)に代表される滑走運動,淡水魚病原菌Mycoplasma mobile(マイコプラズマ・モービレ,以下モービレと表記)に代表される滑走運動,上海ガニの病原菌であるSpiroplasma eriocheiris(スピロプラズマ・エリオケイリス,以下スピロプラズマと表記)に代表される遊泳運動の三つである(図2).
1)ニューモニエタイプ滑走運動のメカニズム
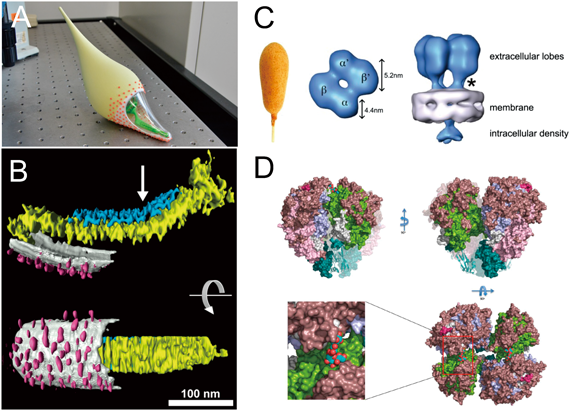
ニューモニエは長さ1~3マイクロメートルで,細胞前側に細胞膜が少し突出した接着器官(attachement organelle)を形成し,動物細胞やガラスなど固形物表面に結合し前方に向かって滑走する(図2)6, 10).速度は毎秒1マイクロメートルくらいと特段に速いわけではないが,細胞が小さいのでビデオを見ればリアルタイムでも動いていることが容易にわかる.結合対象はインフルエンザウイルスと同じシアル酸オリゴ糖で,その結合はシアル酸オリゴ糖の構造に依存する11).受容体複合体は低解像度の電子顕微鏡像からNap(短柔毛の意味)と呼ばれていたが,2020年に粒子状の高分解能構造が発表された12–16).アメリカンドッグのような形状の2種類のタンパク質が膜に刺さるように並んだヘテロ二量体を形成し,さらにこのヘテロ二量体が二つ集合したヘテロ四量体であり,軸方向からみると二回転対称体である.著者らはNapとシアル酸オリゴ糖の結合解離が細胞軸に対して方向性を持つことを期待していたため,回転対称体であることは意外であった.シアル酸オリゴ糖をつかむのは,約170 kDaの受容体,P1 adhesinと長年信じられてきたが17–20),実際には結合部位は同じオペロンにコードされている別のタンパク質に存在していた(図3)21).ただ,モノクローナル抗体の特異的な阻害効果からP1 adhesinが結合や滑走運動に関与していることは間違いない22).構造でみると大きい方のアメリカンドッグがP1 adhesinで,小さい方の先端にシアル酸オリゴ糖結合サイトがあり,複合体全体の構造変化によりP1 adhesinが,結合サイトを覆ってしまうのである.接着器官内部は電子顕微鏡切片像などで明るくみえるため,translucent areaと呼ばれる6, 23, 24).これは力学的な特徴を付与するために細胞質の他の部分と物性の異なる物質で満たされていて,電子の透過性が異なるものと思われる.細胞を界面活性剤で処理するとすぐに滑走運動は止まってしまうが,そのときにATPを同時に加えると運動が維持されることから,運動の直接のエネルギー源はATPであると考えられる25).translucent area中央にコアと呼ばれる構造が縦方向に配置されている.コアは約300ナノメートルの長さで,前方からterminal button, paired plates, bowlの三つに分けられる24, 26).terminal buttonは膜への固定,paired platesは力の伝達,bowlは力の発生を行っている.接着器官を構成するタンパク質はこれまでに約15種類が報告されているが,蛍光タンパク質との融合実験で,さらに多くの構成タンパク質の存在が示唆されている24, 27–30).paired platesはcoiled coilタンパク質が縦方向に並んでできたthick plateと,それを支えるハチの巣構造を特徴とするthin plateを持ち,paired platesが欠損した株では滑走速度が極端に遅くなる31).ニューモニエタイプ滑走運動は,モリクテス綱の三つの運動能の中で最も多くのグループにより研究がなされてきたが,メカニズムについていまだにこれといった答えは得られていない.この運動は他の2種類に比べて観察が難しいため,実験の自由度が低いことも一因となっている.現時点で考えられるメカニズムは以下である(図4).すなわち,bowlで発生された動きがpaired platesとtranslucent areaの伸び縮みとして接着器官全体に伝わる.与えられた動きによってNapはシアル酸オリゴ糖をつかんだり離したりするが,接着器官内部構造がシアル酸結合に方向性を与えることで,ランダムな結合解離から,前方向への動きを生み出す.
2)モービレタイプ滑走運動のメカニズム
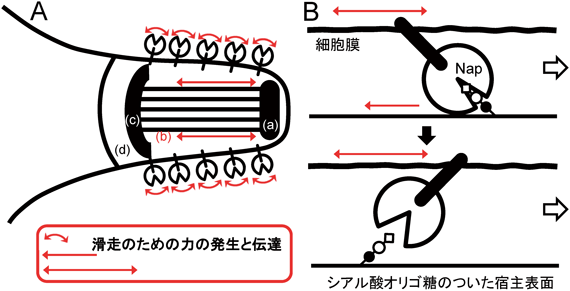
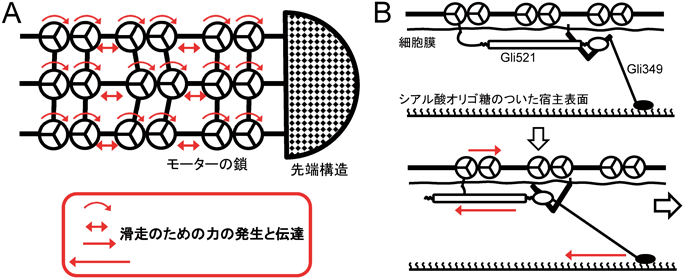
モービレは長さ1マイクロメートル弱で,だるまやボーリングのピンのような形状をしている(図2).細く突き出た側が滑走装置であり,装置の方向に向かって動物細胞や組織の表面などを滑走する32, 33).速度は毎秒4マイクロメートルくらいに達し,スムースにしかも高速に全細胞が動き回る様は圧巻である34–38).結合対象はシアル酸オリゴ糖で,鍵と鍵穴の関係で認識する11, 39, 40).これらのことから,著者はいつか前項のニューモニエタイプとモービレタイプ2種類の運動能が同じメカニズムに帰納する日が来ると何となく期待していたが実際は逆であった.あしとして働くタンパク質は3183アミノ酸からなる349 kDa,長さ約95ナノメートルの八分音符(♪)形状のタンパク質Gli349で41–45),それとは別に表面で力伝達を行う4727アミノ酸からなるクエッションマーク形状(?)のGli521と46–50),滑走装置の形成に関わる1111アミノ酸からなるGli123が滑走装置表面に細胞あたり約450分子ずつ存在している(図5)51).すなわち細胞周囲にGli349の一部で構成される約50ナノメートルの“あし”が約450本生えている52, 53).Gli349はN末端に膜貫通セグメントを持ち,C末端側にToll様受容体などで知られるleucine-rich repeatを持つ受容体ドメインを有している41, 54).分子形状などから,Gli349が抗原性の変化を担うタンパク質と共通の祖先を持つ可能性がある55–57).表面タンパク質分子の大きさと数,局在している細胞表面積から考えると,これらの分子は細胞表面で規則正しく並んでいると思われる.装置内部には驚いたことにATP合成酵素から進化した六量体のモーターがセットとなり二量体を形成し,それが約17個連なった鎖となっている58–61).この鎖は細胞あたり約28本存在し,お会式の万灯のように先端で束ねられており,さらに鎖が横方向に結合して巨大なグリッドを形成している.もっと驚いたことに鎖やグリッドのつなぎ部分には解糖でATPを合成する酵素として知られる,ホスホグリセリン酸キナーゼが含まれている(未発表).モリクテス綱細菌は呼吸を行わないが,Phytoplasma属やUreaplasama属などを除いてはATP合成酵素を持っている.これらはATPのエネルギーを使って膜電位を形成するために使われていると思われる.モービレのゲノムにもそのATP合成酵素の遺伝子一式が存在する.滑走装置内部構造の構成タンパク質をみると,触媒部分を形成するαとβサブユニットはATP合成酵素と30~40%のアミノ酸相同性があるが,それ以外のタンパク質のアミノ酸配列はあまり保存されていない32, 58, 62).鎖は細胞膜を内側から裏打ちするように配置され,2種類の架橋が膜側へ伸びている.これらの構造で細胞膜外側の巨大タンパク質と接続しているのであろう59).触媒部分の膜に対しての方向はATP合成酵素と共通である.細胞を界面活性剤などで透過化すると細胞は瞬時にガラス上にはりついたまま止まってしまうが,そこにATPを加えると再び元の速さで動くようになる(滑走ゴースト)ことなどから,直接のエネルギー源はATPであると結論づけられた63–66).また,ATP加水分解反応の各ステージとシアル酸オリゴ糖の結合解離にはカップリングが存在する34, 59, 67, 68).内部構造を原子レベルで明らかにしたところ,ATP合成酵素由来の六量体モーターの中央に軸として存在する,ATP合成酵素のγサブユニットに対応する部分が回転している可能性が示唆された(未発表).また,異なったATPアナログを加えて構造を解析すると,鎖の上でモーターの二量体間の距離が変化していた59).モービレの滑走運動はきわめてスムースだが,シアル酸オリゴ糖を加えることでそれぞれの動きを知ることができる.あしの1回のステップは約70ナノメートルと真核生物のモータータンパク質などと比べて非常に大きく64),逆に発生している力は数分の1であった46).このことは,モーターの動きが細胞表面の巨大なタンパク質を伝わる間に増幅されていることを示唆している.
力発生からシアル酸オリゴ糖を引っぱる動きに至るメカニズムについては,現在以下のような作業仮説を持っている(図6).すなわち細胞内部に存在するATP合成酵素から進化した滑走モーターが,ATPのエネルギーを使って軸を回転させる.軸の回転がモーターの鎖の伸び縮み,すなわちモーター位置の軸方向の動きを起こす.軸方向の動きは細胞表面に存在するクランク構造により増幅され,70ナノメートル幅のあしの動きとなり,それぞれのあしが宿主のシアル酸オリゴ糖をつかんだり引っぱったり離したりして細胞を前方に引っぱる.
3)スピロプラズマ遊泳のメカニズム
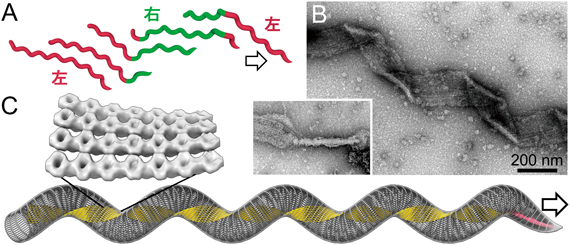
節足動物と植物を往来する病原菌として知られるスピロプラズマは,らせん状の細胞構造をとり,そのらせんの向きを反転することで高粘度の液中を遊泳する(図2,図7)69, 70).これは細胞外膜の内側,ペリプラズム空間に細胞軸に沿うようにらせん状のべん毛を走らせ,自身をべん毛のようにして動くスピロヘータとはまったく異なる動きである5).らせん反転は細胞の先端部から起こり,後端部へと移動する.前方端を固定して考えると,らせん反転するためには後端部に角度変化が蓄積するため,これを解消するために水を後ろに押す方向に回転することとなる.
著者らはよくモリクテス綱の運動能の目的について質問される.上述のモリクテス綱の二つの滑走運動については走性が存在しないが,新天地を広げるだけで生き残りにはかなり有利になると考えられる.ところが,スピロプラズマの遊泳能にはアミノ酸や糖に対する明瞭な走性がみられる.さらに興味深いことに,スピロプラズマゲノム中には細菌の他の化学走性すべてに関わっている二成分制御系が見つからない.これは,スピロプラズマの化学走性がまったく別の方法で行われているか,アミノ酸配列が大きく変化した二成分制御系(?)が働いていることを意味している71).
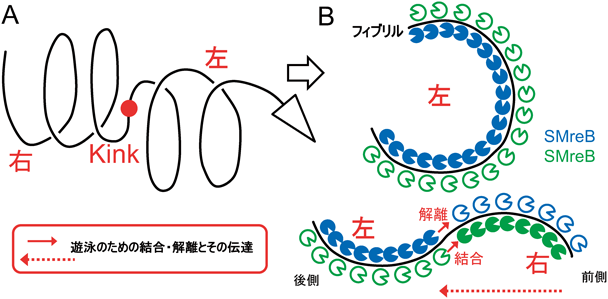
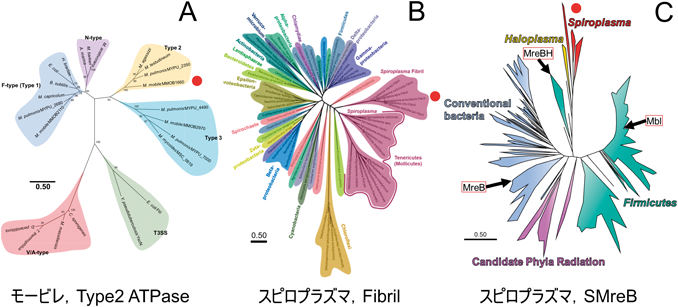
スピロプラズマの特徴的ならせん形状は,細部内部を走るリボン構造によりもたらされている(図7)71–73).さらにリボン構造は,“フィブリル(fibril,微小繊維の意)”と命名されたタンパク質からなる,繊維(プロトフィラメント)数本が横方向に結合して構成されている.プロトフィラメントはらせんを持ち,そのらせんがそのままリボンのらせんに反映される(未発表).リボンの細胞先端側はダンベルと名づけた構造に束ねられている71).フィブリルタンパク質のN末端側ドメインは,メチオニン代謝などに必須でS-アデノシル-L-ホモシステイン(SAH)を加水分解する酵素74),アデノシルホモシステインヌクレオシダーゼとアミノ酸配列の相同性がある72).このタンパク質がタンデムに融合してフィブリルへ進化したと考えられる.フィブリル内部にはラチェットのような機能が存在するらしく,右向きと左向きの二つの決まったねじれ角で固定される(未発表).リボンには真核生物のアクチンのホモログで,多くの細菌に保存されているMreBが含まれる.一般的にMreBは桿状の細菌細胞中でATPのエネルギーを用いて重合し短いフィラメントを形成する.この繊維が細胞膜の曲率を検出することで,ペプチドグリカン合成酵素を細胞の適切な位置に配置し,正常な円柱形状を維持する役割を果たしている75).スピロプラズマのMreB(SMreB)は,他のMreBから系統上離れた位置に存在しており,たとえば,モリクテス綱以外では最も近縁であるフィルミクテス門の桿状細菌,枯草菌のMreBとの間でもアミノ酸の相同性が約40%しかない76, 77).興味深いことにスピロプラズマのゲノムには5クラスものSMreBが存在する.進化の過程で遊泳能を獲得した後にさらにチューンアップが行われたものと思われる.これまでの遺伝解析の結果は,5クラス存在するSMreBのそれぞれが異なった役割を果たしていることを示している(未発表)78, 79).また,SMreBにはATPに依存する重合能が観察される78).
スピロプラズマの遊泳運動について,現在著者らが考えている単純化したメカニズムは以下である(図8).すなわち,フィブリルから構成されるリボンのコア部分は,右向きと左向きの二つの決まったねじれ角を持つ.リボンのコア部分を異なったSMreBが反対方向へ引っぱることでらせんを反転させる.SMreBが起こす反応はそれぞれのサブユニットが固定された状態で起こす重合と脱重合といったもので,らせん反転部位に対応する.スピロプラズマ遊泳運動におけるらせんの反転が細胞軸に沿って移動する速さは,毎秒10~15マイクロメートルであり,真核細胞中でみられる調節タンパク質によって加速されたアクチン重合速度よりも1桁速い80, 81).しかしSMreBがすぐにSMreB線維の端に結合できる位置に固定されていれば,結合は濃度に依存せず常に速くなるため,遊泳に必要な速度が達成できると期待される.
このようにして俯瞰すると,モリクテス綱細菌と真核生物の運動能の発生にはいくつかの共通点がある.(i)細胞の進化によりそれまで使っていた運動能が使えなくなった.(ii)細胞のハウスキーピングな活性で動きのあるものが基となった.(iii)細胞膜周辺に硬い構造がないので,細胞内部の動きが外側に伝わった.逆にモリクテス綱以外の細菌の場合については,ここではあまり解説できなかったが,以下が共通項になっている.(i)トランスポーター(膜輸送装置)を基とする.(ii)べん毛と線毛の影響が大きい.(iii)ペプチドグリカン層を足場として使う.
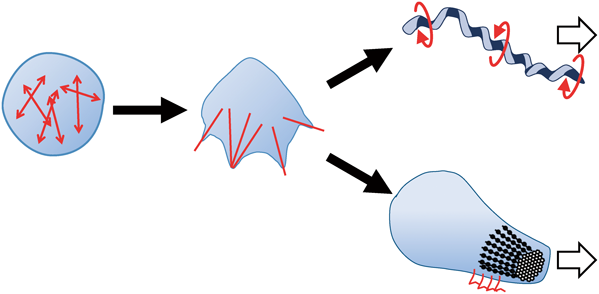
運動能の発生と進化の観点で見たとき,モリクテス綱のモービレタイプ滑走運動とスピロプラズマの遊泳運動は特に興味深い.なぜなら,元は動きに関係なかったハウスキーピングタンパク質,すなわちATP合成酵素,アデノシルホモシステインヌクレオシダーゼ,MreBが運動能のためのモーターに進化した過程が現存するゲノム情報を基にたどれるようにみえるからである(図9).これが唯一モリクテス綱でのみ可能で他の生物では困難にみえるのは,モリクテス綱が若い細菌のグループであるためかもしれない.そしてこの過程を実験室で再現することができれば,我々は運動能とはなにか,さらには生命とはなにかについて,少しだけ理解を深めることになるように思う(図10).なぜなら,突き詰めて考えれば,生命は制御可能なエネルギーである電子を中心に展開する,“エネルギーの流れ”と考えられるからだ.