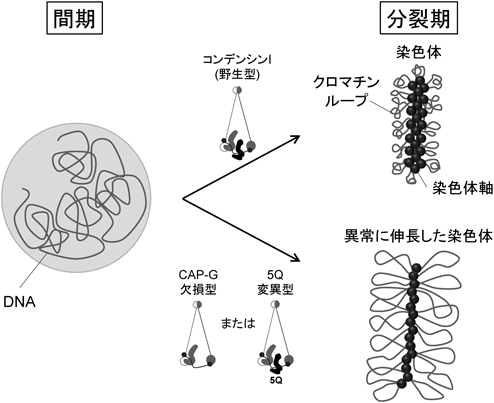
コンデンシンIにおけるHEAT-kleisin相互作用の構造生物学的研究Structural study of the interaction between HEAT repeats and kleisin subunit of human condensin I
静岡県立大学薬学部Department of Physical Biochemistry, School of Pharmaceutical Sciences, University of Shizuoka ◇ 〒422–8526 静岡県静岡市駿河区谷田52–1 ◇ 52–1 Yada, Suruga-ku, Shizuoka 422–8526, Japan