非標準アミノ酸(non-canonical amino acid:ncAA)をタンパク質の部位特異的に導入する技術は,生命科学や医療の研究などにきわめて有用である1–5).従来にない性質をタンパク質に導入できるようになり,タンパク質の機能解明や,新しい機能を持つタンパク質の創出など,さまざまな応用可能性を持つ技術である.近年,200種類以上の非標準アミノ酸を特定のコドン(主に停止コドンUAG)に導入した例が報告されている.化学反応性の高い非標準アミノ酸は生体内で特異的に化学反応を進めることが可能であり抗体薬物複合体(antibody-drug conjugate:ADC)や検出プローブ等への応用が報告されている.また,タンパク質の翻訳後修飾は,タンパク質の機能や活性を調節して細胞内のシグナル伝達ネットワークの制御などに関わるため重要な研究課題の一つである.そのような研究において,あらかじめ修飾されたアミノ酸を非標準アミノ酸として直接に導入することにより,任意の位置が修飾されたタンパク質の調製が可能になる.
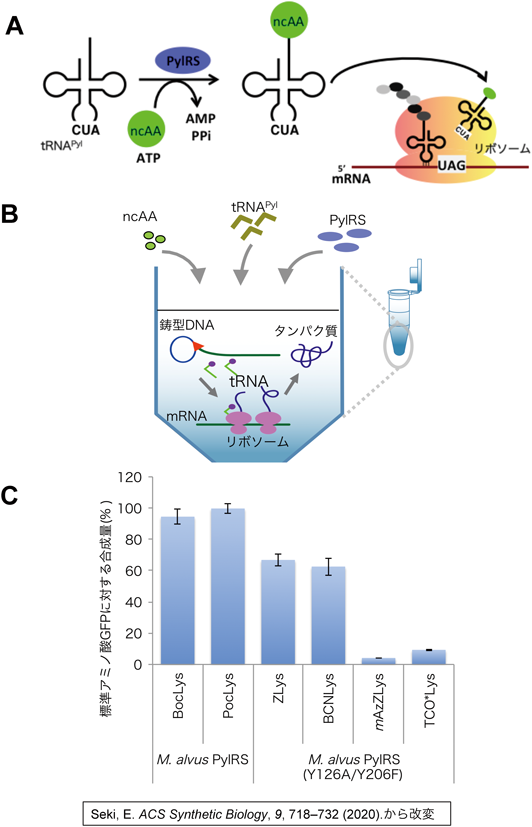
タンパク質への非標準アミノ酸導入は,標準の20種類のアミノ酸と相互作用しない(「直交する」),細菌や古細菌由来の特異的な転移RNA(tRNA)とアミノアシルtRNA合成酵素(aminoacyl-tRNA synthetase:aaRS)を,目的タンパク質を作らせる真核生物や細菌のタンパク質合成システムに導入することで行われる(図1A).特定のコドンを認識するtRNAに非標準アミノ酸を結合させるには,それぞれの非標準アミノ酸に対応するようにaaRSの改変が行われ,立体構造情報に基づく設計や,変異体ライブラリーを用いたランダムセレクションなどの手法を用いて開発されている6, 7).
非標準アミノ酸の導入には,さまざまな生物種の生きた細胞を用いてタンパク質を調製した例が報告されてきた.中でも大腸菌を用いた導入は,タンパク質の大量調製の優位性もあり,最も用いられている手法となってきた.近年では生きた細胞を使用しない,無細胞タンパク質合成法を用いた高効率な合成が報告されている(図1B).特に,大腸菌の細胞抽出液を用いた方法ではミリグラム量の合成が可能であり8),標準のアミノ酸と同等の導入効率を有した大量調製に成功している.本稿では,細胞抽出液を用いた無細胞タンパク質合成法による非標準アミノ酸導入の利点と実際の導入例について紹介する.
タンパク質の調製方法には,生体から精製してくる方法,生きた細胞を使用して強制的に発現させる方法,試験管内で生体のシステムを使用して合成する方法(無細胞タンパク質合成法,図1B),および化学的に合成する方法などがある.無細胞タンパク質合成法の一番の特徴は,試験管内でタンパク質を合成するため系が開いていることであり,それゆえにさまざまな利点を有している.
まず,細胞の生育に害のある条件下での合成や,細胞内に悪影響を及ぼすタンパク質の合成が可能である.いくつかの非標準アミノ酸は生きた細胞内への取り込みが困難だと報告されているが,これら非標準アミノ酸の使用も可能である.また,液体を添加するだけで合成条件が変更できるので,さまざまな因子を追加するなどの改良検討も容易である.PCRで調製した直鎖状DNA鋳型を直接使用でき9),タンパク質の多検体調製にも適している.生きた細胞を扱わないということは,遺伝子組換え実験や細胞増殖の待ち時間の必要なしに,生化学実験スペースさえあればタンパク質を合成できる,というのも利点の一つである.このように無細胞タンパク質合成法の特性を利用して,現在では膜タンパク質やタンパク質複合体などを含むさまざまなタンパク質が合成されているが10–14),標準の20種類のアミノ酸とは異なる合成条件が求められる非標準物質などを取り入れた合成や標識や修飾の導入などにおいても,きわめて適したタンパク質合成方法といえる.
無細胞タンパク質合成法は大きく分けて二つに分類できる.一つは大腸菌,コムギ胚芽,酵母,ウサギ網状赤血球,昆虫細胞,哺乳類培養細胞などの細胞抽出液を用いた合成系である.細胞抽出液に含まれる合成に必要なさまざまな因子をそのまま利用するのが特徴である.特殊なタンパク質の合成に必要な因子,たとえばシャペロンや,非標準アミノ酸導入に必要な本来は細胞内に存在しない外来因子等を細胞に強制発現させると,それらの因子を含んだ細胞抽出液を得ることができる.この強制発現させた細胞抽出液を用いると,これらの因子を個別調製することなく,通常のタンパク質と同等の容易さで難しい種類のタンパク質の調製が可能になる.難しいタンパク質の大量調製を簡便に行うのに適した系ともいえる.もう一つの合成系は,個別に精製した因子(転写,翻訳,エネルギー再生に必要な因子等)を用いた再構築型の合成系である15).再構築型の合成系はタンパク質の合成を阻害する因子や無関係の因子が混入しないため,合成産物の分解などが起こらない純度の高い合成なのが最大の特徴である.不純物を含まないので,人工細胞の構築など,合成生物学などの生命現象解明の研究に適した系といえる.また,現在では開発が進み,一部の細胞抽出液系と同等量の合成が可能になってきている16).
非標準アミノ酸を導入したタンパク質の合成において,特定コドン部位への導入効率はきわめて重要である.導入効率が低い状態でタンパク質合成を行うと,標準の20種類のアミノ酸での合成と比べて得られるタンパク質の量が低下する.複数部位へ導入する場合にはその影響は大きく,タンパク質合成量は導入部位の数に応じて指数的に低下してしまう17, 18).また,導入効率が低い状態でタンパク質を合成すると,望まないアミノ酸の挿入や,タンパク質合成の途中停止,ミスフォールディングなどが報告されており,合成したタンパク質の性質も低下することが問題になる.
非標準アミノ酸のUAG停止コドンへの導入における改善方法として,細胞内で停止コドンUAGを認識して,非標準アミノ酸導入用tRNAと競合する,翻訳終結因子を除いた大腸菌株の作製が複数報告されている17–21).これら改変株で非標準アミノ酸を導入すると,停止コドンUAGで伸長反応が停止したタンパク質の割合が減少し,非標準アミノ酸が導入された全長のタンパク質量が増加する.同様に,これらの改変大腸菌株の抽出液を用いて無細胞タンパク質合成を行うと,非標準アミノ酸が導入された全長のタンパク質量が増加し,導入効率が改善される7, 17, 20, 22–24).
非標準アミノ酸と導入に用いるaaRS/tRNAとの相性も導入効率に影響を与える.今までにさまざまな非標準アミノ酸導入に適した細菌や古細菌由来のaaRS/tRNAの改変と改良が報告されている.ピロリシン(pyrrolysine:Pyl)はメタン生成古細菌や複数の細菌において,天然でUAG停止コドンに導入される22番目のアミノ酸である25, 26).ピロリシンをアミノアシル化するピロリシルtRNA合成酵素(PylRS)とピロリシン用の転移RNA(tRNAPyl)の組合わせ(PylRS/tRNAPyl)は,細菌,古細菌,真核生物の翻訳システムのaaRS/tRNAと相互作用しないため,非標準アミノ酸導入においてとても有効なaaRS/tRNA系である(図1A).古細菌由来のPylRS/tRNAPylの組合わせでは,Methanosarcina mazei(M. mazei)やMethanosarcina barkeri(M. barkeri)などのPylRS/tRNAPylを用いた導入例が多く報告されてきた.立体構造情報に基づく設計で得た変異体や,変異体ライブラリーからのスクリーニングなどで,さまざまな大きさの非標準アミノ酸を導入するための改変PylRSが報告されている7, 22, 27–34).導入効率が高いものもあるが,依然導入効率が低いものも多い35).また,本方法では,立体構造が異なる新規の非標準アミノ酸を導入するたびに新たな変異体の探索が必要になるなどの課題もある.
近年,Methanomethylophilus alvus(M. alvus)のPylRS/tRNAPylが,複数の非標準アミノ酸においてM. mazeiよりも高い導入効率を示すことが報告された36–39).また,M. alvusのPylRSは安定で調製が容易であり,M. mazeiのPylRSと比べて20倍の収量を得られ,かつ5倍高濃度に濃縮が可能なことが明らかになった.これらは,非標準アミノ酸導入の合成系に使用するのに優位な性質であり,大量調製を行うに際にはとても重要な性質である.
4. 無細胞タンパク質合成法を用いた非標準アミノ酸の導入
1)M. alvusのPylRS/tRNAPylを用いた導入
細胞抽出液を用いた無細胞タンパク質合成法での非標準アミノ酸の導入は,通常のタンパク質合成反応液に3点の追加,すなわち非標準アミノ酸,PylRS, tRNAPylの添加だけで可能になる(図1B).上記の翻訳終結因子を除いた大腸菌抽出液を用いると,さらにより質のよいタンパク質調製が可能になる.
モデルタンパク質として改変緑色蛍光タンパク質(GFP)40)を合成すると,タンパク質合成反応液をそのまま蛍光測定するだけで無細胞タンパク質合成法での非標準アミノ酸の導入効率が簡便に確認できる.翻訳終結因子を除いた大腸菌抽出液20, 23)を用いた無細胞タンパク質合成反応液に1 mMの非標準アミノ酸と,それぞれ10 µMずつのM. alvus PylRSとtRNAPylを添加するだけで,ピロリシンに立体構造が近いボックリシン[Nε-(tert-butyloxycarbonyl)-L-lysine:BocLys]やポックリシン[Nε-propargyloxycarbonyl-L-lysine:PocLys]を導入したGFPは,それぞれ1 mL反応液あたり約2 mg得られた(図1C)41).これは,標準のアミノ酸だけで合成したGFPと同等のタンパク質量である.また,合成されたGFPは質量分析法での確認で,望まないアミノ酸の挿入や,途中で停止したもの等が含まれない,高品質の状態であることが確認できた.したがって,細胞抽出液を用いた無細胞タンパク質合成法では,高い収量で高品質のBocLysおよびPocLysが導入されたタンパク質調製が可能である.
ピロリシンよりも立体構造の大きいZリシン[Nε-benzyloxycarbonyl-L-lysine:ZLys]29, 34, 42)やビーシーエヌリシン[Nε-((((1R,8S)-bicyclo[6.1.0]non-4-yn-9-yl)methoxy)carbonyl)-L-lysine:BCNLys]36, 39)の導入では,M. mazei PylRSで知られたY306AとY384Fの変異と同等のM. alvus PylRSの変異体,(Y126AとY206Fの二重変異体(Y126A/Y206F),を用いることで高い合成量のGFPを調製できた(図1C).しかしながら,より立体構造が大きいメタアジドZリシン[Nε-(m-azidobenzyloxycarbonyl)-L-lysine:mAzZLys]やTCOリシン[Nε-((((E)-cyclooct-2-en-1-yl)oxy)carbonyl)-L-lysine:TCO*Lys]では,標準アミノ酸での合成量の20%以下の合成量となり,さらなる合成系の改良が必要なことが明らかになった.
2)立体構造解析をもとにしたPylRSの改変
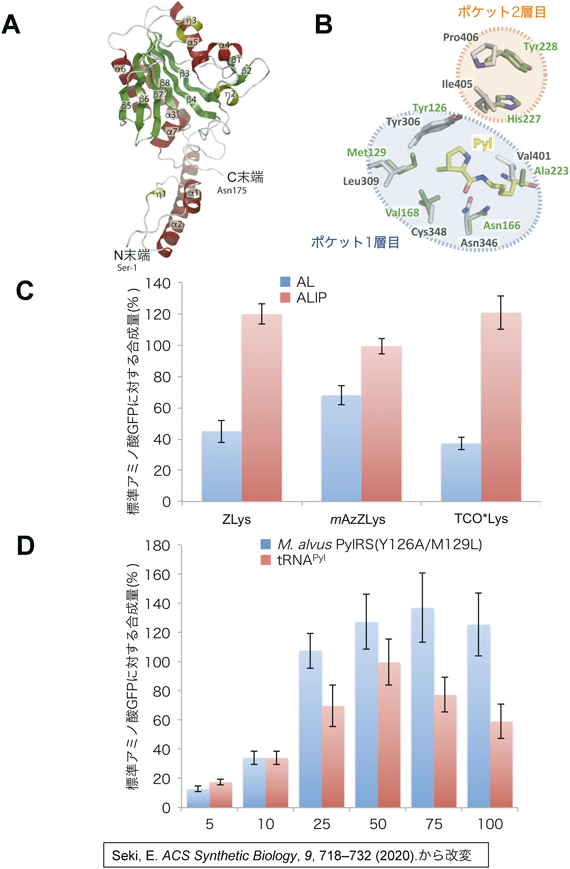
我々が決定した構造を含むM. alvus PylRSの立体構造(図2A)や他のPylRSの構造情報41, 43–52)を比較し,アミノ酸が結合するポケット状の立体構造と,mAzZLysやTCO*Lysの構造に基づいて,新たな変異体の設計を行った(図2B).M. alvus PylRSのアミノ酸結合ポケットを形成している部位を改変した複数の変異体の中で,mAzZLysとTCO*Lysに共通して合成量が上昇したのはY126AとM129Lの二重変異体(Y126A/M129L:AL)であった.しかしながらその合成量は,標準アミノ酸での合成量と比べてmAzZLysは70%前後,TCO*Lysは45%前後だった.
従来の設計では,非標準アミノ酸が直接に接するポケットの部位(アミノ酸結合ポケットの1層目,図2B)に変異を入れるのが一般的だった.そこで新たに,より広い範囲で,直接非標準アミノ酸に接しないが1層目のアミノ酸のいずれかと相互作用してポケットの立体構造に影響を与える部位(アミノ酸結合ポケットの2層目)を含めて設計を行うと,従来にはない,さまざまな非標準アミノ酸の導入に有効な変異体を作製することに成功した.H227IとY228Pの二重変異(H227I/Y228P)を先のM. alvus PylRS(Y126A/M129L)に追加して四重変異(Y126A/M129L/H227I/Y228P:ALIP)にすると,TCO*Lysの合成量は標準アミノ酸での合成量の121%(2.8 mg/mL)に上昇した(図2C).同様に,ZLysとmAzZLysにおいても合成量が上昇した.これらの合成されたGFPでも,望まないアミノ酸の挿入や,途中で停止したものが含まれておらず,高い効率で非標準アミノ酸が導入された質のよいタンパク質調製ができたことが明らかになった.なお,標準アミノ酸での合成量よりも高い合成量(120~130%)になったのは,このGFPの導入部位の配列で,DNAまたはRNA上の本来のコドン(GCC)が最適な配列でないため,アラニンが導入される際の転写または翻訳の効率が低くなっており,非標準アミノ酸の方が入りやすくなっているという逆転現象が生じていると思われる.
この四重変異(ALIP)の高い導入効率は,合成時のTCO*Lys使用量の削減を可能にした.GFPの合成では,従来の使用量1 mMから1/4の0.25 mMに削減しても同等のタンパク質が得られた.これは高額な非標準アミノ酸を用いたタンパク質の大量調製において大きな利点といえる.
この追加された二重変異(H227I/Y228P)の応用範囲は広く,アミノ酸結合ポケットの1層目の変異がZLysとは異なる種類の変異体にも有効なことが明らかになった.たとえばアセチルリシン(Nε-acetyl-L-lysine:AcLys)の導入では従来のM. mazei PylRSで知られた変異と同等のM. alvus PylRSの変異体(L121V/L125I/Y126F/M129A/V168F)に二重変異(H227I/Y228P)を追加すると合成量が2.6倍に上昇した.また,ヨードフェニルアラニン(3-iodo-L-phenylalanine:IPhe)でも,従来のM. mazei PylRSで知られた変異53)と同等のM. alvus PylRSの変異体(N166A/V168A)に二重変異(H227I/Y228P)を追加することで合成量が上昇した.
3)高濃度PylRSを用いた導入
非標準アミノ酸のアミノアシル化効率が低いことが原因の合成量の低下は,合成反応に用いるアミノアシル化されたtRNAの絶対数を上げれば改善すると期待できる.その場合,合成反応組成中に存在するPylRSとtRNAPyl量を上げるのだが,生きた細胞内でPylRSとtRNAPylの発現量を上げすぎると細胞増殖に悪影響を及ぼして合成量が下がることが報告されている.一方,無細胞タンパク質合成法は生育の影響を受けない試験管内での開いた系での合成である.非標準アミノ酸導入に必要な因子の添加量の調節をすることが容易であり,PylRSとtRNAPyl量の増量も容易であり,生きた細胞では不可能な濃度でのタンパク質合成も可能である.M. alvus PylRSは安定で可溶性が高く,M. mazei PylRSと比べて5倍濃縮可能で,高濃度で使用できるため,無細胞タンパク質合成反応液に大量に添加できる点が優れている.
先の二重変異(H227I/Y228P)追加による最適化をしていないM. alvus PylRS(Y126A/M129L)と合成量の低いTCO*Lysを用いて,PylRSとtRNAPyl量を変えたタンパク質合成を比較した(図2D).M. alvus PylRS(Y126A/M129L)の添加量を上げると,50 µM以上の濃度で標準アミノ酸での合成量に対して120~130%のGFPが合成されて一定になる.一方,tRNAPylの添加量を上げると合成量は一度上昇後に低下することから,過剰なtRNAPyl添加は,細胞の生育等とは関係なく,タンパク質合成自体に悪影響を及ぼすことが明らかになった.最適な合成条件である10 µM tRNAPylと50 µMのM. alvus PylRS(Y126A/M129L)を用いて合成されたGFPは,望まないアミノ酸の挿入や,途中で停止したものが含まれない,高品質な状態であることも確認できた.本方法は,単純に無細胞タンパク質合成反応液への添加量を変えるだけの簡単な方法であり,新しい非標準アミノ酸に適した変異体の探索が難しい場合において,とても有効な手段だといえる.
4)抗体への導入と蛍光標識
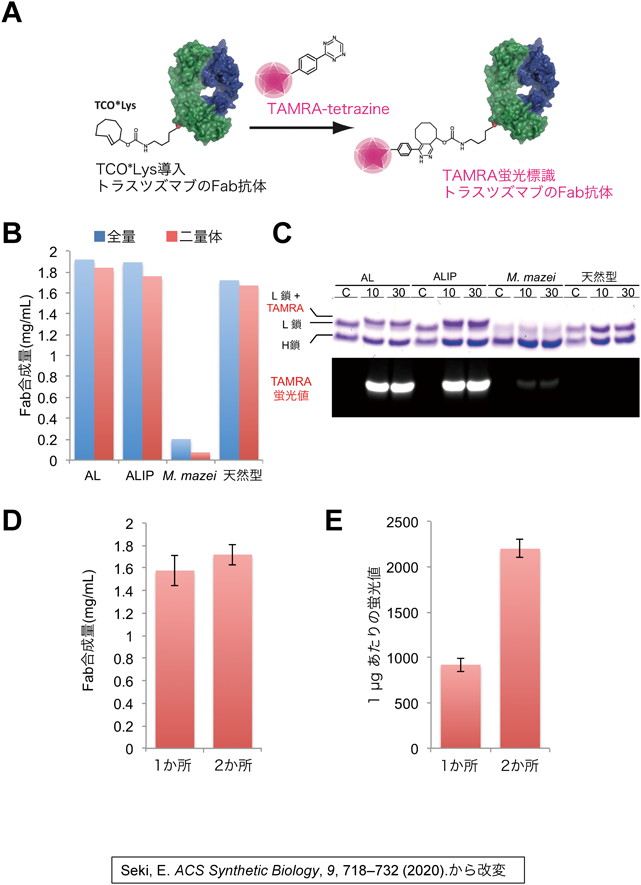
抗体薬物複合体(ADC)等に用いる,化合物を非標準アミノ酸にクリックケミストリーで結合する方法の一つであるstrain-promoted inverse electron demand Diels–Alder cycloaddition(SPIEDAC)は,反応性の高い方法である.中でもTCO*Lysを用いた方法は特段に反応性が高く,有用である(図3A)54, 55).抗体作製に適した無細胞タンパク質合成反応液56)でトラスツズマブのFab抗体にTCO*Lysを導入し,SPIEDAC反応でのTAMRAテトラジン(TAMRA-tetrazine)を用いた蛍光標識を行い,抗体作製効率と標識効率の確認を行った.2種類の合成方法,10 µMのM. alvus PylRSの四重変異体(Y126A/M129L/H227I/Y228P)と少量(0.2 mM)のTCO*Lysを用いた無細胞タンパク質合成と,高濃度(50 µM)のM. alvus PylRSの二重変異体(Y126A/M129L)と従来の1 mMのTCO*Lysを用いた無細胞タンパク質合成を行うと,それぞれ合成反応液1 mLあたり1.9 mg弱の精製TCO*Lys入りFab抗体が合成できる(図3B).それぞれのFab抗体は95%前後の効率で正しい二量体を形成している.また,Fab抗体には,望まないアミノ酸の挿入や,途中で停止したものが含まれないことも確認できた.これは標準アミノ酸だけで合成したものと同等の合成効率であった.一方,従来のM. mazei PylRSを用いた方法は,合成量が低く,かつ二量体形成率も低い状態にとどまった.
SPIEDACクリックケミストリー反応での蛍光標識体TAMRA-tetrazineの結合では,10分という短時間でほとんどの結合が完成した(図3C).これは従来の数時間かかる反応2, 54, 57)と比べると圧倒的な速さである.ほとんどのTCO*Lysを導入したFab抗体のL鎖はTAMRA-tetrazineの結合で高分子側に移動し,強い蛍光を示していることから,高効率で結合されていることが明らかである.M. alvus PylRSの四重変異体(Y126A/M129L/H227I/Y228P)での合成は,従来のM. mazei PylRSを用いた合成よりも,Fab抗体合成効率が9.4倍,1 µgあたり二量体形成効率が2.5倍であり,正しく二量体を形成した精製産物は20倍も高い.TAMRA-tetrazine結合効率,すなわち正しくTCO*Lysが挿入された効率は3.8倍高く,最終産物であるTAMRA-tetrazineが結合されたFab抗体は36倍多く調製できていた.
5)抗体の複数部位への導入
従来の低い効率の非標準アミノ酸の導入では,導入箇所が増えるごとに,対数的にタンパク質合成量が低下する傾向があった.しかし,高効率な非標準アミノ酸の導入が可能になった無細胞タンパク質合成法を用いると,トラスツズマブのFab抗体の同じL鎖上の2か所へ導入した場合,従来とは異なり,タンパク質合成量は低下せず,1か所導入と同等の高いままだった(図3D).TAMRA-tetrazineの結合部位が2倍になったことにより蛍光値は2倍になっており(図3E),2か所目も高品質でTCO*Lysが挿入されていることが明らかである.
このように,非標準アミノ酸の導入効率を上げることは,合成量だけでなく,合成品質の上昇につながることが明らかになった.これに対して,導入効率が低い状態で収量を上げるために単純に合成スケールを上げる手法では,品質の低いタンパク質の調製につながってしまい,非標準アミノ酸導入においては適さないことが示された.
無細胞タンパク質合成法を用いた一部の非標準アミノ酸導入において,立体構造解析に基づいて改良したM. alvus PylRSを用いることで,標準のアミノ酸導入と同等の品質で導入できることが可能になった.従来の非標準アミノ酸に接する位置の変異に加え,直接非標準アミノ酸に接しないがポケットの立体構造に影響を与える部位(アミノ酸結合ポケットの2層目)の二重変異(H227I/Y228P)を加えることで,TCO*Lys等の複数の大きい構造の非標準アミノ酸の導入が,標準アミノ酸と同等の効率で導入することが可能になった.
SPIEDACクリックケミストリー反応は,銅を用いない,迅速で反応性の高い方法である.その中でも,TCO*Lysを用いたSPIEDACクリックケミストリー反応の速度は特段に速く,ADC等を含むタンパク質の標識に有効な方法である.我々の開発したM. alvus PylRSを用いた無細胞タンパク質合成法でのTCO*Lysの導入では,標準アミノ酸と同等まで導入効率を上げることが可能になった.Fab抗体への標識効率は従来の36倍まで上昇し,合成に使用するTCO*Lysの使用量を80%減少させることで合成費用の削減にも成功した.本方法で調製したFab抗体は,収量が上昇しただけでなく,二量体形成やクリックケミストリー反応の点からも高品質だった.
タンパク質に非標準アミノ酸を導入する技術は,生命科学や医療の研究などにとても有用であり,今後もさまざまな種類の非標準アミノ酸の導入が行われるであろう.新しい形状の非標準アミノ酸に対し,最適な変異体の開発が望ましいが,困難な場合もある.必ずしも最適な変異体でなくても,生きた細胞では不可能なaaRS濃度を使用して無細胞タンパク質合成で導入すると,標準のアミノ酸と同等の導入効率での合成が可能になったことはとても重要である.これは,従来の,細胞の生育に害のある条件下での合成や,細胞内に悪影響を及ぼすタンパク質の合成に追加して,無細胞タンパク質合成法を用いた非標準アミノ酸導入の新たな利点といえるだろう.