現在普及している医薬品の多くは,有機分子などの低分子医薬品および抗体などの高分子医薬品であるが,両者にはいずれも医薬品として一長一短がある(表1).たとえば,低分子は従来から医薬品の主流として用いられてきたが,標的タンパク質に対する特異性が低いため毒性・副作用の懸念がある.一方の高分子は,低分子の弱点を克服する高い標的特異性を持ち優れた活性を有するものも多いが,短所として腸管吸収性が低く経口投与が不可能である点,細胞膜透過性を持たないため標的が細胞表面の受容体などに限られる点,さらに製造コストが非常に高い点などがあげられる.
表1 医薬品群の比較 | 低分子医薬品 | 中分子医薬品 | 高分子医薬品 |
|---|
| 形態 | 有機小分子 | 特殊環状ペプチド | 抗体 |
| 分子量 | 500以下 | 1000~3000 | 150,000以上 |
| 標的特異性 | 低い | 高い | きわめて高い |
| 毒性・副作用 | 多い | 少ない | 少ない |
| 経口投与 | 可能 | 一部可能 | 不可能 |
| 細胞膜透過性 | 高い | 低い~高い | なし |
| 製造コスト | 低い | 低い~普通 | 高い |
| 免疫原性 | 低い | 低い | 普通~高い |
| 標的結合面積 | 狭い | 普通~広い | 広い |
| PPI*阻害 | 一部可能 | 可能 | 可能 |
| 生体内安定性 | 低い~普通 | 普通~高い | きわめて高い |
| *PPI:タンパク質間相互作用. |
これに対し,低分子と高分子の長所を併せ持つ次世代の中分子医薬品として,ペプチドが近年注目されている.ペプチドは,低分子の長所としての低製造コスト,低免疫原性といった特徴に加えて,高分子の長所としての標的特異性,標的に対する広い結合表面積,タンパク質間相互作用(protein–protein interaction:PPI)阻害能1),低毒性・副作用といった特徴を持つ.中でも,非タンパク質性アミノ酸(特殊アミノ酸)を含有し大環状骨格を持つ「特殊環状ペプチド」は,20種類のタンパク質性アミノ酸(19種類のL-α-アミノ酸およびグリシン)のみからなる「通常のペプチド」と比較して高い腸管吸収性および細胞膜透過性を有する2, 3).通常のペプチドは消化管内や血中のプロテアーゼにより数分程度で分解されてしまうため4),医薬品としてはインスリン(1型糖尿病治療薬)など数が限られる一方で5),特殊環状ペプチドはその特殊骨格の寄与により医薬品として必要な生体内安定性を獲得している.
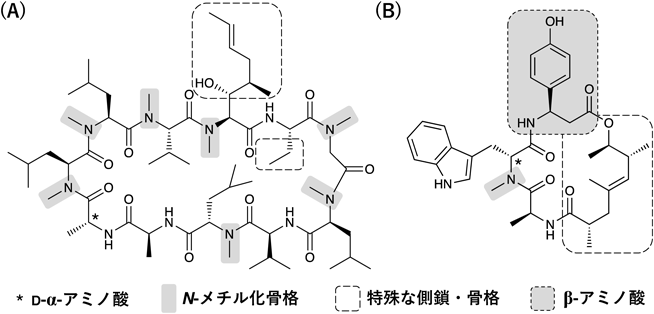
このような特殊環状構造は,微生物が産生する生理活性ペプチドの中に多く含まれており,実際に抗生物質や抗がん剤,免疫抑制剤等の医薬品として利用されている.たとえば,自己免疫疾患の治療に用いられるシクロスポリンは,細胞内標的であるシクロフィリンと複合体を形成して免疫反応の中枢であるカルシニューリンの活性化を阻害し,結果的に免疫抑制効果を発揮する(図1A).特筆すべきは,その投与方法が静脈注射のみならず経口投与も可能なことである.この高い生体内安定性および細胞膜透過性,腸管吸収性をもたらしているのは,シクロスポリンを構成する11アミノ酸残基からなる大環状骨格,そして骨格のほとんどを占める特殊アミノ酸である.配列中のD-α-アミノ酸はプロテアーゼ耐性の向上に加え,L-α-アミノ酸のみでは形成しえないフォールディングを誘起することでペプチドの二次構造安定化や生理活性に貢献していると考えられる.また,7か所のN-メチルアミノ酸および2か所の非天然アルキル側鎖を持つL-α-アミノ酸はペプチドの疎水性を向上させ,細胞膜透過性や腸管吸収性に寄与している.別の例として,腫瘍細胞増殖抑制効果を示し細胞膜透過性を有するコンドラミドCの構造中には,N-メチル-D-α-アミノ酸に加えてβ-アミノ酸が含まれている(図1B)6).β-アミノ酸は通常のペプチドとは異なるヘリックス構造やターン構造を誘起し,ペプチドの二次構造を安定化させることで標的特異性,生体内安定性,細胞膜透過性の向上に寄与することが知られている7).このように,特殊アミノ酸の種類によって特殊環状ペプチドの活性や安定性,さらには二次構造に与える影響が大きく異なるため,ペプチド医薬品開発においては多種多様な特殊アミノ酸を含有するペプチドを網羅的に取りそろえ,ハイスループットスクリーニングに適用することが強力な手段となる.そのためには,天然からの発見に頼らず,人工的な特殊環状ペプチドライブラリーを用いることが有用である.
たとえば,現在広く用いられている人工的なペプチドライブラリー構築法の一つとして,スプリットプール法と呼ばれるペプチド固相合成法を基盤とした有機化学的手法がある8).この方法では配列中にタンパク質性アミノ酸のみならず特殊アミノ酸を導入可能であるが,問題点としてライブラリー構築に膨大な時間を要すること,合成スケールの関係で106程度の多様性が限界であること,スクリーニング後のヒット配列の同定が煩雑であることなどがあげられる.一方,リボソーム翻訳系を用いることで大規模なペプチドライブラリーの合成とハイスループットスクリーニングが可能である.たとえば,ファージディスプレイ法9, 10)やmRNAディスプレイ法11, 12)では,それぞれ109ないし1013種類に及ぶペプチドをワンポットで合成しそのままスクリーニングに用いることができる上に,ペプチドの配列情報はそれをコードするDNAのシーケンシングにより容易に得られる.しかし,上記のシクロスポリンやコンドラミドCのような特殊環状ペプチドは一般に非リボソームペプチド合成酵素13)や翻訳後修飾酵素14)といった専用の酵素群によって生合成されており,利用可能なアミノ酸が20種類のタンパク質性アミノ酸に限られたリボソーム翻訳系では直接合成できないという問題がある.
そこで我々は,D-α-アミノ酸やβ-アミノ酸等の特殊アミノ酸を利用可能なリボソーム翻訳系の開発を目指し,人工翻訳系「Flexible In vitro Translation(FIT)システム」の開発を行ってきた.FITシステムでは,既存のタンパク質性アミノ酸を特殊アミノ酸に置き換えて翻訳を行うことで特殊環状ペプチドを合成できる.さらに,FITシステムとmRNAディスプレイ法を組み合わせたスクリーニング法「Random non-standard Peptides Integrated Discovery(RaPID)システム」の開発により,1013規模の特殊環状ペプチドライブラリーを用いたハイスループットスクリーニングを可能にした.本稿では,FITシステムの根幹となる「遺伝暗号リプログラミング」の概念を解説した後,近年確立された改良型FITシステムによるD-α-アミノ酸やβ-アミノ酸の連続導入の達成や特殊アミノ酸基質許容性の拡大について述べる.続いて,RaPIDシステムを用いたスクリーニングの原理とその事例を紹介する.
1)遺伝暗号リプログラミング
翻訳系とは,生体内においてリボソームがペプチドやタンパク質の生合成を行う代謝系である.その際,mRNA配列を鋳型とし,タンパク質性アミノ酸を基質とした正確かつ高速なペプチド重合反応が行われる(原核細胞では毎秒40アミノ酸の重合を触媒する).mRNA上の三つの連続する塩基(3塩基の組み合わせをコドンという)とアミノ酸の対応関係は遺伝暗号と呼ばれ,地球上の生物種に広く保存されている.翻訳系では,アミノアシルtRNA合成酵素(aminoacyl-tRNA synthetase:aaRS)が固有のtRNAとそれに対応するアミノ酸を結合させ(アミノアシル化反応),生じたアミノアシルtRNAはリボソームに運び込まれたのちペプチド重合反応に用いられる.この際,mRNAのコドンとtRNAのアンチコドンとの対合が確認されることで,鋳型mRNAどおりのペプチド配列へと変換される.この合成機能を改変して特殊アミノ酸を重合反応の基質に用いることができれば,特殊ペプチドを迅速かつ簡便に翻訳合成することが可能になる.ただし,単に特殊アミノ酸を翻訳系に加えただけではtRNAのアミノアシル化が行われず,特殊ペプチドが合成されることはない.また,人工的に合成した特殊アミノアシルtRNAを加えたとしても,同じコドンと対応関係を持つタンパク質性アミノ酸が共存している状態では,通常のペプチドと特殊ペプチドの両方が合成されるため,配列制御の面から望ましくない.
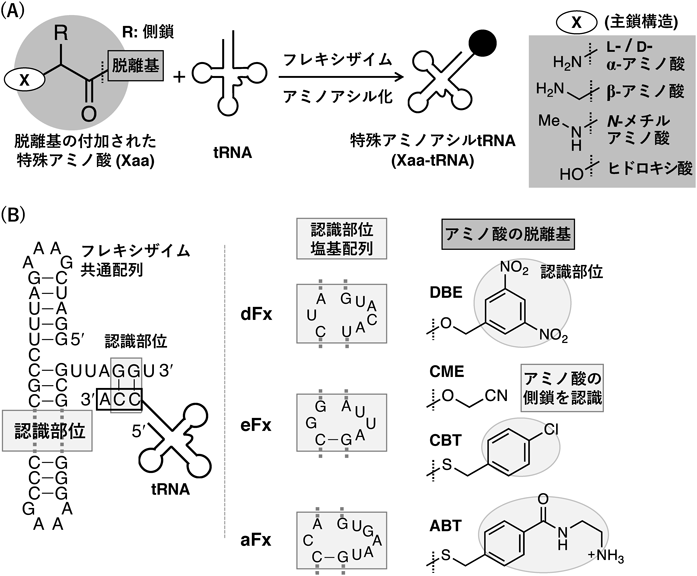
ここで鍵となる概念が「遺伝暗号リプログラミング」である.この概念は,天然の遺伝暗号が規定するコドンとタンパク質性アミノ酸との対応関係を解消し,新たに特殊アミノ酸との対応関係に書き換えることである.遺伝暗号リプログラミングにより特殊ペプチドを合成した例はいくつか存在するが15, 16),我々のFITシステムでは人工RNA酵素「フレキシザイム」および「再構成無細胞翻訳系」を組み合わせることで翻訳系を利用した特殊ペプチドの合成を達成した17).
我々の開発したフレキシザイムは,任意のtRNAとアミノ酸とのアミノアシル化反応を触媒するRNA酵素である(図2A).2001年に最初のフレキシザイムが報告されて以降18),改良が重ねられ,現在は主に3種類のフレキシザイム,ジニトロフレキシザイム(dFx),エンハンストフレキシザイム(eFx),そしてアミノフレキシザイム(aFx)が用いられている(図2B)19, 20).アミノアシル化に用いるアミノ酸基質は事前に脱離基の付加による活性化が必要であり,dFxに対しては3,5-ジニトロベンジルエステル(DBE)基,eFxに対してはシアノメチルエステル(CME)基あるいは4-クロロベンジルチオエステル(CBT)基,aFxに対しては4-[(2-アミノエチル)カルバモイル]ベンジルチオエステル(ABT)基を用いる.フレキシザイムは,反応点であるカルボニル基と,アミノ酸側鎖あるいは脱離基の芳香環のみを認識している.したがって,主鎖構造が天然のL-α-アミノ酸と異なるD-α-アミノ酸やβ-アミノ酸,N-メチルアミノ酸,さらにはアミノ基を持たないヒドロキシ酸をもアミノアシル化の基質にすることができる.また,アミノ酸側鎖の芳香環の有無によって脱離基を使い分けることができるため,側鎖の異なる基質に対しても幅広い許容性を持つ.
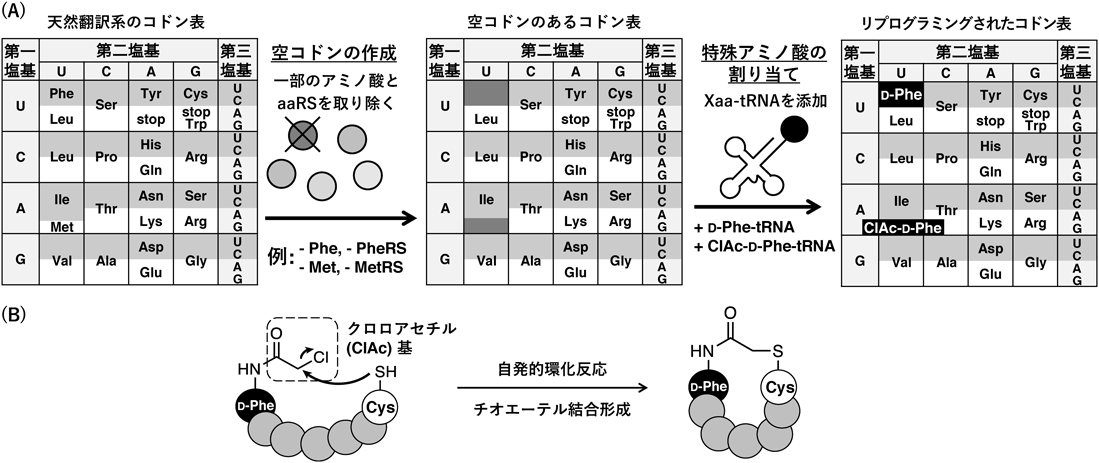
一方,再構成無細胞翻訳系では試験管内に単離精製したリボソーム,翻訳因子等の酵素,tRNA,タンパク質性アミノ酸および対応するaaRSなどの必要な因子群を適切な濃度で混ぜ合わせることで,人工的な翻訳系を再構築することができる21).その際,任意の酵素やアミノ酸を除くことで空コドンを作製可能であり,たとえばPheおよび対応するaaRS(PheRS)を除くことで,PheのコードされていたUUUおよびUUCコドンを空コドンにすることができる(図3A).そして,アンチコドンGAAを持つtRNAに対してフレキシザイムを用いてD-Pheをアミノアシル化し,この系中へと加えることで,UUC(およびwobble塩基対を組むUUU)コドンがD-Pheへとリプログラミングされた翻訳系を構築できる.
FITシステムでは,上記のような伸長コドンのリプログラミングだけではなく,開始コドンのリプログラミングも可能である.原核生物のmRNAの開始コドン(AUG)には本来N-ホルミルメチオニン(fMet)が割り当てられているが,リプログラミングにより任意の特殊アミノ酸を割り当ててペプチドのN末端に導入できる.この手法を利用してクロロアセチル(chloroacetyl:ClAc)基を持つClAc-D-Pheで翻訳を開始し,このペプチドの下流にCysを配置することで,Cysのチオール基とClAc基の間で自発的な分子内求核置換反応を起こし,生体内で安定なチオエーテル結合を介したペプチドの大環状化が可能である(図3B)22).このように,遺伝暗号リプログラミングとフレキシザイムを組み合わせたFITシステムを利用することで,ワンポットで特殊環状ペプチドの翻訳合成が可能となった.2016年までに,D-α-アミノ酸23, 24),β-アミノ酸25),N-メチルアミノ酸26–28),非タンパク質性側鎖を持つアミノ酸29),さらにはヒドロキシ酸(ポリエステルの合成)30)などさまざまな特殊アミノ酸を含有する特殊ペプチドの合成が達成された.
2)改良型FITシステムによる特殊アミノ酸基質の拡大
FITシステムを用いてペプチド鎖中に多様な特殊アミノ酸を導入できるようになったものの,シクロスポリンのように多数の特殊アミノ酸を連続して導入することは難しかった.2016年時点のFITシステムにおいて連続導入可能な特殊アミノ酸は,特殊な側鎖を持つL-α-アミノ酸,あるいはある種のN-メチルアミノ酸に限られており(3種類のN-メチルアミノ酸を10残基連続で導入することに成功した26)),特殊骨格として重要なD-α-アミノ酸やβ-アミノ酸はペプチド鎖中に1残基ないし不連続に2残基程度の導入が限界であった.そこで近年,これらの特殊アミノ酸の連続導入に向けたtRNAの改変や翻訳因子の最適化が行われた.その結果,構築された改良型FITシステムではD-α-アミノ酸やβ-アミノ酸の連続導入のみならず,これまで用いることのできなかった多様な特殊アミノ酸の導入が実現され,特殊ペプチド合成の大きな進展をもたらした.本項では,改良型FITシステムが構築された経緯について解説する.
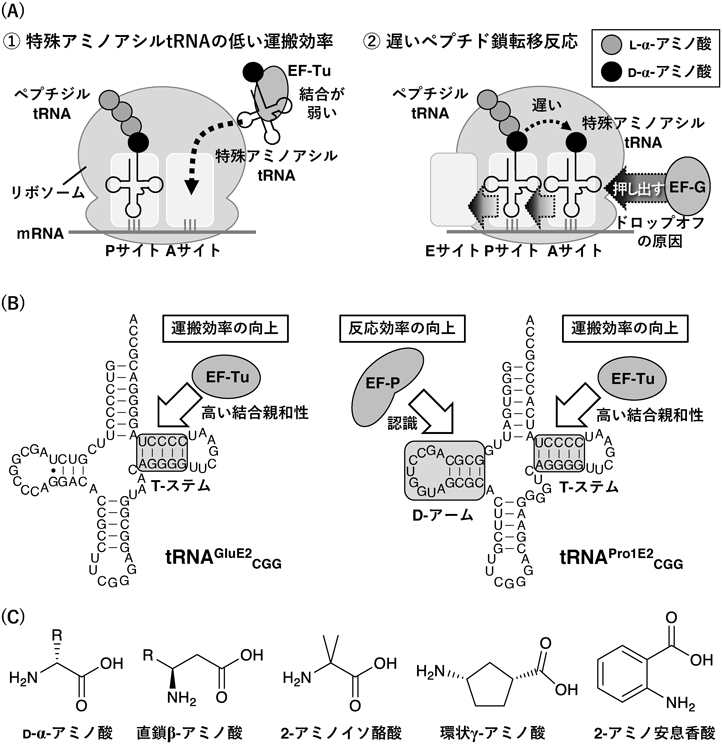
D-α-アミノ酸等の特殊アミノ酸の連続導入ができなかった要因として主に二つの点があげられる(図4A).まず,①特殊アミノ酸のアミノアシル化されたtRNA(特殊アミノアシルtRNA)がリボソームのAサイトへ適切に運搬されない点である.アミノアシルtRNAを運搬する伸長因子EF-Tu(elongation factor thermo unstable)は,タンパク質性アミノ酸のアミノアシルtRNAの認識に適した構造をとっており,特殊アミノアシルtRNAに対する結合親和性が低い.そのため,Aサイトへの運搬効率が低く連続導入には対応できなかった.もう一つは,②リボソームの触媒環境に対して特殊アミノアシルtRNAが適応していない点である.特殊アミノアシルtRNAがリボソームのAサイトへと運び込まれたとしても,PサイトのペプチジルtRNAとの間のペプチド結合形成(ペプチド鎖転移反応)が遅く,結合が形成される前に伸長因子EF-G(elongation factor G)によってペプチジルtRNAが押し出され,ドロップオフを起こしてしまう.その結果,不完全な短いペプチド鎖が生成する.これら二つの要因に対し,我々はそれぞれ以下の方法でFITシステムの改良を行った.
まず要因①に対して,EF-Tuと特殊アミノアシルtRNAの間の結合力を高めるために,tRNAのT-ステム領域の塩基配列を最適化した(図4B)31).大腸菌のtRNAGluのT-ステムはEF-Tuとの結合親和性が特に高いことが知られているため,tRNAGluを基盤としてアンチコドンループを改変したtRNAGluE2を作製し,アミノアシル化に用いた.加えて,3種類の翻訳因子EF-Tu, EF-G, IF2[開始因子2(initiation factor 2)]の濃度を最適化した.特殊アミノアシルtRNAのAサイトへの運搬効率の促進に向けてEF-Tu濃度を増加,ペプチジルtRNAのドロップオフを引き起こすEF-G濃度を減少,ペプチド全体の翻訳効率に関わるIF2濃度を増加させたことで,最終的にD-Ala含有ペプチドの発現量を従来のFITシステムと比較して5倍に増加させることに成功した.また,この最適化された翻訳系により,最大10残基のD-Serの連続導入が達成された.
要因②に対しては,ペプチド鎖転移反応の促進を図るために翻訳伸長因子EF-Pの導入とtRNAのD-アーム領域の最適化を行った.EF-Pは天然の翻訳系において,連続したPro間のペプチド鎖転移反応を促進することが知られており32, 33),我々はEF-Pが天然のtRNAPro1のD-アーム構造を認識していることを明らかにした34).実際に,D-AlaをtRNAPro1にアミノアシル化して翻訳反応を行ったところ,EF-P存在下で翻訳効率の大幅な向上が確認された35).そこで,tRNAPro1に対し前述のtRNAGluE2のT-ステム部位を導入した人工tRNAを設計し,EF-TuとEF-Pの両方から翻訳反応の促進を受けるtRNAPro1E2の開発に成功した(図4B).実際に,tRNAPro1E2に対してD-Alaをチャージし,連続した2残基のD-Alaを含有するペプチドの発現量を定量したところ,EF-Pを加えることで翻訳が5倍促進される結果となった35).このように,EF-P存在下でtRNAPro1E2に特殊アミノ酸をチャージすることで,連続導入の促進が確認された.
上記の改良型FITシステムにより,4残基のD-α-アミノ酸(ClAc-D-Phe,D-Ser,D-Ser,D-Cys)のみからなる特殊環状ペプチドの合成が可能となった他35),直鎖β-アミノ酸を7残基連続でペプチド鎖へと導入することにも成功した36).直鎖β-アミノ酸の連続配列からなるペプチドは,10-,12-,14-ヘリックスなどの特徴的な構造をとり,高い安定性を持つことが知られているため,医薬品候補として期待される.さらに,これまで翻訳導入例のなかった2-アミノイソ酪酸(Aib,小川の稿参照)35),環状β-アミノ酸(cβAA)37),環状γ-アミノ酸38),さらには主鎖に芳香環を持つ2-アミノ安息香酸誘導体39)を含有する特殊ペプチドの合成に成功し,AibやcβAAについてはそれぞれ3残基,10残基の連続導入が可能であった(図4C).いずれの特殊アミノ酸を持つ特殊ペプチドについてもチオエーテル結合による大環状化が可能であり,改良型FITシステムにより合成可能な特殊環状ペプチドの多様性が一気に広がった.
3. 特殊環状ペプチドスクリーニング法「RaPIDシステム」
1)RaPIDシステムの概要
続いて我々は改良型FITシステムによる特殊環状ペプチド翻訳合成技術をハイスループットスクリーニングによる薬剤探索へと展開した.RaPIDシステムでは,1013規模の多様性を持つ特殊環状ペプチドライブラリーを用いて標的タンパク質に対するアフィニティーセレクションを行うことにより,わずか10日程度で高活性な(pMからnMオーダーの標的結合能および阻害能を持つ)特殊環状ペプチドを獲得することができる40).本節では,RaPIDシステムによるセレクションの仕組みについて解説した後,近年報告された二つのスクリーニング事例について紹介する.
RaPIDシステムは,FITシステムによる特殊環状ペプチド合成法とmRNAディスプレイ法を組み合わせた技術である41).mRNAディスプレイ法とは,試験管内で翻訳合成されたペプチドとその鋳型mRNAを連結させた状態にすることで,ペプチドライブラリーからセレクションした活性ペプチドの配列をmRNA情報の解析によって同定可能にする手法である11, 12).具体的には,鋳型mRNAの3′末端にピューロマイシン(アミノアシルtRNAの3′末端と構造が類似)を連結させた状態で翻訳反応を行う.反応が進行してmRNAの3′末端に近づくと,ピューロマイシンがアミノアシルtRNAの代わりにリボソームのAサイトに取り込まれ,伸長中のペプチドのC末端とピューロマイシンが共有結合により連結された状態で翻訳が停止し,翻訳ペプチド–鋳型mRNA複合体が得られる.この反応をFITシステムにおいて行うことで,特殊環状ペプチド–mRNA複合体が容易に合成できる.この方法であれば,ランダム配列を持つmRNAライブラリーを用意するだけで,わずか30分程度の反応時間で対応する特殊環状ペプチドライブラリーが翻訳合成され,その多様性は数百μLの溶液中で1013にも上る.化学合成法によって作られたライブラリー(106程度)とは桁違いの多様性かつ簡便さである.
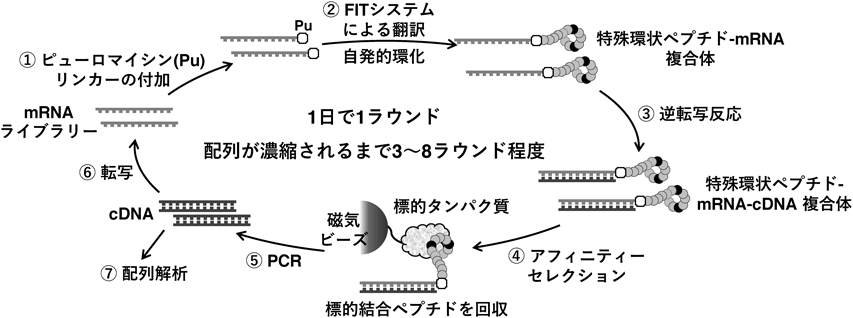
RaPIDシステムによる活性ペプチドのセレクションは以下の手順で進行する(図5,①~⑦).まず上記のとおり,mRNAランダムライブラリーに対し,ピューロマイシン(Pu)の付加されたリンカーをライゲーションする(①).Puの付加されたmRNAライブラリーを鋳型としてFITシステムにより翻訳合成を行い,特殊環状ペプチド–mRNA複合体ライブラリーを合成する(②).mRNAの逆転写反応により特殊環状ペプチド–mRNA–cDNA複合体ライブラリーを形成する(③).標的タンパク質をあらかじめ固定化した磁気ビーズと,③で形成した複合体ライブラリーを混合し,標的タンパク質に結合した特殊環状ペプチドの複合体を回収する(④).回収したペプチド複合体の持つcDNAをPCRにより増幅させ(⑤),続けて転写反応を行うことで,セレクションにより得られた標的結合ペプチドの鋳型配列からなるmRNAライブラリーを生成し(⑥),次のラウンドに用いる.この1ラウンド(手順①から⑥)を3~8回程度繰り返すことで,標的結合能の高い配列が濃縮される.最後に,回収されたcDNAを次世代シーケンサーにより網羅的に解析することで,標的に対する結合能を持つ特殊環状ペプチド配列が同定できる(⑦).1日で1ラウンドのセレクションを行うことができ,配列の同定まで含めてもわずか10日程度で候補ペプチドを取得可能な画期的なスクリーニング法である.
2)スクリーニング事例1:23種類のアミノ酸を含有する特殊環状ペプチドライブラリーを利用したIL6R結合ペプチドの取得42)
ここでは,近年のRaPIDシステムによるスクリーニング事例について解説する.1例目は,23種類の疎水性アミノ酸(12種類のタンパク質性アミノ酸および11種類の特殊アミノ酸)を一度に翻訳系へ導入し,スクリーニングを行った事例である.
シクロスポリンなどの天然の生理活性特殊環状ペプチドの多くは,その構造中に疎水性側鎖を持つアミノ酸やN-メチル化された主鎖を持つアミノ酸を多く有しており,低分子医薬品と同様に細胞膜透過性を有する.一方で,一般的なin vitroスクリーニング法(mRNAディスプレイ法やファージディスプレイ法など)によって得られる環状ペプチドは,高い標的結合能や阻害能を示す場合でも細胞膜透過性が低いことが多い.なぜなら,遺伝暗号リプログラミングを行わずに翻訳した場合は,ペプチド鎖を構成するタンパク質性アミノ酸の化学的性質(親水性・疎水性,電荷の有無)を制御できず,性質の異なるアミノ酸が偏りなく含まれたペプチド配列が得られることが多いためである.そのため,タンパク質性アミノ酸のみからなる環状ペプチドを医薬品として利用するには,細胞膜透過性を付与する工夫が必要とされていた.
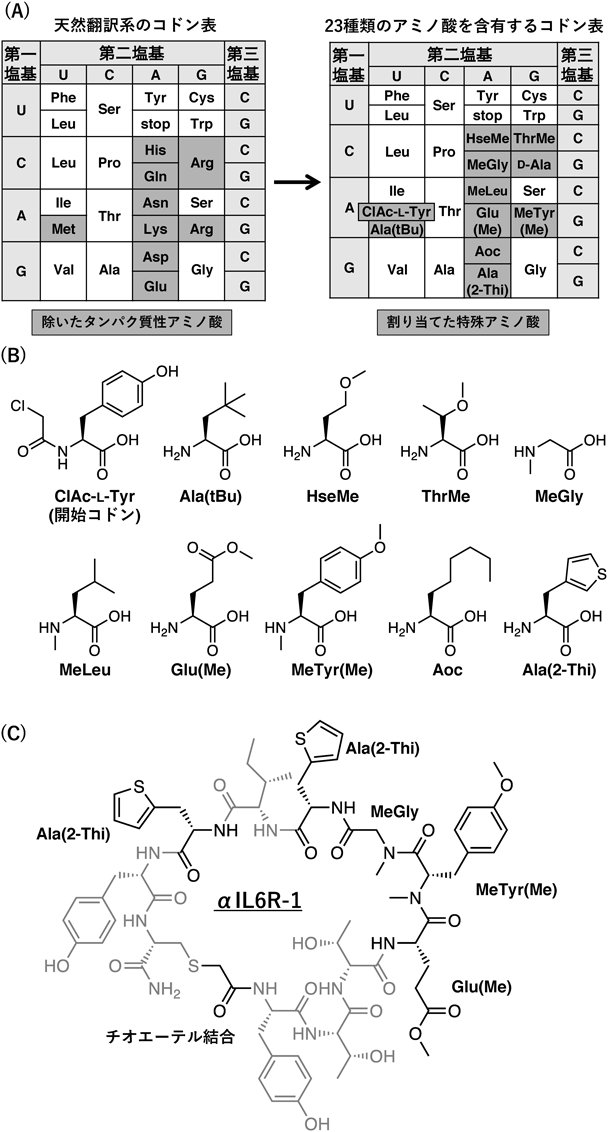
そこで,我々は疎水性ペプチドの取得に特化した翻訳系の構築を目指し,以下のように遺伝暗号をリプログラミングした.7種類の親水性タンパク質性アミノ酸(Gln, Glu, Asn, Asp, Lys, Arg, His)およびMetを除いて作製した空コドンに対し,D-Alaおよび主鎖や側鎖にメチル化が施されたアミノ酸を中心とした10種類の疎水性特殊アミノ酸を割り当てた(図6A, B:この翻訳系においては第三塩基がUまたはAとなるコドンは使用しないためコドン表から割愛してある).ここで,合計のアミノ酸が23種類となり,天然に用いられる20種類を超えていることに注目したい.天然のコドン表において利用されるアミノ酸が20種類に限られる要因の一つとして,天然tRNAの多くは一つのtRNAが複数(2~4か所)のコドンに対応することが知られている.これは,天然tRNAにはRNAを構成する4塩基U, C, A, Gに加えて修飾塩基と呼ばれる特殊な塩基が含まれており,複数の塩基に対してwobble塩基対を形成するからである.それに対し,再構成無細胞翻訳系では天然tRNAを取り除き,代わりに修飾塩基を持たない人工tRNAを利用することで,tRNAとコドンの対応関係を制限することができる.たとえば,今回取り除いたArgはもともとCGC, CGG, AGGコドンに割り当てられていたが,人工tRNAを利用することでArgの代わりに三つの異なる特殊アミノ酸を割り当てることに成功した.また,MetのAUGコドンについては,開始AUGコドンと伸長AUGコドンにそれぞれ異なる特殊アミノ酸[ClAc-L-TyrおよびAla(tBu)]を割り当てることで特殊アミノ酸の種類を増加させることができた.このように,23種類ものアミノ酸を含む翻訳系が構築されたことで,多種多様な疎水性特殊環状ペプチドをワンポットで合成可能になった.
この翻訳系を用いてペプチドライブラリーを構築し,がん細胞の増殖や分化に関わるインターロイキン6受容体(interleukin-6 receptor:IL6R)を標的としたスクリーニングを行った.その結果,強い結合能(結合解離定数KD=44 nM)を示す特殊環状ペプチドαIL6R-1が得られ,その構造には5残基の疎水性特殊アミノ酸およびチオエーテル結合が含まれていた(図6C).このように,天然の特殊環状ペプチドを模倣した疎水性ペプチドライブラリーの構築とスクリーニングへの応用が達成された.
3)スクリーニング事例2:環状β-アミノ酸を含有するフォルダマーペプチドの取得37)
2例目は,特殊環状ペプチドに環状β-アミノ酸(cβAA)を導入することで,高い標的結合能,標的阻害能,生体内安定性を実現した事例である.β-アミノ酸を含有するペプチドは剛直性の高い安定な二次構造をとることが知られており,このようなペプチドはフォルダマーと呼ばれ医薬品への応用が期待されている7).中でも,主鎖に環状炭素骨格を持つcβAAはペプチドの二次構造形成に対して強く寄与するため,特殊環状ペプチドのさらなる安定化や標的結合能の向上が見込まれる.そこで我々はcβAAを含有する特殊環状ペプチドライブラリーを構築し,RaPIDシステムによるスクリーニングを試みた.
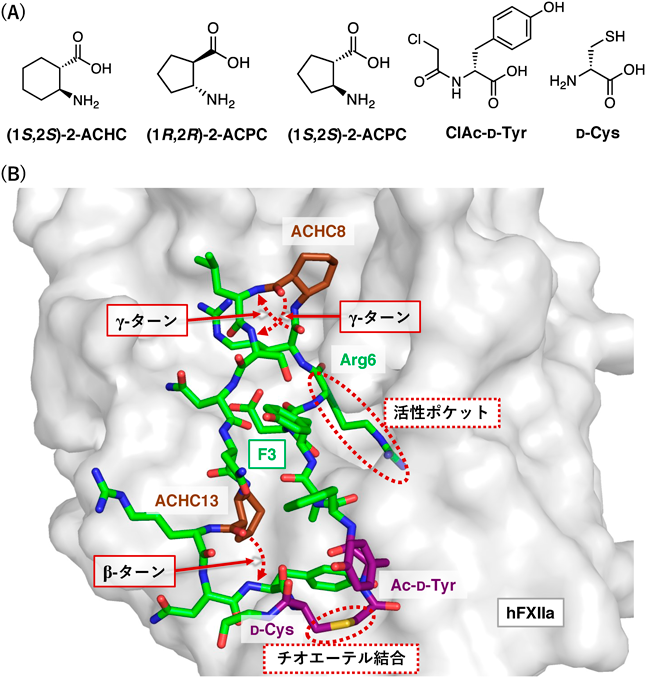
前述の改良型FITシステムを用いてcβAAの導入を試みたところ,六員環骨格を持つ2-aminocyclohexanecarboxylic acid(2-ACHC)および五員環骨格を持つ2-aminocyclopentanecarboxylic acid(2-ACPC)について,それぞれ4種類の立体異性体すべてを翻訳合成に用いることに成功した.それらのうち3種類のcβAAを含む15種類のアミノ酸によるランダム配列を持ち,その両端には大環状骨格を形成する2種類のD-α-アミノ酸(ClAc-D-TyrおよびD-Cys)が固定された特殊環状ペプチドライブラリーを構築した(図7A).ヒト活性化血液凝固第12因子(human activated factor XII:hFXIIa)に対するスクリーニングを行った結果,高い結合能を持つcβAA含有特殊環状ペプチドが多数得られ,中でも2つの(1S,2S)-2-ACHCおよび2つのD-α-アミノ酸を含有するF3は非常に高い結合能(KD=0.98 nM)および阻害能(阻害定数Ki=1.5±0.2 nM)を示した.さらに,F3を血清中に加えてプロテアーゼ耐性を測定したところ,その半減期は59時間と非常に高い安定性を示した.ネガティブコントロールとして,F3の2か所の(1S,2S)-2-ACHCをAlaに置換したペプチドF3Aの半減期を測定したところ13時間にまで減少したため,cβAAが生体内安定性に貢献していることが確認できた.一方で,F3Aの残る2か所のD-α-アミノ酸とこれらが形成する大環状骨格の寄与により,通常のペプチド(数分程度で分解)よりも高い安定性を有することも示された.
さらに,F3のhFXIIaに対する作用機序や,ペプチドの二次構造に対するcβAAの寄与を解明するために,hFXIIaとF3のX線共結晶構造解析を行った(図7B).得られた回折像からは,F3が二つの(1S,2S)-2-ACHC(ACHC8およびACHC13)によって折りたたまれ,シート構造をとりながらhFXIIaに結合していることがわかった.具体的には,ACHC8はhFXIIaの酵素活性中心付近に位置し,2種類の分子内γ-ターンによる安定な逆平行β-シート構造を誘起していた上,その環状炭素骨格がhFXIIa表面の疎水性ポケットと疎水性相互作用により結合していることが確認された.また,ACHC8の誘起するγ-ターンの近傍では,Arg6が酵素の活性ポケットに入り込んでおり,F3によるhFXIIaの阻害機構が判明した.また,ACHC13は活性中心から遠い位置で分子内β-ターンを構築し,ペプチドを安定化させていた.
このように,わずか2残基のcβAAおよび2残基のD-α-アミノ酸がペプチド鎖に導入されることによって,特殊環状ペプチドの構造的な安定性や生体内安定性,さらには結合能や阻害能が大きく向上した.この剛直なシート構造をとる特殊環状ペプチドF3は,フォルダマーと呼ぶのにふさわしいだろう.今後は,cβAAを他の特殊アミノ酸と組み合わせることや,ヘリックスペプチドの合成に応用することにより,ペプチドライブラリーの構造多様性や活性の向上が見込まれる.