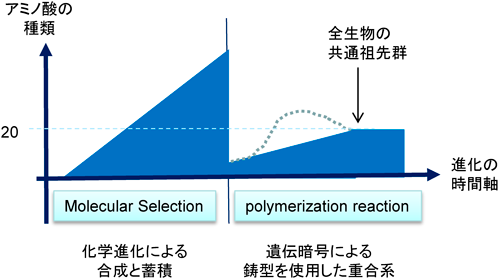
1. はじめに:生物が用いるアミノ酸ビルディングブロックの数についてのシナリオ
鋳型依存的に複製することが可能な核酸の配列情報に従ってアミノ酸を重合させる原始遺伝暗号が成立して以後,遺伝暗号で使用されるアミノ酸の数がだんだん増えてきたと考えられている2–4).さらに,重合の正確さも向上してきただろう.重合装置の識別能力が未発達な原始的な暗号系では,一つのコドンに対して側鎖の物理化学的性質が類似した複数のアミノ酸を指定せざるをえなかった状況から,特定のアミノ酸のみを指定できるようになる進化である.この意味では,遺伝暗号に含まれるアミノ酸の数は,増えてきたとも,減ってきたともいえる(図1).
アミノ酸の数が増えるメリットの第一は,新規な官能基をタンパク質に付与することにある.また,立体構造中での官能基の位置を微調整することで,酵素活性や基質特異性を調節できることもある.生体高分子の進化能の観点からは,ある活性を持つ酵素について,中立変異の数が増えることで,遺伝子重複後にその酵素から別の活性が生まれる確率を上昇させられることにある5).このような中立経路の概念は,RNAの進化についての配列空間の観点からも論じられてきた6).
アミノ酸の数が増えることにはデメリットもある.それは,個々の分子の認識能力が向上したとしても,システムの構成要素が増えたことに起因して避けることができない,合成の忠実度の低下である.坂本の稿に記されるように,遺伝暗号拡張の研究の鍵は,20個共存しているアミノ酸・tRNA・アミノアシルtRNA合成酵素(aaRS)のグループについて,新しいグループを加える際に,各グループの排他性を保つことにある7).これらの研究では,不完全な排他性から生じる新奇なアミノ酸の天然アミノ酸コドンへの混入や,新奇なアミノ酸用のコドンに天然アミノ酸が混入してしまう問題を解決することが必要であった.現在の標準遺伝暗号に19, 20番目のアミノ酸が加わってきた際にも,同様の問題が生じたかもしれない.
本稿でこの遺伝暗号の進化の議論を進めるために,まず,遺伝暗号の成立以前のRNAとアミノ酸およびペプチドそれぞれの化学進化について整理する.続いて,遺伝暗号による酵素の生産が可能になった時点よりも前と後それぞれについて,あるアミノ酸が標準アミノ酸たるための条件を考察する.その条件は,下記(A)~(C)の三つに大別される.(条件A)遺伝暗号に含まれるアミノ酸は,供給される必要がある.(条件B)遺伝暗号に含まれるアミノ酸は,重合系で使用できる必要がある.現在の生物の重合系が,あるアミノ酸を使用するか否かの関門が三つある.最初の関門は,アミノ酸とtRNAを結合させるaaRSである.遺伝暗号の成立前はリボザイムが,成立後はタンパク質酵素がこの役割を担っている.二つ目の関門は,アミノアシルtRNAをリボソームに供給させるEF-Tuである.三つ目の関門は,リボソームとなる.(条件C)生態系で標準の遺伝暗号が一時的にでも偶然凍結されると8),ここからの逸脱は遺伝子の水平伝搬による利益を失ってしまうため,後から入り込むアミノ酸は,生物にメリットを提供できなければ固定されない.
遺伝暗号に関わる合成生物学の進展により,本特集の多くの記事が紹介する新奇アミノ酸を追加できる拡張遺伝暗号の創出だけでなく,本記事が後述するように,どのようなmRNAからもいくつかの特定の標準アミノ酸を含まないポリペプチドを合成する「単純化遺伝暗号」も構築されている.これを用いたタンパク質の人工進化実験は,過去のタンパク質がどこまでの性能を発揮できるか,ということの検証を可能とするだけではない.メチオニンは酸化されてしまう,など,特定のアミノ酸が一定の使用条件下では弱点となってしまうことがある.一見非力にみえるこの単純化遺伝暗号は,そのような弱点となるアミノ酸を持たないままタンパク質の人工進化を可能にすることができる.また,タンパク質合成の正確性が低い過去型の遺伝暗号に従った進化の様相も,そのような人工遺伝暗号を用いたタンパク質の人工進化から解析することが可能となる.興味深いことに,適切な翻訳エラーは,進化の局所解トラップの問題を解決できる可能性がある.
2. 遺伝暗号の成立以前に並行進化したペプチドとRNAの出会いと,その後の統合プロセス
「RNAワールド」の概念は古くからあり8, 9),現在では化学進化でそれぞれ生じたRNAとペプチドの相互作用を活用しつつ,RNAを遺伝情報の主体とする生命の一段階を想定した研究が進んでいる.しかし,「RNAワールド」の語を明示的に示した1986年の記事において10),生体高分子としてRNAのみを使用する世界としてRNAワールドが記されたため,研究者間の議論に混乱が生じることがあった.実際,化学進化で合成されるアミノ酸の量に対してヌクレオチドの化学進化実験で生じる条件が狭いことが,「RNAのみワールド」に対する否定的な意見となっている.RNA研究者にとっても,RNAワールドにおける化学進化由来ペプチドの存在を前提として,ペプチドによるリボザイムの高機能化に焦点を当てた研究が進んでいる.たとえば,実験進化の分野で,複製リボザイムの機能がペプチドで活性化される,という研究も進展している11).これらの研究では,核酸鋳型によってアミノ酸配列を指定できる以前の段階の化学進化で蓄積した特定の配列のペプチドを,リボザイムが活用した,という見方になる.また,リボソームの立体構造で,コアなところはRNAが主体でありつつ,リボソームタンパク質の一部領域がタンパク質様の立体構造を形成せず,RNAに構造を支えられたペプチドとしてペプチド転移部位近傍に位置していることも12),ペプチドとRNAの相互作用による共進化の仮説を後押しする.
遺伝暗号の成立以前に,特定の配列のペプチドが濃縮されうるだろうか.最も単純なシナリオは,原始地球環境下で合成される種々のアミノ酸のうち,安定なアミノ酸が重合し,さらに分解されにくいペプチドが蓄積する,というメカニズムになる.近年では,ジペプチドが化学進化を促進させた,ということを模した実験が,RNAの重合や膜構造の化学進化実験で報告されている13, 14).もし,化学進化で生じるペプチドが自身のアミノ酸モノマーの合成反応や両アミノ酸間の重合反応を促進するならば,それは鋳型依存的ではないまでも,自己複製の起原ともいえる.ただし,このような反応で,立体構造を形成可能な長さまでアミノ酸が重合したポリペプチドが合成されるとは考えにくい.
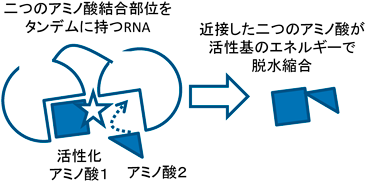
非リボソーム性のペプチド合成のように(小笠原と勝山の記事参照),RNAが鋳型依存的ではなくとも特定の配列のジペプチドを合成できた可能性は高い(図2).アミノ酸に結合できるRNAアプタマーを連結して,ジペプチドを合成しようという試みがあり15),同様の実験で,活性化アミノ酸ともう一つのアミノ酸の近接効果により,若干の重合反応の促進も報告されている16).また,アミノ酸をATPで活性化するリボザイムも報告されている17).
遺伝暗号の成立以前に供給されたペプチドの寄与は生命の起原の研究に無視できないものの,それらを供給する合成メカニズムは,進化の次の段階では核酸鋳型によって配列を指定できる翻訳系による合成に乗っ取られたであろう.セントラルドグマによるポリペプチド配列の進化メカニズムの強みは,遺伝子として複製可能なデジタル配列である核酸の1塩基置換によって,ペプチド配列中の1アミノ酸を置換し,その変異ペプチドの活性に応じてこれをコードする遺伝子の生き残り確率を変化させられることにある.デジタルな配列の場合の数は,ビルディングブロックの数の残基数乗であり,これは膨大な数である.そのため,離散的な配列でありつつ,タンパク質の原子団の立体配置を連続的に変化させる試行が,セントラルドグマによって可能になる.一方,非リボソーム型ペプチド合成酵素(NRPS)様の合成によってでは,遺伝子の種々の1塩基置換に対応してさまざまな1アミノ酸置換を試行することは困難であろう.このペプチドの進化能力の差によって,遺伝暗号の成立以前のペプチド供給メカニズムは,現在の遺伝暗号の直接の祖先である翻訳系による供給メカニズムに乗っ取られることになる.つまり,化学進化で供給されていたペプチドをコードする遺伝子を翻訳系が探し当てると,その後は遺伝子の変化によってペプチドの進化が始まり,進化した結果の配列は翻訳系によってのみ生産される,というシステムの乗っ取りが生じる.
3. 条件A:遺伝暗号に含まれるアミノ酸は,供給される必要がある
現在の生態系で使用される標準アミノ酸分子は,生態系のいずれかの生命が生合成して供給されたものがほとんどであろう.一方,初期の遺伝暗号のメンバーとなったアミノ酸は,化学進化で供給され,生命が発生した原始地球環境において安定である必要がある.ACGUがRNAのヌクレオチドに選択された際にも,“molecular selection”という概念が紹介されている18).本稿では,遺伝暗号の成立によるタンパク質酵素の出現を境として,化学進化による供給と生合成による供給に区分する.現在の代謝系でアミノ酸前駆体を供給する中心代謝系の補酵素の多くがヌクレオチド骨格を持つために,RNAワールドにおけるアミノ酸の生合成も興味い研究対象であるが,現時点では未解明な点が多く,本稿では論じない.
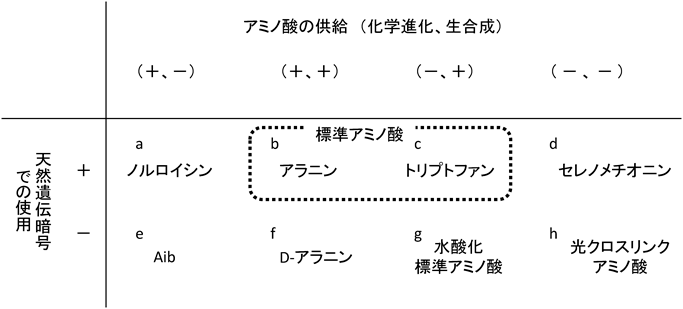
化学進化で合成され,標準遺伝暗号を持つ生物によってタンパク質に取り込むことが可能であっても,標準遺伝暗号に含まれていないアミノ酸があることは(図3a),生合成系も標準アミノ酸を規定していることの証左である.遺伝暗号改変の研究の初期でメチオニンの代替アミノ酸として培地に添加してタンパク質合成に使用されたノルロイシンは(坂本の記事参照)19),化学進化で合成もされるが,生合成ではほとんど生産されないため,天然タンパク質には検出されない.セレノメチオニンも同様にメチオニン合成系欠損株でタンパク質に取り込ませることが可能であり,結晶構造解析の重原子として重用されたが,これは化学進化でも生合成でも供給されないアミノ酸である(図3d).各種アミノ酸の生合成のバランスは,翻訳エラーを軽減するためにも重要である.もし細胞内のアミノ酸プール内での各アミノ酸の濃度比が変化すると,翻訳エラーが生じてしまい致命的になりうるだろう.
ミラーの化学進化実験の驚きは,単純な化合物から酵素なしに合成された高分子の分解によって,アラニンなどの標準アミノ酸が供給されうることを示したことにある(図3b).一方,標準アミノ酸のいくつかは,古典的な化学進化実験では合成されにくいということも知られており,これらは,酵素による生合成が可能になってから遺伝暗号システムに参画したのであろう(図3c).ある標準アミノ酸が別の標準アミノ酸の前駆体であった場合に,これらのアミノ酸のコドンの差異が1塩基にとどまっており,古代の暗号では同一のコドンセットであった,という“coevolution theory”の主張もある3, 20).また,酵素による生合成系の確立以前の標準アミノ酸は,合成されるだけでなく,化学的な安定性も重要となる.原始の地球にはオゾン層がなく,地表または浅水には紫外線が大量に照射されていた.このため,紫外線によって分解されやすいCys, Met, His, Phe Tyr,およびTrpは初期の標準アミノ酸としては使用されにくかった,という指摘がある21).加熱によって脱アミド化してしまうGlnとAsnにも同様の安定性の課題がある.一方,近年においても,より多くの種類の標準アミノ酸が化学進化によって供給されうることを示した実験が続いている.最近,Sutherlandらは,アルギニンとアスパラギンの前駆体の非生物的合成経路を報告している22).さらに,ヒスチジンの非生物的合成についての報告もある23).
現在の生物の生合成によって,古くから指摘されるオルニチンに限らず,本特集の多くの記事で示されたように,magic20以外にも多くのアミノ酸が生産される.これら生合成される非標準アミノ酸と標準アミノ酸との違いは,重合系による選択(polymerization selection)にある.オルニチンが標準遺伝暗号に含まれていない理由は,オルニチン専用のaaRSが存在しないためである.化学進化実験や太陽系内の各種環境で,D-アミノ酸やα-amino-n-butyric acidも合成されるが,現在の翻訳系はこれらをタンパク質に取り込むことは難しい(図3e, f).一方,ひとたびtRNAにアミノアシル化しさえすれば,多くの非標準アミノ酸はタンパク質に取り込ませることが可能である(坂本,横山,寺本,菅の記事参照).アミノアシルtRNAと結合してこれをリボソームに運搬するEF-Tuも,標準アミノ酸を選択する要素となりうる.一連の芳香族性非標準アミノ酸の取り込み実験から,Trpのように芳香環が横に伸びた非標準アミノ酸が,縦に伸びた非標準アミノ酸よりも取り込みが悪いことは24),Trpに対して現在の翻訳系が適応していないことを示しているのかもしれず,Trpが標準アミノ酸として最終盤で加わったことの名残かもしれない.EF-Tuとアミノ酸の親和性の差が,EF-TuとtRNAの親和性の差によって補完され,どの標準アミノ酸でアミノアシル化されたtRNAもEF-Tuとの同等の結合力を持っており,この同等性が翻訳の精度に重要である,という報告もある25).
リボソームもまた,標準アミノ酸を規定する重合装置である.プロリンはイミノ酸である,という点で,他の標準アミノ酸と異なっていることと対応して,プロリンが頻度高くコードされている場合,翻訳を補助する因子であるEF-Pが重要となることが知られている.さらに,菅の記事が示すように,EF-Pを多く添加することで,リボソームは天然型であっても,D-アラニンやα-amino-n-butyric acidもタンパク質に取り込まれる.両アミノ酸は,L-アミノ酸では水素原子となる位置に原子団としては小さいサイズのメチル基が位置する,という意味で,立体障害が小さいため可能になったと思われる.さらに,リボソームの立体構造が現在とは大きく異なったものとして,L-アミノ酸よりもD-アミノ酸を好んでいたリボソームが存在した可能性もある.ただし,田村らが示したように,活性化アミノ酸とRNAとの結合を酵素なしで行わせた場合のリボースとアミノ酸の光学活性の対応関係により26),D-リボースとL-アミノ酸の対応関係がRNAワールドで決定され,RNPワールドまで引きずられた可能性がある.
aaRSの進化は,化学構造が類似したアミノ酸群をひとまとめにしたコドンセットが,それぞれのアミノ酸特異的なコドンに分割されていった様子も記録している.その典型的な例が,立体構造解析によって明かされてきた,脂肪族アミノ酸に対するaaRS群である.メチオニン,ロイシン,イソロイシン,バリンはコドン2文字目がUで共通しており,また,これらそれぞれのaaRSの立体構造のコア部分はきわめて類似している.さらに,MetRSを除くこれらaaRSにはコア部分に追加された校正ドメインがあり,本来の基質ではないが類似した構造を持つ標準アミノ酸が結合してしまったtRNAやその中間産物を積極的に分解している27).古代においては,これら脂肪族標準アミノ酸および,標準アミノ酸以外で類似した化学構造を持つアミノ酸がまとめて,コドン2文字目がUであった大きなコドンボックスに指定されていたのであろう.標準遺伝暗号表中のアミノ酸の配置として,1塩基置換が生じても化学的な性質が似たアミノ酸を指定するようになっている,ということがある.このような配置は,遺伝暗号上のアミノ酸の配置が多種試されて現在の配置が選ばれた,という場合だけでなく,上述したaaRSの校正機能の進化によるコドンの細分化によっても生じうる.同様に,グルタミン,アスパラギン,システインのtRNA上における前駆体標準アミノ酸からの生合成も28, 29),物理化学的性質が類似したアミノ酸間のコドン塩基組成の類似性の原因となる.
5. 条件C:遺伝暗号に後から入り込む標準アミノ酸は,生物にメリットを提供できなければ固定されない
微生物に頻繁にみられる遺伝子の水平伝搬は,遺伝暗号の「標準化」を進める原動力となる.微生物は,他の生物が進化によって発明した遺伝子を取り入れることで,進化競争の舞台に立ち続けている30, 31).もし遺伝暗号が異なれば,他の生物が進化によって発明したDNA分子を獲得してもその遺伝子産物は機能しない.たとえば,ワープロソフト一太郎が本邦でもMS Wordsに置換されていったように(また表計算ソフトやその他ソフトの栄枯盛衰も同様に),デファクトスタンダードから逸脱してしまった場合,マイナーなソフトが優れていたとしても,そのソフトを使用し続けるデメリットは大きい.ミトコンドリアや寄生体としてのマイコプラズマでは遺伝暗号が標準とは異なっているが,これらは水平伝搬DNAを受け取りがたい細胞内共生体である.遺伝暗号を改変してピロリシンの導入を行っている古細菌は,メタン代謝を行うための特殊な残基を活用することと引き換えに,進化の袋小路に入ったともいえる.遺伝暗号の差異が水平伝搬の障壁となることを工学的に活かす方法として,それぞれ異なる遺伝暗号に対して専用の遺伝子を構築することで,組換え体の予期しない進化を防ぐことも可能であることが提案され実験でも示されている32–34).
現在の生命だけでなく,生命の共通祖先の段階(最後の共通祖先ではなく)においても遺伝子の水平伝搬がみられたという視点に立つならば35),ある時点の標準からの逸脱を意味する遺伝暗号へのアミノ酸の追加は,既存の生物からの遺伝子の水平伝搬による獲得の効率を低下させることよりも大きなメリットがなくてはいけないことになる.標準アミノ酸ではないがmRNAの構造依存的にリボソーム上でのタンパク質合成に用いられるセレノシステインは,その強い酸化還元能力に特徴があるが,いくつかの生物でこの導入系を放棄している事例がみられる36).このような新規アミノ酸の脱落は,新規採用のメリットが,環境変化によりメリットではなくなってしまったためかもしれない.本稿の冒頭で述べたように,遺伝暗号中のアミノ酸の数が増えることは翻訳の忠実度の低下につながる.リボソームでの合成で使用するアミノ酸の数を20から増やすことのデメリットも大きくなっているのだろう.また,リボソームによる合成を経ずとも,生物は翻訳後修飾やNRPSによって非標準アミノ酸を高分子中で活用することができる.
ある時点で標準アミノ酸として採用されたアミノ酸が遺伝暗号に残り続けるためには,そのアミノ酸の化学的特性によるメリットに頼るという方策以外に,そのアミノ酸が一斉に他のアミノ酸に置換されると多くの遺伝子産物の機能が失われてしまうようにする,という方策がある.新規採用されたアミノ酸がaaRSなどの翻訳系タンパク質に複数部位で使われ,また,その新規アミノ酸の導入による構造の乱れを補正する二次的な置換が蓄積していくと,翻訳系はその新規アミノ酸を手放すことはできなくなる.AsnやGlnは標準遺伝暗号の最終段階で加わったアミノ酸であると考えられているが,もしある酵素のすべてのAsn残基をGln残基に置換すると,活性が大幅に低下してしまうことが想定される.このような観点から,現在の標準アミノ酸のいくつかは,現時点でそのアミノ酸の特性がもたらすメリットがなくなる,または小さくなっているとすると,そのアミノ酸は「利己的な」アミノ酸として振る舞っているともいえる5).あるアミノ酸を一斉に置換するとタンパク質の活性がなくなってしまう,という点に注目し,必須遺伝子に非天然アミノ酸残基を導入し,その残基を標準アミノ酸に置換すると活性がなくなるようにすることで,その非天然アミノ酸が培地に存在しない限り組換え体が死滅してしまう,という安全装置も開発されている37).
6. magic20が決まる前の状況の天然進化を模した実験室内進化
現在の標準遺伝暗号が定まるよりも前,アミノ酸の数が20種類未満であった時代のタンパク質の性質を知るために,アミノ酸の種類が限定された単純化タンパク質の研究が進んでいる.これまでも,9~13アミノ酸からなる限定されたセットでタンパク質を構成し,その活性を確認することが行われていたが,対応する天然タンパク質の活性よりも活性がきわめて低いことが知られている38–40).しかし,この結果をもって,そのアミノ酸セットでのタンパク質の性能を示したことにはならない.なぜならば,天然のタンパク質が進化を経て活性が上昇し維持されていることに対して,これら単純化タンパク質は,人間の知恵を投入したデザインが行われてはいるものの,進化サイクルによる活性向上が試みられていないためである.タンパク質の機能向上には,人間によるデザインは非力で,進化工学が必要となったことに相当する.そこで単純化タンパク質の進化工学が望まれていたが,標準遺伝暗号を使用する限り困難であった.その理由は,限定したセットに含まれない標準アミノ酸への復帰変異で活性が向上してしまう,偽陽性のためである.この問題の解決のために,どのような遺伝子からも特定のアミノ酸を含まないタンパク質を合成する単純化遺伝暗号の開発が必要であった32, 41).
筆者の研究室では,リシンを持たずとも活性を向上させたインターフェロンや,トリプトファンを持たずとも活性を持つGFPの開発を報告しており42, 43),遺伝暗号の進化についての理学的な観点から,より少ないアミノ酸種でのタンパク質の進化が可能であるかを現在追求している.アミノ酸種を限定した単純化タンパク質は,野生型のタンパク質に比べて非力になると想像されることが多いが,工学的観点から特定の使用環境下では標準アミノ酸が弱点となることがあり,そのアミノ酸を持たない単純化タンパク質に強みが生まれる.遺伝暗号改変の初期の研究であった,メチオニンの代わりにノルロイシンを使用したEGFの合成も,メチオニンの硫黄原子が酸化されてしまう問題を回避するための方策であった19).同様に,産業用酵素やタンパク質製剤からメチオニンやシステインを除去するために,単純化遺伝暗号を使用した人工進化が有効となるであろう.また,上述したリシンを持たないインターフェロンの開発も42),この変異体の任意の位置をリシン置換することで,そのリシン残基を標的としてタンパク質を部位特異的にPEG修飾することが目的であった.
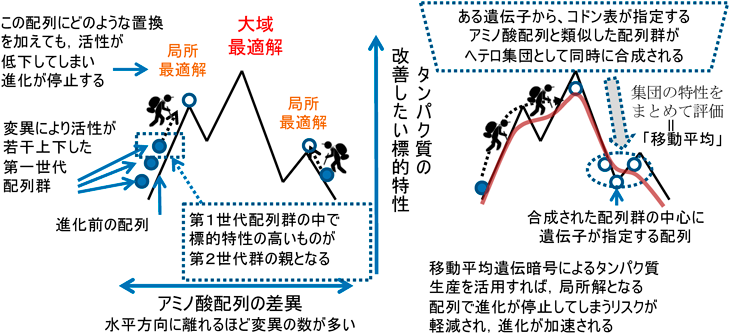
過去の遺伝暗号による進化の様相を追及するためには,現在よりも翻訳エラーが多い状況での人工進化を観察することも重要である.抗生物質の添加も一つの手段であるが,特定のコドンに対してアラニン,セリン,ロイシンの混入度合いを調節できる人工遺伝暗号表の活用も有効な手段となるであろう.興味深いことに,このような翻訳エラーが,遺伝子への変異と選択を繰り返す進化における局所解でのトラップを緩和することが想定されている(図4)44).翻訳エラーが,遺伝子への変異がなくともプロテオームを多様化して適応を可能にする,という種々の報告と併せ45),翻訳エラーが利点となる研究の理学的・工学的な広がりが期待される.
本特集であらためて確認できたように,種々の非標準アミノ酸が生合成可能であり,遺伝暗号も多種構築可能であることが明らかになった.現在の地球生命の標準暗号は,いくつかのコドンの配置は物理化学的に決まった可能性も否定できないものの,64コドン全体の配置は偶然凍結された,と考えることが妥当である.火星探査が進んで火星に微生物が見つかり(宇宙探査初期の汚染でないことを確認する必要がある),その遺伝暗号が地球の標準遺伝暗号と同一であった場合,隕石に付着した微生物が両惑星間を移動した,というパンスペルミア仮説が確からしくなる.遠い将来人類が別の恒星系で見つけた生物が地球の遺伝暗号と同じであった場合,遺伝暗号は文字どおりの“universal” genetic codeとなる.ただし,微生物が恒星系間を宇宙船なしに移動する確率はきわめて低く,何らかの先史文明が生物を恒星系間に広めた,ということになるだろう.
引用文献References
1) Crick, F.H., Griffith, J.S., & Orgel, L.E. (1957) Codes without Commas. Proc. Natl. Acad. Sci. USA, 43, 416–421.
2) Ikehara, K., Omori, Y., Arai, R., & Hirose, A. (2002) A novel theory on the origin of the genetic code: A GNC-SNS hypothesis. J. Mol. Evol., 54, 530–538.
3) Wong, J.T. (2005) Coevolution theory of the genetic code at age thirty. BioEssays, 27, 416–425.
4) Shepherd, J.C.W. (1984) Fossil remnants of a primeval genetic-code in all forms of life. Trends Biochem. Sci., 9, 8–10.
5) Amikura, K. & Kiga, D. (2013) The number of amino acids in a genetic code. RSC Advances, 3, 12512–12517.
6) Schuster, P., Fontana, W., Stadler, P.F., & Hofacker, I.L. (1994) From sequences to shapes and back—A case-study in rna secondary structures. Proc. Biol. Sci., 255, 279–284.
7) Kiga, D., Sakamoto, K., Kodama, K., Kigawa, T., Matsuda, T., Yabuki, T., Shirouzu, M., Harada, Y., Nakayama, H., Takio, K., et al. (2002) An engineered Escherichia coli tyrosyl-tRNA synthetase for site-specific incorporation of an unnatural amino acid into proteins in eukaryotic translation and its application in a wheat germ cell-free system. Proc. Natl. Acad. Sci. USA, 99, 9715–9720.
8) Crick, F.H. (1968) The origin of the genetic code. J. Mol. Biol., 38, 367–379.
9) Orgel, L.E. (1968) Evolution of the genetic apparatus. J. Mol. Biol., 38, 381–393.
10) Gilbert, W. (1986) Origin of life: The RNA world. Nature, 319, 618.
11) Tagami, S., Attwater, J., & Holliger, P. (2017) Simple peptides derived from the ribosomal core potentiate RNA polymerase ribozyme function. Nat. Chem., 9, 325–332.
12) Ban, N., Nissen, P., Hansen, J., Moore, P.B., & Steitz, T.A. (2000) The complete atomic structure of the large ribosomal subunit at 2.4 Å resolution. Science, 289, 905–920.
13) Adamala, K. & Szostak, J.W. (2013) Competition between model protocells driven by an encapsulated catalyst. Nat. Chem., 5, 495–501.
14) Wieczorek, R., Dorr, M., Chotera, A., Luisi, P.L., & Monnard, P.A. (2013) Formation of RNA phosphodiester bond by histidine-containing dipeptides. ChemBioChem, 14, 217–223.
15) Turk-Macleod, R.M., Puthenvedu, D., Majerfeld, I., & Yarus, M. (2012) The plausibility of RNA-templated peptides: Simultaneous RNA affinity for adjacent peptide side chains. J. Mol. Evol., 74, 217–225.
16) Harada, K., Aoyama, S., Matsugami, A., Kumar, P.K., Katahira, M., Kato, N., & Ohkanda, J. (2014) RNA-directed amino acid coupling as a model reaction for primitive coded translation. ChemBioChem, 15, 794–798.
17) Kumar, R.K. & Yarus, M. (2001) RNA-catalyzed amino acid activation. Biochemistry, 40, 6998–7004.
18) Engelhart, A.E. & Hud, N.V. (2010) Primitive genetic polymers. Cold Spring Harb. Perspect. Biol., 2, a002196.
19) Koide, H., Yokoyama, S., Kawai, G., Ha, J.M., Oka, T., Kawai, S., Miyake, T., Fuwa, T., & Miyazawa, T. (1988) Biosynthesis of a protein containing a nonprotein amino acid by Escherichia coli: L-2-aminohexanoic acid at position 21 in human epidermal growth factor. Proc. Natl. Acad. Sci. USA, 85, 6237–6241.
20) Wong, J.T. (1975) A co-evolution theory of the genetic code. Proc. Natl. Acad. Sci. USA, 72, 1909–1912.
21) Wong, J.T. (1984) Evolution and mutation of the amino acid code, in Dynamics of Biochemical Systems (Richard, J. & Cornish-Bowden, A. eds.), pp. 247–257, Plenum Press, New York.
22) Patel, B.H., Percivalle, C., Ritson, D.J., Duffy, C.D., & Sutherland, J.D. (2015) Common origins of RNA, protein and lipid precursors in a cyanosulfidic protometabolism. Nat. Chem., 7, 301–307.
23) Shen, C., Yang, L., Miller, S.L., & Oro, J. (1987) Prebiotic synthesis of imidazole-4-acetaldehyde and histidine. Orig. Life Evol. Biosph., 17, 295–305.
24) Doi, Y., Ohtsuki, T., Shimizu, Y., Ueda, T., & Sisido, M. (2007) Elongation factor Tu mutants expand amino acid tolerance of protein biosynthesis system. J. Am. Chem. Soc., 129, 14458–14462.
25) LaRiviere, F.J., Wolfson, A.D., & Uhlenbeck, O.C. (2001) Uniform binding of aminoacyl-tRNAs to elongation factor Tu by thermodynamic compensation. Science, 294, 165–168.
26) Tamura, K. & Schimmel, P. (2004) Chiral-selective aminoacylation of an RNA minihelix. Science, 305, 1253.
27) 濡木 理(2006)アミノアシルtRNA合成酵素のX線結晶構造解析.日本結晶学会誌,48, 327–336.
28) 伊藤拓宏,横山茂之(2011)真正細菌におけるグルタミニルtRNA生合成の構造的基盤.生化学,83, 1047–1050.
29) 姚 閔,陳 美容(2018)tRNA依存的アミノ酸システインの生合成複合体の分子機構.生化学,90, 512–518.
30) Nakama, T., Nureki, O., & Yokoyama, S. (2001) Structural basis for the recognition of isoleucyl-adenylate and an antibiotic, mupirocin, by isoleucyl-tRNA synthetase. J. Biol. Chem., 276, 47387–47393.
31) Dyke, K.G., Curnock, S.P., Golding, M., & Noble, W.C. (1991) Cloning of the gene conferring resistance to mupirocin in Staphylococcus aureus. FEMS Microbiol. Lett., 61, 195–198.
32) Kawahara-Kobayashi, A., Masuda, A., Araiso, Y., Sakai, Y., Kohda, A., Uchiyama, M., Asami, S., Matsuda, T., Ishitani, R., Dohmae, N., et al. (2012) Simplification of the genetic code: Restricted diversity of genetically encoded amino acids. Nucleic Acids Res., 40, 10576–10584.
33) Forster, A.C. & Church, G.M. (2007) Synthetic biology projects in vitro. Genome Res., 17, 1–6.
34) Fujino, T., Tozaki, M., & Murakami, H. (2020) An amino acid-swapped genetic code. ACS Synth. Biol., 9, 2703–2713.
35) Theobald, D.L. (2010) A formal test of the theory of universal common ancestry. Nature, 465, 219–222.
36) Zhang, Y., Romero, H., Salinas, G., & Gladyshev, V.N. (2006) Dynamic evolution of selenocysteine utilization in bacteria: A balance between selenoprotein loss and evolution of selenocysteine from redox active cysteine residues. Genome Biol., 7, R94.
37) Rovner, A.J., Haimovich, A.D., Katz, S.R., Li, Z., Grome, M.W., Gassaway, B.M., Amiram, M., Patel, J.R., Gallagher, R.R., Rinehart, J., et al. (2015) Recoded organisms engineered to depend on synthetic amino acids. Nature, 518, 89–93.
38) Walter, K.U., Vamvaca, K., & Hilvert, D. (2005) An active enzyme constructed from a 9-amino acid alphabet. J. Biol. Chem., 280, 37742–37746.
39) Shibue, R., Sasamoto, T., Shimada, M., Zhang, B., Yamagishi, A., & Akanuma, S. (2018) Comprehensive reduction of amino acid set in a protein suggests the importance of prebiotic amino acids for stable proteins. Sci. Rep., 8, 1227.
40) Kimura, M. & Akanuma, S. (2020) Reconstruction and characterization of thermally stable and catalytically active proteins comprising an alphabet of ~13 amino acids. J. Mol. Evol., 88, 372–381.
41) Amikura, K., Sakai, Y., Asami, S., & Kiga, D. (2014) Multiple amino acid-excluded genetic codes for protein engineering using multiple sets of tRNA variants. ACS Synth. Biol., 3, 140–144.
42) Imada, T., Moriya, K., Uchiyama, M., Inukai, N., Hitotsuyanagi, M., Masuda, A., Suzuki, T., Ayukawa, S., Tagawa, Y.I., Dohmae, N., et al. (2018) A highly bioactive Lys-deficient IFN leads to a site-specific Di-PEGylated IFN with equivalent bioactivity to that of unmodified IFN-alpha2b. ACS Synth. Biol., 7, 2537–2546.
43) Kawahara-Kobayashi, A., Hitotsuyanagi, M., Amikura, K., & Kiga, D. (2014) Experimental evolution of a green fluorescent protein composed of 19 unique amino acids without tryptophan. Orig. Life Evol. Biosph., 44, 75–86.
44) 木賀大介,榎本利彦 特願2017-041685.
45) Pouplana, L.R., Santos, M.A.S., Zhu, J.-H., Farabaugh, P.J., & Javid, B. (2014) Protein mistranslation: Friend or foe? Trends Biochem. Sci., 39, 355–362.
著者紹介Author Profile
 木賀 大介(きが だいすけ)
木賀 大介(きが だいすけ)早稲田大学理工学術院電気・情報生命工学科 教授.博士(理学).
略歴1994年東京大学理学部生物化学科卒業.99年同大学院理学系研究科生物化学専攻博士課程単位取得退学.同年博士号取得.理化学研究所など各所でのポスドクの後,2004年東京大学助手,05年東京工業大学助教授・准教授を経て,16年より現職.
研究テーマと抱負合成生物学,タンパク質合成系,遺伝子ネットワークのデザイン.トリプトファンを持たない人工細胞をつくりたいです.
趣味歴史に想いを巡らすこと.