小胞体は,全タンパク質のおよそ1/3のタンパク質が生合成される場であり,細胞にとって必要不可欠な役割を果たす.小胞体において,新規合成される膜タンパク質や分泌タンパク質は,小胞体膜に結合したリボソームによって,翻訳共役的に小胞体へと送り込まれる.小胞体の内腔では,最もメジャーな小胞体シャペロンBiPなどが新生タンパク質に結合し,膜内への挿入に寄与する.小胞体におけるタンパク質の翻訳後修飾として,PDI(protein disulfide isomerase)ファミリーが媒介するジスルフィド結合形成や,Asn-X-Ser/Thr(X≠Pro)からなるコンセンサス配列のAsnに結合するN型糖鎖修飾がよく知られている1–3).N型糖鎖は小胞体に入る80%のタンパク質を修飾し,細胞内外の生命現象に深く携わっているため非常に重要である.
これらの過程や小胞体シャペロンなどに介助されて,タンパク質はそれぞれが機能発揮できる構造を獲得し,ゴルジ体以降の分泌経路へと進む.一方でどうしても正しい立体構造を獲得できないタンパク質は,小胞体から細胞質へと再び送り返され,最終的に細胞質のユビキチン–プロテアソーム系によって分解される.この一連のシステムを小胞体関連分解機構(ER-associated degradation:ERAD)と呼ぶ4–6).構造異常タンパク質は,分化の過程7),遺伝的変異8),薬剤による影響下9)などにおいて生じやすいことがわかっている.
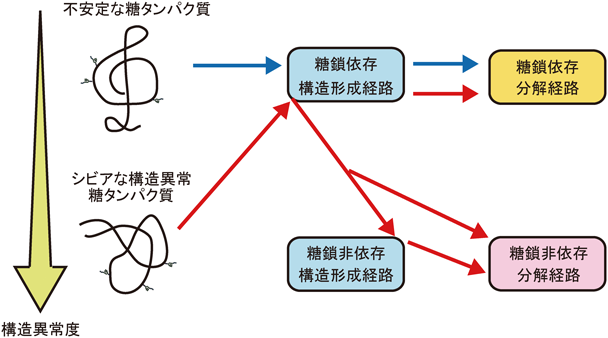
このように小胞体の恒常性は,タンパク質の構造形成と分解を厳密に制御することによって保たれる.小胞体での構造形成経路と分解経路の基本的な分子メカニズムは,酵母から高等動物まで保存されているが,高等動物では進化の過程において遺伝子が多岐化し,それぞれが重複もしくは差別化した遺伝子機能を持つようになっている.これによって,高等動物の小胞体タンパク質品質管理機構は,それぞれの組織の要求に応えられるような柔軟性も持ち合わせるようになったと考えられる.高等動物における小胞体の構造形成経路と分解経路は,N型糖鎖との関わりによってN型糖鎖依存経路とN型糖鎖非依存経路に大別される.糖鎖依存経路では,特定のN型糖鎖構造を認識するレクチン分子がタンパク質品質管理において重要な役割を果たす.
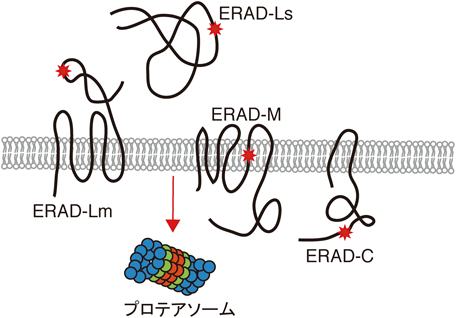
また小胞体関連分解において,その基質のトポロジーと構造異常部位によって分解経路が分類される.酵母では,小胞体の内腔領域に欠陥部位があるタンパク質をERAD-L(lumen),膜内に欠陥部位がある膜タンパク質をERAD-M(membrane),細胞質側に欠陥部位がある膜タンパク質をERAD-C(cytosol)によって処理する10).高等動物では,ERAD-Lは,欠陥部位を持つ小胞体可溶性タンパク質を処理するERAD-Ls(luminal soluble),小胞体内腔領域に欠陥部位を持つ膜タンパク質を処理するERAD-Lm(luminal membrane)に分けられる(図1)11).これらのERAD経路では,それぞれに関わる小胞体関連分解因子群が異なっている.本稿では,進化の過程で洗練されてきた上で複雑化している高等動物における小胞体タンパク質品質管理機構について,特に筆者が研究を進めてきた糖鎖依存分解経路に着目し,まとめたい.小胞体関連分解における酵母と高等動物の相同点と相違点は,最近筆者が執筆したレビューをご覧いただきたい5).
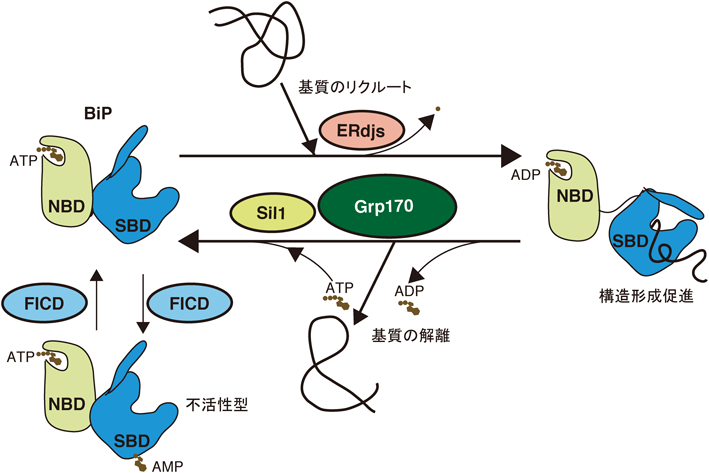
高等動物の糖鎖非依存構造形成経路と分解経路において最もよく機能しているとされている分子は,分子シャペロンであるBiP(GRP78)である.ヒトBiPは654アミノ酸からなり,N末端にはシグナル配列,nucleotide-binding domain(NBD)(25~408アミノ酸),C末端にはsubstrate-binding domain(SBD)(419~633アミノ酸)を持つ.BiPも含めたheat shock protein 70(Hsp70)ファミリーは,ATP依存的に基質との結合・解離を行う.Hsp70ファミリー分子は,ATPが結合しているとオープンな構造になっており,この状態において基質の結合を待つ.そして,ATPが加水分解されADP結合型になると,分子シャペロンはクローズドな状態に構造変化し,基質と結合する12).その後,nucleotide exchange factor(NEF)によって,分子シャペロンからADPが放出され新たなATPが結合すると,分子シャペロンは基質を解離し,オープンな構造に再び戻る(図2).このサイクルはATPase活性を上昇させるJドメインを有するDnaJタンパク質群によって制御されている.これらは分子シャペロンの機能を支援し,コシャペロンとも呼ばれる.特にBiPの場合,小胞体局在DnaJファミリー分子(ERdjs)が,その時々においてBiPとうまく協働することによって,基質の構造形成を助けたり分解を促進したりする必要不可欠な役割を果たしていると考えられている13).このERdjsのうち,ERdj1, 2, 7は膜タンパク質として,ERdj3~6は可溶性タンパク質として発現する.以下にERdjsの役割を概説する.ERdj1は,小胞体へと生合成されている新生鎖へBiPをリクルートすることに寄与する14–16).ERdj2は,Sec63としても知られ,小胞体へのポリペプチド挿入の中心因子Sec61トランスロコンのコンポーネントであり,BiPと協働してSec61のチャネルを開くことを助ける17, 18).ERdj3は,基質と直接相互作用し,BiPへと基質を受け渡す19).ERdj4とERdj5は,非糖タンパク質であるsurfactant protein Cの小胞体関連分解を促進すると報告された20, 21).ERdj4の遺伝子破壊マウスでは周産期において死に至りやすく,小胞体においてはプロインスリンの蓄積などがみられる22).チオレドキシン様ドメインを持つERdj5については,永田,潮田らによって精力的に解析がなされ,BiPと協働して機能し,小胞体関連分解に必要な還元酵素としての役割が報告された23).さらにERdj5は,LDL受容体の適切なジスルフィド結合形成にも寄与していることがわかった24).ただしERdj5の遺伝子破壊マウスは,目立った表現型が出ないため25),ERdj5の機能は,小胞体の酸化還元酵素が重複した機能を果たしていると推測される.実際,小胞体において最もメジャーな酸化還元酵素PDIが,還元酵素として機能していることも明らかになっている26, 27).また最近では,ERdj5が,小胞体に局在するカルシウムポンプであるSERCA2bのシステインを還元することによってSERCA2bを活性化させ,小胞体のカルシウム濃度上昇に寄与することも明らかにされた28).小胞体ストレスによって転写誘導されるERdj6(p58)29)は,シグナル配列を有しているが,小胞体のストレス状態によって細胞質局在30)と小胞体局在31)が報告されている.ERdj6は,細胞質では翻訳共役的な分解促進効果30),小胞体ではBiPと協働するコシャペロンとしての機能が提案されている31).ERdj7は,プロテオミクス解析によって同定されたが,機能的な解析は進んでいない32).
小胞体のNEFとしては,Grp170(HYOU1, ORP150)とSil1(BAP)がある.Grp170はNEFとしての役割だけではなく33, 34),直接基質に結合しシャペロンとしても機能する35, 36).近年では,Grp170が非糖タンパク質の分解に寄与すること37–39)や,基質の凝集を防ぐ効果も報告されている40).Sil1のNEF活性は2002年に示され,BiPが多く存在する分泌組織にSil1も多く存在する41).ただしSil1は,Grp170とは異なり小胞体関連分解には寄与しない38).BiPに対するNEF活性だけでは小胞体関連分解を促進するには至らないようである.細胞内においてBiP, ERdj3, ERdj4, ERdj5, Grp170がどのようなポリペプチド鎖と直接結合しやすいか調べたところ,BiPとERdj3はさまざまなポリペプチド鎖に広く結合した一方で,ERdj4, ERdj5, Grp170は凝集しやすい特定のポリペプチド鎖との結合がみられた42).コシャペロン,NEFに基質の嗜好性があると考えられ,それぞれがうまく使い分けられることによって,小胞体内の恒常性維持に貢献していると推測される.
またBiPのSBD内のThr518は,FICD(FIC domain-containing ER-localized enzyme)によってadenosine monophosphate(AMP)化,脱AMP化される43, 44).AMP化したBiPは,不活性化状態となり,ERdjsによるATPase活性促進効果をあまり受けず,基質との解離を6倍早める45).BiPの基質に対するジスルフィド結合の導入は,BiPと相互作用する酸化還元酵素P546)や,PDIなど47, 48)のPDIファミリーによってなされると考えられる.数あるPDIファミリー分子がそれぞれどのような基質を好んで酸化するのかは,これからの解析が待たれる.
これらのようにBiPを中心として,小胞体の糖鎖非依存構造形成,糖鎖非依存分解経路が形成されている.BiPによって構造形成が促進されているタンパク質がどのタイミングで分解へと向かうのかは明確にはわかっていない.ERdj4, ERdj5, Grp170は分解への寄与も報告されているため,これらの分子の基質への結合が一つの引き金になるのかもしれないが,今後明らかにされるべき課題である.
1)N型糖鎖依存構造形成経路とグルコースのトリミング
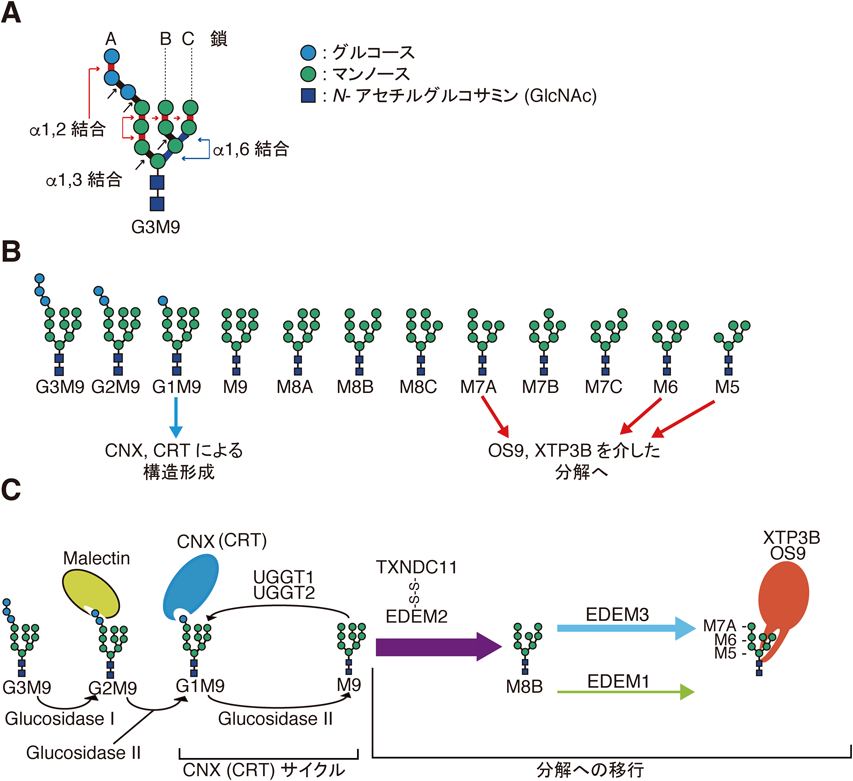
新生タンパク質に対して翻訳共役的に付加されるN型糖鎖は三つのグルコース,九つのマンノース,二つのN-アセチルグルコサミン(GlcNAc)によって構成され,これをGlc3Man9GlcNAc2(G3M9)と呼ぶ(図3A, B).三つのグルコースのうち,α1,2結合した最外部のグルコースがGlucosidase Iによって一つトリミングされる49).次に,Glucosidase IIによってα1,3結合した二つのグルコースがトリミングされるが,この二つ目と三つ目のグルコーストリミング反応は連続的には起こらない50, 51).最初のグルコースがトリミングされることによって生じるG2M9は,糖鎖を認識するレクチンタンパク質Malectinによって認識される52, 53).このMalectinによるG2M9の認識は,構造異常タンパク質の分泌の抑制に寄与する可能性が提示されている.というのも,Malectinの過剰発現は,N型糖鎖付加部位を三つ持つ可溶性モデル分解基質であるNHKのメディウム中への分泌を抑制し,小胞体のタンパク質品質管理を強めるからである54)(NHKは,null Hong Kongの略称で,全長418アミノ酸のタンパク質であるα1-antitrypsinの342番目のアミノ酸のコドンにTCの2塩基欠損が起きフレームシフトした結果,357アミノ酸のタンパク質として発現している.NHKとα1-antitrypsinの全長を比較すると,NHKはC末端領域を20%欠失した変異体である).G1M9は,小胞体のレクチンシャペロンであるCalnexin(CNX)や,そのホモログであるCalreticulin(CRT)に認識され,糖タンパク質の構造形成に寄与する55, 56).CNXは細胞質側のC末端に小胞体局在するためのRKPRREモチーフ(ダブルリシンモチーフに類似)を持つ膜タンパク質である.CRTは小胞体内腔側のC末端に小胞体局在シグナルであるKDEL配列を有する可溶性タンパク質である57).CNX, CRTともにproline-richドメイン(Pドメイン)を有し,ここで酸化還元酵素ERp57と結合する58–60).ERp57は,Pドメイン結合領域を介してこれらレクチンシャペロンと結合し,基質のジスルフィド結合形成を促進する.CNX, CRTにはそれぞれの基質特異性があり,CNXはヘマグルチニンなどの構造形成を促進しやすく61, 62),CRTはMHC class I63),bradykinin receptor64),A1AT ATZ65)などの構造形成を促しやすい.一般的には,CNXは膜に近い基質を,CRTは可溶性の基質を好むともいわれている3, 66).M9までトリミングされた際に,糖タンパク質が構造形成していれば各々の機能すべき場へと輸送されるが,構造形成されていないと,UGGT1によって糖タンパク質のM9糖鎖にグルコースが再付加されG1M9となり,再びCNX/CRTによって構造形成が試みられる.この点からUGGT1はフォールディングセンサー酵素とも呼ばれている.N型糖鎖のグルコースの糖鎖構造を基盤としたこの精巧な構造形成サイクルをCNX(CRT)サイクルと呼ぶ(図3C)55, 56).UGGT1のパラログとしてUGGT2があり,UGGT1と比べて弱いグルコース付加活性を持つ67).最近,UGGT1は比較的大きな膜タンパク質を,UGGT2は比較的小さな可溶性のタンパク質を基質として好むと報告された68).またグルコースの再付加による糖タンパク質の構造形成への寄与は限定的であり,CNX, CRTによって少なくとも一度G1M9の糖鎖を持つ基質の構造形成が促進されることの方が大きな意義を持つことが明らかになった.分泌タンパク質は,これらの構造形成過程を経て正しい立体構造を獲得し,ゴルジ体以降の分泌経路へと進むが,どのようなN型糖鎖構造を有した状態において分泌されていくかは明確にはわかっていない.構造形成に時間がかかる糖タンパク質は,よくトリミングされたN型糖鎖を有して次の区画へ輸送されると推測される.レクチン輸送因子ERGIC53はさまざまな構造のN型糖鎖に広く結合するが,レクチン輸送因子VIP36, VIPLはあまりトリミングが進んでいないN型糖鎖と相互作用しやすい69).小胞体から輸送された糖タンパク質はゴルジ体へ到達し,ゴルジ体では,高マンノース型N型糖鎖のマンノースをさらに刈り込んだ後に種々の糖転移酵素が複合型N型糖鎖へと修飾することが知られている.単糖ずつ刈り込むゴルジ体マンノシダーゼ群に加えて70–72),1~3糖のグルコースが連結されたA鎖の最外部のマンノースを切除し,一度に2~4糖を遊離させる活性を持つendo-α-1,2-mannosidase(MANEA)73–75)も主にはゴルジ体(84%)や小胞体-ゴルジ体中間コンパートメント(15%)に存在しているため76),細胞はさまざまなN型糖鎖を持つ糖タンパク質に対して,ゴルジ体において柔軟に対応できるようにしていると考えられる.
2)N型糖鎖依存分解経路とマンノーストリミング
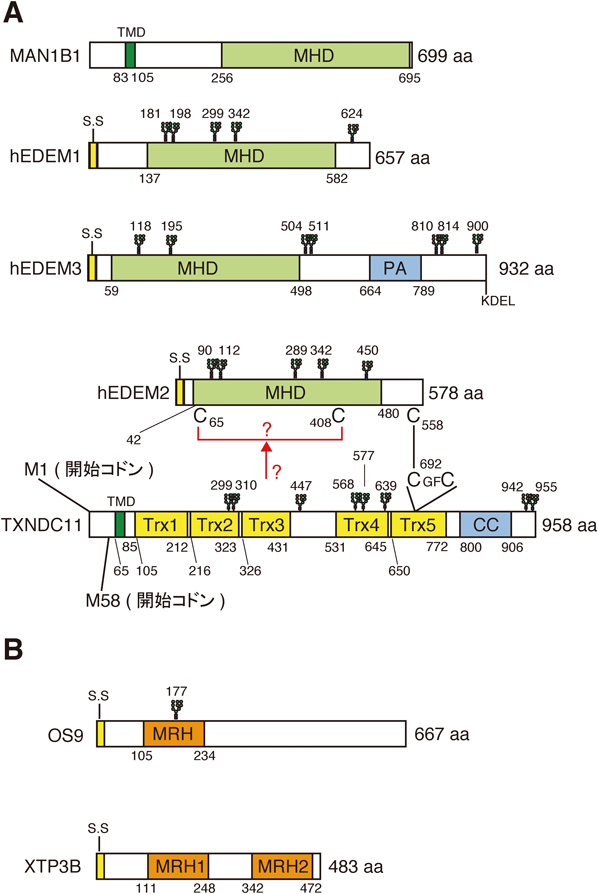
小胞体におけるM9からのマンノースのトリミングは,糖タンパク質が構造形成する段階から分解される段階へと導くことを意味する.そのためマンノースのトリミングは糖タンパク質の運命を決定するため,分子メカニズム解明の意義が大きい.しかし,高等動物におけるN型糖鎖のマンノース切除機構は,長年議論されていた.マンノースの切除を実行するマンノシダーゼ酵素の候補は,glycosyl hydrolase family 47(GH47ファミリー)に属しmannosidase homologyドメイン(MHD)を持つMAN1B1(ER manosidase I, ERmanI)と,EDEM1(ER degradation enhancing α-mannosidase like protein1),EDEM2, EDEM3からなるEDEMファミリーである(図4A).MAN1B1は,II型の膜タンパク質として発現し,内腔側に大きなドメインを持ち,M9からM8Bへのトリミング活性が,細胞内の過剰発現やin vitro活性測定によって示された77, 78).また酵母のM9からM8Bへとトリミングを行うMns1とオーソログであることからも,この知見は広く受け入れられた.MAN1B1の小胞体局在に関してはMAN1B1の細胞内における過剰発現により,CRTと共局在することから示されていた.そしてMAN1B1のM9からM5までのトリミング活性も遺伝子抑制法79)やin vitro活性80, 81)により支持され,小胞体における中心的なマンノシダーゼとしての機能は2000年代には疑念はなかった.しかし,2011年のSifersらの仕事によって状況が一変した.というのも,内在性MAN1B1を認識できる抗MAN1B1抗体による免疫染色によって,内在性MAN1B1はゴルジ体に存在することが明確に示されたからである71, 82).またMAN1B1にはゴルジ体においてのみ起こる修飾であるシアル酸の糖鎖付加も起こっていた.その後MAN1B1の結合タンパク質が網羅的に同定され,細胞質側にてγ-COPと作用することによって,小胞体関連分解基質をゴルジ体から小胞体へ送り返し,小胞体関連分解に貢献するという新規機能が示された83).MAN1B1の小胞体関連分解を促進する機能に,小胞体内腔側の酵素活性ドメインは必要なく,細胞質側が重要なことが明らかにされた84, 85).MAN1B1の細胞質側に小胞体関連分解を促す機能があることは驚きではあるが,進化的にも保存された酵素活性ドメインの細胞内の意義の解明が今後待たれる.
内在性MAN1B1のゴルジ体局在が明らかになり,小胞体においてM9からM8Bへとトリミングを行う酵素の分子実体が大きな問題となり,2014年に糖質の専門誌にも取り上げられている86).その候補として残るのはEDEM1, EDEM2, EDEM3であったが,その酵素活性については当時議論され続けていた.小胞体ストレス応答によって転写誘導される膜タンパク質として同定されたEDEM1は,精製しても酵素活性は得られなかったことや,マンノシダーゼとして機能するためのシステインが保存されていないことからも,糖鎖を認識するレクチン活性があるのではないかと提案された87).そしてEDEM1のレクチン活性によって,小胞体膜上の小胞体関連分解因子SEL1Lを認識することで,基質を逆行輸送複合体へ運ぶことも示唆された88).一方でEDEM1の細胞内過剰発現によって,A鎖89)もしくはC鎖90)のマンノースをトリミングする活性が提案された.またCNXの膜領域とEDEM1の膜領域が相互作用し,CNXから基質を受け取っていることも報告された91, 92).ただしその後,EDEM1は可溶性タンパク質であると示されている93–95).EDEM1とのホモロジーサーチによってEDEM1のパラログであるEDEM2とEDEM3が同定された.EDEM2は,その細胞内過剰発現により小胞体関連分解を促進することが示された94, 96).またEDEM2の精製タンパク質は酵素活性を発揮せず,マンノシダーゼ活性はないと報告されていた96).またEDEM3の細胞内過剰発現によってマンノースのトリミングを促進した結果,基質の分子量が小さくなったためにSDS-PAGEにおいて移動速度が上がった位置にバンドが出現することが示され,その酵素活性が支持されていた97).これらのようにEDEMファミリーのマンノシダーゼ活性の有無は議論され,サイエンスが大きく前進しない状況が続いていた.そこで,筆者らは内在性の遺伝子機能を強力に解析するために,組換えの起こりやすいニワトリBリンパ細胞であるDT40細胞と,その後のゲノム編集の技術革新に伴ってヒト結腸がん細胞であるHCT116細胞を用いて小胞体マンノシダーゼ候補群の遺伝子破壊株を作製した.そして細胞全体の糖鎖組成を調べると,予想外なことにEDEM2ノックアウト(KO)細胞では,マンノシダーゼ阻害剤であるキフネンシン処理と同程度までM9の量が顕著に増加していた.EDEM3 KO細胞,EDEM1 KO細胞ではM8Bの量が,この順に増加していた.ニワトリDT40細胞においてのみ作製したMAN1B1 KO細胞では,M9の量はあまり増加しなかったため,MAN1B1よりもEDEM2の方がはるかにM9からのトリミングに貢献していることもわかった.個別の基質に関してもEDEM2 KO細胞やキフネンシン処理において,糖鎖のトリミングが抑制された結果,SDS-PAGEにおいてバンドが上方に出現した.EDEM2 KO細胞にEDEM2を戻すとバンドが下方にシフトし,マンノーストリミング活性が検出されたが,EDEM2の活性中心に点変異を入れたEDEM2 E117Q変異体では,マンノシダーゼ活性は戻らず,下方へのバンドシフトはみられなかった.EDEM2を遺伝子破壊することによって他のタンパク質が不活性化したということではなく,EDEM2の酵素活性中心が,機能していることがわかる.これらの明快な結果より,M9からはEDEM2が,M8BからはEDEM1と主にはEDEM3がトリミングを行うという従来のモデルを一新する小胞体でのN型糖鎖のマンノースのトリミングモデルを提唱した(図3C)98).酵母では,M8Bのみが多く検出されることに対して,高等動物ではM9とM8Bが蓄積されていたため,M9→M8B, M8Bからのマンノーストリミングによって分解基質を少なくとも2段階で選別するというダブルチェック機構の提案も初めて行った.筆者らが内在性の分解基質として同定した,糖鎖を三つ持ち糖鎖依存分解経路によってもっぱら分解されるATF699)も,糖鎖トリミングの貢献の大きい順,すなわちEDEM2 KO細胞,EDEM3 KO細胞,EDEM1 KO細胞の順に分解がよく遅延していた.
しかし上記モデルは,EDEM2にはin vitro活性がない96),もしくは微弱である100)と主張され,一部の研究者には受け入れられなかった.新規モデルの確立には,細胞内の強力な活性を支持するようなEDEM2の明確なin vitro活性の検出が必要であった.EDEMの酵母オーソログHtm1は,酸化還元酵素Pdi1とジスルフィド結合を形成し,酵素活性を示すことがわかっていた101–103).筆者らはEDEM2単独ではin vitro酵素活性の検出が難しいことがわかっていたため,Htm1と同様に,酸化還元酵素などとEDEM2が複合体を形成するのではないかと考え,細胞溶解液に対して非還元SDS-PAGE実験を行った.すると,75%程度のEDEM2が何かとジスルフィド結合を作り,複合体を形成していることが明らかになった.ジーントラップ法やCRISPRスクリーニング法により,小胞体関連分解の基質としてMHC class Iの分解を促す機能を持つと同定されたTXNDC11は,チオレドキシンドメインを五つ有しており,そのうちのCXXCモチーフを唯一持つTrx5に還元活性があるレダクターゼではないかと提案されていた.またTXNDC11は,EDEM2やEDEM3とも結合すると報告されていたため,この分子に筆者らは着目した.そしてTXNDC11 KO細胞ではEDEM2のジスルフィド結合依存的な複合体のバンドが消失することを見いだし,TXNDC11がEDEM2と安定的な複合体を形成することを明らかにした.さらにTXNDC11 KO細胞では,EDEM2の細胞内酵素活性がなくなることがわかった.TXNDC11 KO細胞にTXNDC11を戻すとEDEM2の細胞内酵素活性が戻るが,EDEM2とジスルフィド結合するシステインに点変異を入れたTXNDC11 C692Sを戻してもEDEM2の活性が戻らなかった.またEDEM2のC558A変異体だとTXNDC11とジスルフィド結合による安定的な複合体が消失し,酵素活性も失活していた.これらからTXNDC11 Trx5内CXXCのN末端側のC692がEDEM2のC558と安定なジスルフィド結合形成することが,EDEM2の細胞内の酵素活性発揮に必要であると結論づけた.また当初の目的どおり,EDEM2-S-S-TXNDC11の精製を行い,遊離糖鎖M9と反応させると,明確なM8Bへのトリミング活性がついに検出された104).そして,これは精製の難しいEDEMファミリーにおいてどの糖鎖からどの糖鎖へとトリミングが起こっているか,はっきりと示す初めての報告となった.これにより2014年に筆者を中心として打ち立てた新規モデルが確立されたと考えている(図3C).今後の反響を待ちたい.
EDEM3に関しては,ERp46がそのマンノシダーゼ活性を増強することが示されている.細胞内において,EDEM3単独の過剰発現によって,NHKの糖鎖のトリミングが進んだ結果,SDS-PAGEした際,NHKに由来する下方にスメアなバンドが多く出現する.その実験系にEDEM3に加えてERp46も過剰発現させると,さらに多くのNHKのスメアなバンドが検出された.また精製EDEM3と精製TCRαを反応させても,下方にスメアなバンドが現れたが,精製ERp46をそこに加えると,さらに多くのスメアなバンドが出現することが示された105).ただし,内在性EDEM3は,EDEM2とは違い,細胞内において何かと安定的なジスルフィド結合形成をしていないこともわかっているため(筆者ら未発表データ),ERp46がEDEM3の酵素活性発揮に必須なのかどうかなど,非常に興味深い点も残されている.EDEM3がM8Bからどのような経路によってトリミングを行い,分解シグナルとなるα1,6糖鎖エピトープを露出させているのかも明らかにされていくべき課題である.またEDEM2と,EDEM1, EDEM3にはトリミング活性の違いがあるが,どのようにしてそれぞれの基質に特異的に働いているのかということも未解明である.基質を認識するマンノシダーゼ活性中心のポケットの構造などが明らかになれば,これが解決されると期待される.
3)α1,6糖鎖エピトープ認識レクチン
酵母における小胞体関連分解の結果から,EDEMファミリーによってN型糖鎖のマンノーストリミングが進むと,それをレクチン因子が認識し分解を促進すると考えられるようになっていた.高等動物において,酵母レクチンのオーソログはOS9とXTP3Bの二つがある.OS9には糖鎖を認識するとされるmannose-6-phosphate receptor homology(MRH)ドメインが一つ存在し,XTP3BにはMRHドメインが二つ存在する(図4B).OS9は,骨肉腫106)や骨髄性白血病107)において増加する遺伝子として同定され,細胞質で機能し,HIF-1αと結合し分解を促すことが報告されていた108).しかしOS9にはシグナル配列があるため,小胞体内腔における機能があると自然に考えられた.そしてOS9は,N型糖鎖のC鎖最外部のマンノースが一つトリミングされることによって露出するα1,6糖鎖エピトープをMRHドメインによって認識し109–112),小胞体関連分解を促進することが示された113–115).OS9の構造も解かれ,OS9はWW(ダブルトリプトファン)モチーフを中心として,C鎖の露出したManα1, 6Manα1, 6Manの三糖の認識を行うことが報告された111).また逆行輸送の中心となるE3リガーゼ複合体にGRP94とOS9は協働して分解基質を運ぶことも示唆されている113).
XTP3Bに関しては,アフリカツメガエルにおいて,頭部形成に寄与するKrm2に結合する因子として最初に同定された116).その後XTP3Bの機能はOS9と重複せず,小胞体関連分解には寄与しないと報告されていた113, 114, 117).むしろXTP3Bは,細胞内における過剰発現により小胞体関連分解を阻害するとも報告されていた117, 118).OS9とXTP3Bの両遺伝子抑制実験はいくつかの論文において行われていたが113, 114),一つの報告のみ,基質分解が大きく遅延し,OS9とXTP3Bの機能が重複し,小胞体関連分解にXTP3Bが貢献すると発表していた11).XTP3Bに二つ存在するMRHそれぞれのin vitro活性も解析されており,精製したXTP3B-Fc融合タンパク質を用いて,XTP3BのN末端に存在するMRH1にはレクチン活性がなく,C末端に存在するMRH2がM5などを認識することが示された119).精製したTrigger factor-XTP3Bを用いて,MRH2がマンノースのトリミングが進んでいないM9を認識するという別の報告もあった117).
こうした中,OS9とXTP3BのダブルKO(DKO)細胞の解析が遂行され,OS9とXTP3Bの機能が重複しており,両者が小胞体関連分解を促進しているという報告がなされた.この明確な根拠として,OS9, XTP3BのDKO細胞において,SDS-PAGEすると通常より下方に出現するN型糖鎖を三つ持つ内在性分解基質CD147のバンドが検出された.これは,OS9やXTP3Bがないため,糖鎖がよくトリミングされたCD147が分解されず残ってきたと考えられる.このバンドはそれぞれのシングルKO細胞,すなわちOS9 KO細胞やXTP3B KO細胞では検出されない.また精製XTP3Bのin vitroの結果とは矛盾しているが,XTP3BのMRH1がその分解に必要であることもわかった.さらに意外なことに,OS9 KO細胞では,XTP3Bがより機能するようになり,N型糖鎖付加部位を変異させた糖鎖を持たないNHK-QQQの分解が遅延することも示された.非糖分解基質に対してXTP3Bが機能することは一見驚きであるが,OS9がない場合,XTP3BがMRH2を介してSEL1Lの五つあるN型糖鎖のうち,よくトリミングが進んでいる4番目のN431のN型糖鎖に結合し,非糖分解基質の分解を抑制するようである.まとめると,XTP3Bは,分解すべき糖タンパク質に対してはMRH1ドメインを介して分解を促進し,OS9がない場合,分解すべき非糖タンパク質に対してはMRH2ドメインを介してSEL1Lに結合し分解を抑制する120).XTP3BのMRH1の分解促進機能とMRH2の分解抑制機能の基質に応じた使い分けの意義,in vitro糖鎖結合解析の結果との矛盾点の解決は今後の課題といえる.XTP3BのMRH1は,おそらくα1,6糖鎖エピトープを認識すると考えられるが,in vitroにおいてどのような条件においてそれを認識するのかという点は興味深い.何かと複合体などを形成する可能性も考えられる.XTP3Bにも,MRH2にはWWモチーフが存在し,ヒト,アフリカツメガエル,ゼブラフィッシュ,カタユウレイボヤ,ショウジョウバエまで保存されており(筆者による配列比較),XTP3Bの機能の根幹をなすと予想される.XTP3Bにおいて,そもそもなぜ二つのMRHドメインを持つ機能が必要になったかという疑問点も,MRHそれぞれの役割が整理されてくると明らかになるかもしれない.OS9, XTP3Bともに小胞体ストレス応答によって転写誘導され117),小胞体関連分解の分解キャパシティによってもOS9の量が制御される121).OS9とXTP3Bの量比が,小胞体関連分解における糖タンパク質と非糖タンパク質の分解の微妙な強弱の調節をしているのかもしれない.
4)N型糖鎖依存分解経路と非依存分解経路
酵母において,carboxypeptidase yscY(遺伝子名:Prc1)のG255R変異体であるCPY*は,小胞体関連分解のモデル基質として最もよく使われている.四つあるCPY*のN型糖鎖付加部位をなくしたCPY*0000では,分解が顕著に抑制されるため122),酵母の小胞体関連分解にはN型糖鎖が必須のように解釈されていた.一方で細胞内の遊離糖鎖の量としては,分解シグナルを呈していない糖鎖構造であるM8Bが最も多く,糖タンパク質も糖鎖に依存しない糖鎖非依存分解経路によっても処理されているとも考えられている123, 124).高等動物においては,NHK-QQQの半減期は,NHKよりも早まり,この点において酵母と高等動物は大きく違うシステムによって機能していると考えられた120, 125).そのため筆者らは,高等動物において,N型糖鎖依存分解経路と非依存分解経路の役割を詳細に調べた.ニワトリDT40とヒトHCT116においてN型糖鎖のマンノースのトリミングが停止し,糖鎖依存分解経路がまったく機能しなくなるEDEM1, EDEM2, EDEM3の多重遺伝子破壊株であるEDEMトリプルKO(TKO)細胞を作出し,解析に用いた.比較的構造形成していると考えられるATF6, CD3δ-ΔTM, EMC1の分解は,もっぱら糖鎖依存分解経路に依存していた.一方で,全体の20%の領域を欠失しているシビアな構造異常領域を持ったNHKは,EDEM TKO細胞において初期の分解遅延が確認されたが,最終的には野生型細胞同様に分解処理された.これらの基質は共通の性質としてN型糖鎖を三つ有しており,CD3δ-ΔTMとNHKはともに可溶性タンパク質である.そこでCD3δ-ΔTMとNHKの基質の挙動の違いは,構造異常の度合いに由来すると仮定し,CD3δ-ΔTMのβstrandの中央にプロリンとアラニンの繰り返し配列7アミノ酸を挿入し構造を大きく壊したCD3δ-ΔTM-〈33-7aa-34〉と,EMC1の他のタンパク質との相互作用に関わっていると予想されるPQQ-likeドメインの大部分を欠失させたEMC1-ΔPQQを作製した.その結果,CD3δ-ΔTM-〈33-7aa-34〉とEMC1-ΔPQQでは,NHKの挙動同様,EDEM TKO細胞において早い時間帯での分解遅延が観察されたが,最終的には野生型細胞の場合と同様に分解された.変異を入れたタンパク質の構造がより壊れていることは,トリプシン感受性アッセイ126)や,筆者らが開発したPNGase感受性アッセイ127, 128)によって示した.これらの結果より,高等動物において構造異常の度合いの高い分解基質は,糖鎖依存分解経路に加えて糖鎖非依存分解経路によって分解されることがわかった(図5)129).この報告に次いで,同様の報告が他誌に掲載されている120, 130).より毒性が高いと考えられるシビアな構造異常糖タンパク質を,二つの経路により確実に分解処理するシステムによって,高等動物は小胞体の恒常性を維持していると考えられる.
1)小胞体関連分解に関わるE3ユビキチンリガーゼ
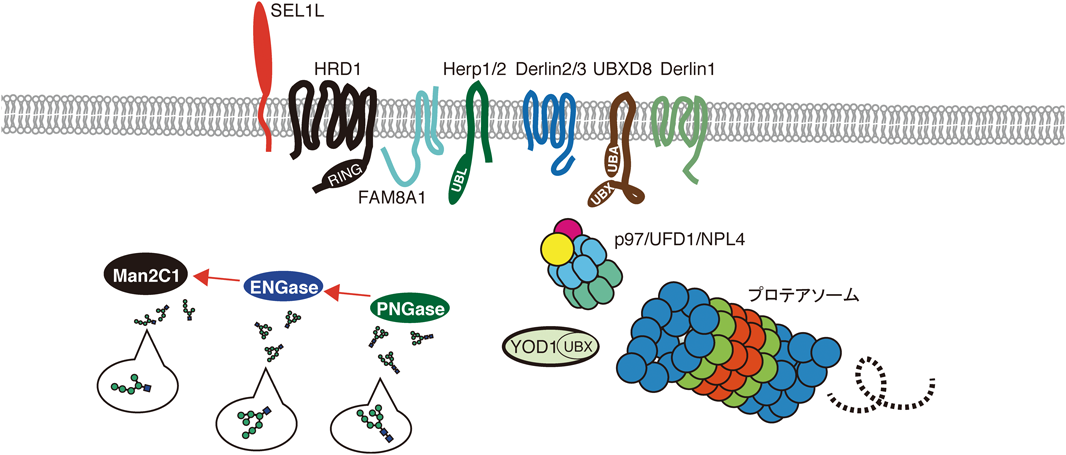
小胞体関連分解における大きな問題の一つとして,逆行輸送チャネルの実体の同定があげられる.現在その中心として考えられているのが,小胞体関連分解における最もメジャーなE3ユビキチンリガーゼであるHRD1である.HRD1は,複数回膜貫通タンパク質で,細胞質側にユビキチン化を促進するRING(really interesting new gene)フィンガードメインを持ち(図6)131, 132),SEL1Lと複合体を形成する.SEL1Lは,小胞体内腔側に大きなドメインを持つ1回膜貫通型タンパク質であり,可溶性分解基質のアクセプターとして機能することが主な役割とされている113, 133, 134).実際に可溶性タンパク質の分解はSEL1Lが減少すると大きく抑制される11, 135).ただしSEL1Lまで基質が運ばれる分子メカニズムは,詳細には解析されていない.EDEM1, EDEM3, OS9, XTP3BがSEL1Lと結合する98, 120, 136)ので,これらの可溶性小胞体関連分解促進因子が基質運搬因子の候補と考えられる.実際にEDEM188)やOS9113)は,その機能が提案されているが,決定打には至っていない印象である.その一方で,SEL1Lが可溶性タンパク質だけでなく内在性の膜タンパク質ATF699)やIRE1137)の分解にも寄与することも報告され,SEL1Lの小胞体関連分解における広範囲な役割が明らかにされている.また酵母のHrd1は,SEL1Lオーソログである酵母Hrd3によって安定化されるが138),高等動物のHRD1によってSEL1Lが逆に安定化されるという報告もある125).
2)HRD1-SEL1L複合体の機能を補助する因子群
小胞体ストレスによって誘導される分子として同定されたHerp1は139),小胞体膜をヘアピン状に貫通し,N末端とC末端が細胞質側にある(図6).そしてHerp1は2006年に酵母Usa1と機能的な相同性があることがわかり,小胞体関連分解への寄与が明らかになった140).HRD1とHerp1は結合し141),HRD1による基質のユビキチン化を正に制御していることも明らかにされた142).またHerp1のパラログであるHerp2は,恒常的に発現しており,Herp1と機能的に重複することがわかった143, 144).Herp1は酵母Usa1とは違いHRD1のオリゴマー化を促進しないが,この機能は,酵母にはオーソログがないFAM8A1によって代替されていると考えられる145, 146).HRD1のオリゴマー化によりHerp1がHRD1に結合しやすくなることも明らかにされている.Herp1はDerlin1141)やDerlin2143)と結合し,HRD1複合体へDerlinsをリクルートする.ただしDerlin1よりもDerlin2の方が,HRD1複合体とより結合が強い147, 148).Derlinファミリーは,膜内プロテアーゼとして機能するRhomboidファミリーに属しDerlin1149, 150),Derlin2, Derlin3147)からなり,それぞれ小胞体関連分解を促進する.これらDerlinファミリーのオーソログである酵母Der1は,ERAD-L経路の基質分解に必要で,酵母Hrd1とともに逆行輸送のチャネルを形成することが,クライオ電子顕微鏡による構造解析の結果から提案されている151).Derlin1とDerlin2はユビキタスに発現しているが,Derlin3の発現は組織特異性が高い152).6回膜貫通タンパク質であるDerlin1は,NHKの分解をRhomboidドメインを介して促進する153, 154).Derlin2とDerlin3には機能的な重複がみられ,Derlin1とは異なる基質をターゲットとしており,SEL1Lを必要とする基質の分解に必要なことが筆者らによって明らかにされた144).Derlin1に関しては,部分的な再構成系により逆行輸送のチャネルを形成していることを示唆する報告がある.しかし,基質としてpro-αファクターの三つのN型糖鎖付加部位をなくした単純な構造タンパク質を用いていることや,膜には種々のタンパク質が存在する精製ミクロソームを用いている.そのため,使われた抗Derlin1抗体により逆行輸送が抑制されることは確かであるが,Derlin1が逆行輸送チャネルの中心を形成しているという決定打になっているとはいいがたい155).それぞれのDerlinファミリーが逆行輸送チャネルの形成に関わっているか,関わっているならどのように貢献しているのか,酵母においてHrd1とDer1が協働してチャネルを形成していたことと同様に151),高等動物のDerlinファミリーがHRD1と協働するのかなど,今後の解析が待たれる.
3)小胞体膜に局在するE3リガーゼ
酵母にはHrd1のパラログは存在していないが,ヒトではHRD1のパラログであるgp78も小胞体関連分解に寄与している.gp78はSEL1Lと複合体を形成しないことが要因として考えられるが116),主に膜タンパク質の分解に関わる.HRD1とgp78のターゲットとなる分解基質は部分的には重複しており,両者ともに膜タンパク質であるBACE476や,NHKにBACEの膜領域を付与したNHKBACEの分解11),またHMG-CoAレダクターゼ156)の分解に関わっている.一方で,gp78独自の基質も存在しており,コレステロール・脂肪酸代謝に関与する酵素遺伝子の発現を増強するSREBP-SCAP複合体を小胞体にとどめるアンカー因子Insigの分解を促進する157, 158).また,CD3δの分解もHRD1には依存せず,gp78によって推進されることが報告されている11, 159).gp78の他にも小胞体膜上に存在するE3リガーゼとして,RMA1(RNF5),RNF185, RNF170, TRC8(RNF139),RNF145, TEB4(MARCH-6)などがあげられる5).これらは高等動物では明確に分けられていない二つの分解経路であるERAD-M, ERAD-Cに関わっていると考えられている.TEB4を除いて酵母にはそれらのオーソログは存在していない(TEB4のオーソログ酵母Doa10は,ERAD-Cの中心因子として機能する10)).進化とともに出現した上記E3リガーゼ群は,HRD1, gp78と重複した基質をターゲットとする場合もある.例をあげるとRNF145は,HRD1, gp78とともにHMG-CoAレダクターゼの分解を促進する156).小胞体膜上にさまざまなE3リガーゼが存在することは,それぞれの組織において,その場の要求に合わせた膜タンパク質の分解を遂行することに寄与しているのではないかと推測される.近年では,RNF185とTEB4が異なる膜タンパク質の構造を認識して分解に導いていることもわかってきており160),高等動物の小胞体関連分解に寄与するE3リガーゼの役割が少しずつ明らかになっている.
高等動物における小胞体タンパク質品質管理機構は,細胞質においてみられるような(糖鎖に依存しない)分子シャペロンによる構造形成,分解機構と,小胞体を通過するタンパク質の大部分に結合するN型糖鎖を巧妙に利用した構造形成,分解機構によって織りなされている.特に分解機構は,最終的には細胞質のプロテアソームによって分解を執行するため,基質の認識,逆行輸送チャネルへのリクルート,逆行輸送,ユビキチン化,細胞質側からの基質の引き抜き,N型糖鎖の遊離,プロテアソームでの分解と,必須ステップが多いため,それに伴って関わる遺伝子も増加し,重要な研究課題が多く提示されている.そして生体内での意義も大きく,小胞体関連分解は,60種類以上の疾患と関わっていることもわかっている178).
小胞体品質管理を行う因子に関して,酵母の遺伝子が進化的に多岐化した場合でも,高等動物の生命活動において必要不可欠であることも多い.小胞体関連分解因子群の知見としては,Derlin1179),Derlin2180),HRD1181)を遺伝子破壊したマウスでは,それぞれ7日,13.5日,13.5日において胚性致死となる.Herp1の遺伝子破壊マウスでは小胞体ストレスの影響からかブドウ糖負荷試験において通常のように血糖値が下がらないという状態になり179),gp78の遺伝子破壊マウスでは,非アルコール性脂肪性肝炎を発症する182).ただし,Derlin3179)の遺伝子破壊マウスは表現型に大きな変化がなく産まれてくる.
構造形成促進する因子の遺伝子破壊マウスに関して,CNX遺伝子破壊マウスでは発育不全や有髄神経繊維の喪失に伴う運動不全になり183, 184),CRT遺伝子破壊マウスでは胎生期18日には心臓の機能不全によりその多くは胚性致死となる185).ERp57遺伝子破壊マウスは,CNX遺伝子破壊マウスよりもシビアな表現型を示し,胚性致死であり,そのB細胞特異的なコンディショナル遺伝子破壊マウスでは,ERp57はMHC class Iの酸化還元には関与しないが,安定的な複合体形成に寄与することが明らかになっている186).近年の論文において,細胞を用いた実験によってUGGT1のグルコースの再付加による構造形成の寄与は限定的でCNX(CRT)による構造形成の方が重要と示された68).こうした中でも,UGGT1遺伝子破壊マウスは胚性13日において致死となり,CNX遺伝子破壊マウスよりもUGGT1遺伝子破壊マウスの方がシビアな表現型を示すことは187),現在までに研究者らによって進んでいる解析の到達点や生体の奥深さも感じさせる.
小胞体関連分解の大きな問題の一つとして取り上げられてきていた逆行輸送のチャネルに関しては,酵母においてはHrd1-Der1を中心とした複合体が形成していることが構造学的にわかってきている151).高等動物では,光架橋解析や構造解析などがまだまだ進んでおらず,逆行輸送チャネルに関する知見は少ないが,HRD1-SEL1Lの複合体だけでなく,他の複数回膜貫通型の小胞体関連分解因子が特定の基質に対して逆行輸送チャネルを形成していることも考えられる.その候補となるE3リガーゼやDerlinファミリーは進化的に遺伝子が多岐化しており,酵母に比べて高度な複合体を形成しているのかもしれない.
酵母では,遺伝子破壊が容易かつ機能的に重複した遺伝子が少ないことを主な理由として,比較的早い段階で統一的見解に至ってきたが,高等動物においては,難問に対して解決まで時間が長くかかってきた印象がある.近年のゲノム編集技術の発展によって,高等動物においても,内在性の遺伝子の機能解析をより強力に進められるようになり,それまで議論されていた遺伝子の機能も明確にされる場合も多い.本稿でも疑問点を提示してきたように現在でも課題は多く,今後どのような研究成果が出されるのか,矛盾点がどのように解決されていくのか,筆者も当該分野の問題解決の一部に貢献できればうれしく想う.